-検索条件
-検索結果
検索 (著者・登録者: savva & c)の結果119件中、1から50件目までを表示しています

EMDB-18290: 
Cryo-EM structure of Cx26 gap junction K125E mutant in bicarbonate buffer (classification on hemichannel)

EMDB-18291: 
Cryo-EM structure of Cx26 solubilised in LMNG - hemichannel classification - NConst conformation

EMDB-18292: 
Cryo-EM structure of Cx26 solubilised in LMNG - Hemichannel classification NFlex conformation
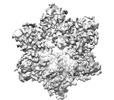
EMDB-18293: 
Cryo-EM structure of Cx26 solubilised in LMNG: classification on subunit A; Nconst-mon conformation

EMDB-18294: 
Cryo-EM structure of Cx26 solubilised in LMNG: classification on subunit A; NFlex conformation

EMDB-18295: 
Cryo-EM reconstruction of Cx26 gap junction K125R mutant (D6 symmetry)
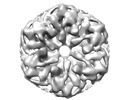
EMDB-18296: 
Cryo-EM reconstruction of Cx26 gap junction K125E mutant in HEPES buffer

EMDB-18297: 
Cryo-EM reconstruction of Cx26 gap junction WT in HEPES buffer

PDB-8q9z: 
Cryo-EM structure of Cx26 gap junction K125E mutant in bicarbonate buffer (classification on hemichannel)

PDB-8qa0: 
Cryo-EM structure of Cx26 solubilised in LMNG - hemichannel classification - NConst conformation

PDB-8qa1: 
Cryo-EM structure of Cx26 solubilised in LMNG - Hemichannel classification NFlex conformation

PDB-8qa2: 
Cryo-EM structure of Cx26 solubilised in LMNG: classification on subunit A; Nconst-mon conformation

PDB-8qa3: 
Cryo-EM structure of Cx26 solubilised in LMNG: classification on subunit A; NFlex conformation

EMDB-50185: 
KS + AT di-domain of polyketide synthase 13 in Mycobacterium tuberculosis

EMDB-39478: 
Structure of Aquifex aeolicus Lumazine Synthase by Cryo-Electron Microscopy to 1.42 Angstrom Resolution

PDB-8yt4: 
Structure of Aquifex aeolicus Lumazine Synthase by Cryo-Electron Microscopy to 1.42 Angstrom Resolution

EMDB-16820: 
Cryo-EM structure of a pre-dimerized murine IL-12 complete extracellular signaling complex (Class 1).

EMDB-16821: 
Cryo-EM structure of a pre-dimerized murine IL-12 complete extracellular signaling complex (Class 2).

EMDB-16822: 
Cryo-EM structure of the murine IL-12 complete extracellular signaling complex (Class 1).

EMDB-16823: 
Cryo-EM structure of the murine IL-12 complete extracellular signaling complex (Class 2).

EMDB-16824: 
Cryo-EM structure of a pre-dimerized human IL-23 complete extracellular signaling complex.

EMDB-17580: 
Cryo-EM structure of a pre-dimerized murine IL-12 complete extracellular signaling complex (Class 1), obtained after local refinement.

PDB-8odz: 
Cryo-EM structure of a pre-dimerized murine IL-12 complete extracellular signaling complex (Class 1).

PDB-8oe0: 
Cryo-EM structure of a pre-dimerized murine IL-12 complete extracellular signaling complex (Class 2).

PDB-8oe4: 
Cryo-EM structure of a pre-dimerized human IL-23 complete extracellular signaling complex.

PDB-8pb1: 
Cryo-EM structure of a pre-dimerized murine IL-12 complete extracellular signaling complex (Class 1), obtained after local refinement.

EMDB-15677: 
Cryo-EM structure for mouse leptin in complex with the mouse LEP-R ectodomain (1:2 mLEP:mLEPR model)

EMDB-15678: 
Mouse leptin:LEP-R complex cryoEM structure (3:3 model)

EMDB-15679: 
Cryo-EM structure for a 3:3 complex between mouse leptin and the mouse LEP-R ectodomain (local refinement)

EMDB-15680: 
Human leptin in complex with the human LEP-R ectodomain fused to a C-terminal trimeric isoleucine GCN4 zipper (2:2 model)

EMDB-15681: 
Human leptin in complex with the human LEP-R ectodomain fused to a C-terminal trimeric isoleucine GCN4 zipper (closed 3:3 model)

EMDB-15683: 
Human leptin in complex with the human LEP-R ectodomain fused to a C-terminal trimeric isoleucine GCN4 zipper (open 3:3 model).

EMDB-15899: 
Cryo-EM structure for the mouse LEPR-CRH2:Leptin:LEPR-Ig complex following symmetry expansion in combination with local refinement

PDB-8avb: 
Cryo-EM structure for mouse leptin in complex with the mouse LEP-R ectodomain (1:2 mLEP:mLEPR model).

PDB-8avc: 
Mouse leptin:LEP-R complex cryoEM structure (3:3 model)

PDB-8avd: 
Cryo-EM structure for a 3:3 complex between mouse leptin and the mouse LEP-R ectodomain (local refinement)

PDB-8ave: 
Human leptin in complex with the human LEP-R ectodomain fused to a C-terminal trimeric isoleucine GCN4 zipper (2:2 model)

PDB-8avf: 
Human leptin in complex with the human LEP-R ectodomain fused to a C-terminal trimeric isoleucine GCN4 zipper (closed 3:3 model)

PDB-8avo: 
Human leptin in complex with the human LEP-R ectodomain fused to a C-terminal trimeric isoleucine GCN4 zipper (open 3:3 model).

PDB-8b7q: 
Cryo-EM structure for the mouse LEPR-CRH2:Leptin:LEPR-Ig complex following symmetry expansion in combination with local refinement

EMDB-13594: 
Cryo-EM structure of Saccharomyces cerevisiae TOROID (TORC1 Organized in Inhibited Domains).

EMDB-13595: 
Helical reconstruction of TOROID (TORC1 Organized in Inhibited Domains) filaments.

PDB-7pqh: 
Cryo-EM structure of Saccharomyces cerevisiae TOROID (TORC1 Organized in Inhibited Domains).

EMDB-14971: 
Cryo-EM structure of the indirubin-bound Hsp90-XAP2-AHR complex

EMDB-14972: 
Cryo-EM structure of the indirubin-bound Hsp90-XAP2-AHR complex (focused map).

PDB-7zub: 
Cryo-EM structure of the indirubin-bound Hsp90-XAP2-AHR complex

EMDB-13664: 
GA1 bacteriophage portal protein

EMDB-13665: 
PhiCPV4 bacteriophage Portal Protein

PDB-7pv2: 
GA1 bacteriophage portal protein

PDB-7pv4: 
PhiCPV4 bacteriophage Portal Protein
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
