+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2xkv | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
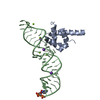
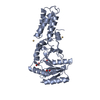
| タイトル | Atomic Model of the SRP-FtsY Early Conformation | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード |  PROTEIN TRANSPORT PROTEIN TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 シグナル認識粒子 / シグナル認識粒子 /  signal-recognition-particle GTPase / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / protein targeting to membrane / signal-recognition-particle GTPase / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / protein targeting to membrane /  ribonucleoprotein complex / ribonucleoprotein complex /  GTPase activity / GTP binding / GTPase activity / GTP binding /  ATP hydrolysis activity / ATP hydrolysis activity /  細胞膜 ... 細胞膜 ... シグナル認識粒子 / シグナル認識粒子 /  signal-recognition-particle GTPase / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / protein targeting to membrane / signal-recognition-particle GTPase / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / protein targeting to membrane /  ribonucleoprotein complex / ribonucleoprotein complex /  GTPase activity / GTP binding / GTPase activity / GTP binding /  ATP hydrolysis activity / ATP hydrolysis activity /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   ESCHERICHIA COLI (大腸菌) ESCHERICHIA COLI (大腸菌) | |||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 13.5 Å クライオ電子顕微鏡法 / 解像度: 13.5 Å | |||||||||
 データ登録者 データ登録者 | Estrozi, L.F. / Boehringer, D. / Shan, S.-o. / Ban, N. / Schaffitzel, C. | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2011 ジャーナル: Nat Struct Mol Biol / 年: 2011タイトル: Cryo-EM structure of the E. coli translating ribosome in complex with SRP and its receptor. 著者: Leandro F Estrozi / Daniel Boehringer / Shu-Ou Shan / Nenad Ban / Christiane Schaffitzel /  要旨: We report the 'early' conformation of the Escherichia coli signal recognition particle (SRP) and its receptor FtsY bound to the translating ribosome, as determined by cryo-EM. FtsY binds to the ...We report the 'early' conformation of the Escherichia coli signal recognition particle (SRP) and its receptor FtsY bound to the translating ribosome, as determined by cryo-EM. FtsY binds to the tetraloop of the SRP RNA, whereas the NG domains of the SRP protein and FtsY interact weakly in this conformation. Our results suggest that optimal positioning of the SRP RNA tetraloop and the Ffh NG domain leads to FtsY recruitment. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2xkv.cif.gz 2xkv.cif.gz | 167.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2xkv.ent.gz pdb2xkv.ent.gz | 133.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2xkv.json.gz 2xkv.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xk/2xkv https://data.pdbj.org/pub/pdb/validation_reports/xk/2xkv ftp://data.pdbj.org/pub/pdb/validation_reports/xk/2xkv ftp://data.pdbj.org/pub/pdb/validation_reports/xk/2xkv | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  シグナル認識粒子 / FIFTY-FOUR HOMOLOG シグナル認識粒子 / FIFTY-FOUR HOMOLOG分子量: 32300.371 Da / 分子数: 1 / 断片: NG DOMAIN, RESIDUES 1-294 / 由来タイプ: 組換発現 / 由来: (組換発現)   ESCHERICHIA COLI (大腸菌) / 発現宿主: ESCHERICHIA COLI (大腸菌) / 発現宿主:   ESCHERICHIA COLI (大腸菌) / 参照: UniProt: O07347, ESCHERICHIA COLI (大腸菌) / 参照: UniProt: O07347,  signal-recognition-particle GTPase signal-recognition-particle GTPase |
|---|---|
| #2: RNA鎖 | 分子量: 36856.863 Da / 分子数: 1 / 由来タイプ: 天然 詳細: ONLY THE PART OF THE 4.5S RNA THAT IS VISIBLE IN THE EM RECONSTRUCTION IS INCLUDED 由来: (天然)   ESCHERICHIA COLI (大腸菌) ESCHERICHIA COLI (大腸菌) |
| #3: タンパク質 |  シグナル認識粒子 / FIFTY-FOUR HOMOLOG / P48 シグナル認識粒子 / FIFTY-FOUR HOMOLOG / P48分子量: 8160.762 Da / 分子数: 1 / 断片: M DOMAIN, RESIDUES 329-430 / 由来タイプ: 天然 詳細: ONLY THE PART OF THE M DOMAIN THAT IS VISIBLE IN THE EM RECONSTRUCTION IS INCLUDED 由来: (天然)   ESCHERICHIA COLI (大腸菌) / 参照: UniProt: P0AGD7 ESCHERICHIA COLI (大腸菌) / 参照: UniProt: P0AGD7 |
| #4: タンパク質 |  細胞分裂 細胞分裂分子量: 32972.230 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   ESCHERICHIA COLI (大腸菌) / 発現宿主: ESCHERICHIA COLI (大腸菌) / 発現宿主:   ESCHERICHIA COLI (大腸菌) / 参照: UniProt: P83749 ESCHERICHIA COLI (大腸菌) / 参照: UniProt: P83749 |
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: RNC-SRP-FTSY / タイプ: RIBOSOME |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 50 mM Hepes-KOH, 100 mM KOAc, 8 mM Mg(OAc)2 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
| 試料支持 | 詳細: HOLEY CARBON |
急速凍結 | 装置: HOMEMADE PLUNGER / 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TECNAI F20 |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 50000 X / 最大 デフォーカス(公称値): 3500 nm / 最小 デフォーカス(公称値): 1500 nm Bright-field microscopy / 倍率(公称値): 50000 X / 最大 デフォーカス(公称値): 3500 nm / 最小 デフォーカス(公称値): 1500 nm |
| 撮影 | フィルム・検出器のモデル: KODAK SO-163 FILM |
| 放射波長 | 相対比: 1 |
 ムービー
ムービー コントローラー
コントローラー

































 PDBj
PDBj





























 解析
解析