-検索条件
-検索結果
検索 (著者・登録者: yun & z)の結果2,675件中、1から50件目までを表示しています

EMDB-46793: 
Cryo-EM structures of full-length integrin alphaIIbbeta3 in native lipids complexed with modified tirofiban

EMDB-46794: 
Cryo-EM Structures of Full-Length Integrin alphaIIbbeta3 in Native Lipids Complexed with Tirofiban

PDB-9deq: 
Cryo-EM structures of full-length integrin alphaIIbbeta3 in native lipids complexed with modified tirofiban

PDB-9der: 
Cryo-EM Structures of Full-Length Integrin alphaIIbbeta3 in Native Lipids Complexed with Tirofiban

EMDB-37522: 
MPOX E5 hexamer AMP-PNP and ssDNA bound form with clear primase domain

EMDB-37523: 
MPOX E5 double hexamer ssDNA bound conformation

PDB-8wgy: 
MPOX E5 hexamer AMP-PNP and ssDNA bound form with clear primase domain

PDB-8wgz: 
MPOX E5 double hexamer ssDNA bound conformation

EMDB-37524: 
MPOX E5 hexamer ssDNA and AMP-PNP bound conformation

PDB-8wh0: 
MPOX E5 hexamer ssDNA and AMP-PNP bound conformation

EMDB-60689: 
Structure of urea-treated empty bacteriophage T5 connector complex

EMDB-60695: 
Structure of the urea-treated empty bacteriophage T5 portal complex
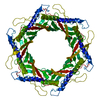
PDB-9imh: 
Structure of urea-treated empty bacteriophage T5 connector complex

PDB-9imv: 
Structure of the urea-treated empty bacteriophage T5 portal complex

EMDB-37527: 
MPOX E5 hexamer apo form

EMDB-37528: 
MPOX E5 hexamer ssDNA bound apo conformation

EMDB-37530: 
MPOX E5 hexamer ADP and ssDNA bound and clear primase domain conformation

PDB-8wh3: 
MPOX E5 hexamer apo form

PDB-8wh4: 
MPOX E5 hexamer ssDNA bound apo conformation

PDB-8wh6: 
MPOX E5 hexamer ADP and ssDNA bound and clear primase domain conformation

EMDB-19907: 
CryoEM structure of human full-length alpha1beta3gamma2L GABA(A)R in complex with GABA and puerarin

PDB-9eqg: 
CryoEM structure of human full-length alpha1beta3gamma2L GABA(A)R in complex with GABA and puerarin

EMDB-60672: 
Structure of the bacteriophage T5 portal complex

PDB-9ilp: 
Structure of the bacteriophage T5 portal complex

EMDB-38538: 
Cryo-EM structure of the tethered agonist-bound human PAR1-Gq complex

EMDB-38539: 
Cryo-EM structure of the tethered agonist-bound human PAR1-Gi complex

PDB-8xor: 
Cryo-EM structure of the tethered agonist-bound human PAR1-Gq complex

PDB-8xos: 
Cryo-EM structure of the tethered agonist-bound human PAR1-Gi complex

EMDB-40796: 
BG505 GT1.1 SOSIP in complex with NHP Fabs 12C11 and RM20A3

PDB-8sw3: 
BG505 GT1.1 SOSIP in complex with NHP Fabs 12C11 and RM20A3

EMDB-43500: 
Cryo-electron tomography of wildtype LAF-1 RGG domain protein condensates with fibrous necks
EMDB-43502: 
Cryo-Electron Tomography of Wildtype LAF-1 RGG Domain Condensate with Core/Shell Structure

EMDB-43503: 
Cryo-Electron Tomography of Wildtype LAF-1 RGG domain Condensate

EMDB-43504: 
Cryo-Electron Tomography of LAF-1 RGG Charge-Separated SH Mutant Condensate

EMDB-60511: 
Structure of the bacteriophage T5 capsid

EMDB-60675: 
Structure of the bacteriophage T5 connector complex

EMDB-60712: 
Structure of bacteriophage T5 tail tube

EMDB-60750: 
Structure of the bacteriophage T5 tail tip complex

PDB-8zvi: 
Structure of the bacteriophage T5 capsid

PDB-9ilv: 
Structure of the bacteriophage T5 connector complex

PDB-9iny: 
Structure of bacteriophage T5 tail tube

PDB-9ioz: 
Structure of the bacteriophage T5 tail tip complex

EMDB-38149: 
Cryo-EM structure of a bacteriophage tail- spike protein against Klebsiella pneumoniae K64,ORF41(K64-ORF41)

EMDB-38151: 
Cryo-EM structure of a bacteriophage tail- spike protein against Klebsiella pneumoniae K64,ORF41(K64-ORF41) in 5 mM EDTA

PDB-8x8m: 
Cryo-EM structure of a bacteriophage tail- spike protein against Klebsiella pneumoniae K64,ORF41(K64-ORF41)

PDB-8x8o: 
Cryo-EM structure of a bacteriophage tail- spike protein against Klebsiella pneumoniae K64,ORF41(K64-ORF41) in 5 mM EDTA

EMDB-37579: 
Cryo-EM structure of URAT1(R477S)

EMDB-37580: 
Cryo-EM structure of OAT4

EMDB-37589: 
Cryo-EM structure of URAT1(R477S)-Urate complex

PDB-8wjg: 
Cryo-EM structure of URAT1(R477S)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
