-検索条件
-検索結果
検索 (著者・登録者: martin & f)の結果2,474件中、1から50件目までを表示しています

EMDB-50296: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

PDB-9fbv: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

EMDB-50090: 
Vibrio cholerae DdmD apo complex

PDB-9ezx: 
Vibrio cholerae DdmD apo complex

EMDB-18864: 
Central glycolytic genes regulator (CggR) bound to DNA operator

PDB-8r3g: 
Central glycolytic genes regulator (CggR) bound to DNA operator
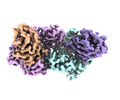
EMDB-42375: 
Deinococcus aerius TR0125 C-glucosyl deglycosidase (CGD), cryo-EM

PDB-8umc: 
Deinococcus aerius TR0125 C-glucosyl deglycosidase (CGD), cryo-EM

EMDB-42430: 
Structure of synaptic vesicle protein 2B with padsevonil

EMDB-42431: 
Structure of synaptic vesicle protein 2A in complex with a nanobody
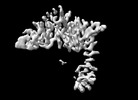
EMDB-42432: 
Structure of the synaptic vesicle protein 2A Luminal domain in complex with a nanobody

PDB-8uo8: 
Structure of synaptic vesicle protein 2B with padsevonil

PDB-8uo9: 
Structure of synaptic vesicle protein 2A in complex with a nanobody

PDB-8uoa: 
Structure of the synaptic vesicle protein 2A Luminal domain in complex with a nanobody

EMDB-42974: 
Myxococcus xanthus EncA 3xHis pore mutant with T=1 icosahedral symmetry

EMDB-42975: 
Myxococcus xanthus EncA 3xHis pore mutant with tetrahedral symmetry

PDB-8v4n: 
Myxococcus xanthus EncA 3xHis pore mutant with T=1 icosahedral symmetry

PDB-8v4q: 
Myxococcus xanthus EncA 3xHis pore mutant with tetrahedral symmetry

EMDB-19886: 
Distinct life cycle stages of an ectosymbiotic DPANN archaeon

EMDB-19887: 
Distinct life cycle stages of an ectosymbiotic DPANN archaeon

EMDB-19888: 
Distinct life cycle stages of an ectosymbiotic DPANN archaeon

EMDB-19889: 
Distinct life cycle stages of an ectosymbiotic DPANN archaeon

EMDB-19890: 
Distinct life cycle stages of an ectosymbiotic DPANN archaeon

EMDB-19891: 
Distinct life cycle stages of an ectosymbiotic DPANN archaeon

EMDB-19892: 
Distinct life cycle stages of an ectosymbiotic DPANN archaeon

EMDB-19893: 
Distinct life cycle stages of an ectosymbiotic DPANN archaeon
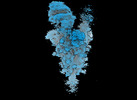
EMDB-18639: 
Locally refined SARS-CoV-2 BA-2.86 Spike receptor binding domain (RBD) complexed with angiotensin converting enzyme 2 (ACE2)

EMDB-18649: 
Local refinement of SARS-CoV-2 BA.2.86 Spike and XBB-7 Fab

EMDB-19002: 
XBB-4 Fab in complex with SARS-CoV-2 BA.2.12.1 Spike Glycoprotein

PDB-8qsq: 
Locally refined SARS-CoV-2 BA-2.86 Spike receptor binding domain (RBD) complexed with angiotensin converting enzyme 2 (ACE2)

PDB-8qtd: 
Local refinement of SARS-CoV-2 BA.2.86 Spike and XBB-7 Fab

PDB-8r8k: 
XBB-4 Fab in complex with SARS-CoV-2 BA.2.12.1 Spike Glycoprotein

EMDB-18664: 
Structure of the native microtubule lattice nucleated from the yeast spindle pole body

EMDB-18665: 
Structure of the native y-Tubulin Ring Complex (yTuRC) capping microtubule minus ends at the spindle pole body

EMDB-18666: 
Structure of the y-Tubulin Small Complex (yTuSC) as part of the native y-Tubulin Ring Complex (yTuRC) capping microtubule minus ends at the spindle pole body

PDB-8qv0: 
Structure of the native microtubule lattice nucleated from the yeast spindle pole body

PDB-8qv2: 
Structure of the native y-Tubulin Ring Complex (yTuRC) capping microtubule minus ends at the spindle pole body

PDB-8qv3: 
Structure of the y-Tubulin Small Complex (yTuSC) as part of the native y-Tubulin Ring Complex (yTuRC) capping microtubule minus ends at the spindle pole body

EMDB-19861: 
Vertebrate microtubule-capping gamma-tubulin ring complex

PDB-9eoj: 
Vertebrate microtubule-capping gamma-tubulin ring complex
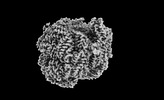
EMDB-41434: 
Bottom cylinder of high-resolution phycobilisome quenched by OCP (local refinement)
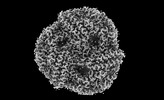
EMDB-41435: 
Central rod disk in C1 symmetry of high-resolution phycobilisome quenched by OCP (local refinement)

EMDB-41436: 
Central rod disk in D3 symmetry of high-resolution phycobilisome quenched by OCP (local refinement)
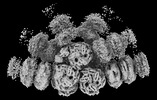
EMDB-41463: 
Synechocystis PCC 6803 Phycobilisome quenched by OCP, high resolution

EMDB-41475: 
Top cylinder bound to OCP from high-resolution phycobilisome quenched by OCP (local refinement)
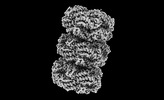
EMDB-41585: 
Rod from high-resolution phycobilisome quenched by OCP (local refinement)

PDB-8to2: 
Bottom cylinder of high-resolution phycobilisome quenched by OCP (local refinement)

PDB-8to5: 
Central rod disk in C1 symmetry of high-resolution phycobilisome quenched by OCP (local refinement)

PDB-8tpj: 
Top cylinder bound to OCP from high-resolution phycobilisome quenched by OCP (local refinement)

PDB-8tro: 
Rod from high-resolution phycobilisome quenched by OCP (local refinement)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
