-検索条件
-検索結果
検索 (著者・登録者: leiman & pg)の結果全50件を表示しています

EMDB-29383: 
Structure of baseplate with receptor binding complex of Agrobacterium phage Milano

EMDB-29353: 
Structure of Agrobacterium tumefaciens bacteriophage Milano curved tail

EMDB-29354: 
Structure of Agrobacterium tumefaciens bacteriophage Milano contracted tail-tube

EMDB-29355: 
Structure of Agrobacterium tumefaciens bacteriophage Milano contracted tail-sheath
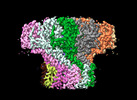
EMDB-29500: 
Portal assembly of Agrobacterium phage Milano
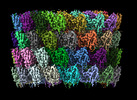
EMDB-29501: 
Collar sheath structure of Agrobacterium phage Milano

EMDB-29503: 
Neck structure of Agrobacterium phage Milano, C3 symmetry
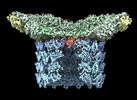
EMDB-29504: 
Structure of neck and portal vertex of Agrobacterium phage Milano, C5 symmetry

EMDB-29512: 
Structure of tail-neck junction of Agrobacterium phage Milano

EMDB-29540: 
Structure of capsid of Agrobacterium phage Milano
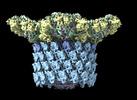
EMDB-29541: 
Structure of neck with portal vertex of capsid of Agrobacterium phage Milano

EMDB-25443: 
CryoEM map of the manually-picked stalled contraction intermediate state of bacteriophage A511.

EMDB-25444: 
CryoEM map of the CNN-picked stalled contraction intermediate state of bacteriophage A511.

EMDB-24763: 
Cryo-EM map of the phage AR9 non-virion RNA polymerase holoenzyme in complex with DNA containing the AR9 P077 promoter
EMDB-24765: 
Cryo-EM map of the phage AR9 non-virion RNA polymerase holoenzyme

EMDB-20526: 
CryoEM Structure of Pyocin R2 - precontracted - trunk

EMDB-20643: 
CryoEM Structure of Pyocin R2 - precontracted - baseplate

EMDB-20644: 
CryoEM Structure of Pyocin R2 - precontracted - collar

EMDB-20646: 
CryoEM Structure of Pyocin R2 - precontracted - hub

EMDB-20647: 
CryoEM Structure of Pyocin R2 - postcontracted - collar

EMDB-20648: 
CryoEM Structure of Pyocin R2 - postcontracted - baseplate

EMDB-7559: 
Bacteriophage A511 baseplate in post-host attachment state (urea-induced)

EMDB-7560: 
Bacteriophage A511 baseplate in pre-host attachment state

EMDB-7561: 
Bacteriophage A511 baseplate in post-host attachment state

EMDB-8767: 
Cryo-EM structure of the T4 tail tube

EMDB-8396: 
Asymmetric reconstruction of bacteriophage MS2 mutant NEO1

EMDB-8360: 
Asymmetric reconstruction of bacteriophage MS2

EMDB-3374: 
Cryo-electron microscopy structure of the hexagonal pre-attachment T4 baseplate-tail tube complex

EMDB-3392: 
Cryo-electron microscopy structure of the hexagonal pre-attachment T4 baseplate-tail tube complex: locally masked refinement of the inner baseplate

EMDB-3393: 
Cryo-electron microscopy structure of the hexagonal pre-attachment T4 baseplate-tail tube complex: locally masked refinement of the intermediate baseplate

EMDB-3394: 
Cryo-electron microscopy structure of the hexagonal pre-attachment T4 baseplate-tail tube complex: locally masked refinement of the upper peripheral baseplate

EMDB-3395: 
Cryo-electron microscopy structure of the hexagonal pre-attachment T4 baseplate-tail tube complex: locally masked refinement of the lower peripheral baseplate

EMDB-3396: 
Cryo-electron microscopy structure of the star-shaped, hubless post-attachment T4 baseplate

EMDB-3397: 
Cryo-electron microscopy structure of the hexagonal pre-attachment T4 baseplate-tail tube complex: locally masked refinement of the tail tube

EMDB-6270: 
Atomic structures of a bactericidal contractile nanotube in its pre- and post-contraction states

EMDB-6271: 
Atomic structures of a bactericidal contractile nanotube in its pre- and post-contraction states

EMDB-5409: 
A 3-D cryo-electron structure of bacteriophage phi92 contractile tail

EMDB-2063: 
A 3-D cryo-electron microscopy structure of bacteriophage phi92 capsid

EMDB-2064: 
A 3-D cryo-electron structure of bacteriophage phi92 baseplate

EMDB-1414: 
Molecular architecture of the prolate head of bacteriophage T4.

EMDB-1333: 
The structures of bacteriophages K1E and K1-5 explain processive degradation of polysaccharide capsules and evolution of new host specificities.

EMDB-1334: 
The structures of bacteriophages K1E and K1-5 explain processive degradation of polysaccharide capsules and evolution of new host specificities.

EMDB-1335: 
The structures of bacteriophages K1E and K1-5 explain processive degradation of polysaccharide capsules and evolution of new host specificities.

EMDB-1336: 
The structures of bacteriophages K1E and K1-5 explain processive degradation of polysaccharide capsules and evolution of new host specificities.

EMDB-1337: 
The structures of bacteriophages K1E and K1-5 explain processive degradation of polysaccharide capsules and evolution of new host specificities.

EMDB-1126: 
The tail structure of bacteriophage T4 and its mechanism of contraction.

EMDB-1086: 
Three-dimensional rearrangement of proteins in the tail of bacteriophage T4 on infection of its host.

EMDB-1089: 
Three-dimensional rearrangement of proteins in the tail of bacteriophage T4 on infection of its host.

EMDB-1075: 
Molecular architecture of the prolate head of bacteriophage T4.

EMDB-1048: 
Three-dimensional structure of bacteriophage T4 baseplate.
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
