[English] 日本語
 Yorodumi
Yorodumi- EMDB-6270: Atomic structures of a bactericidal contractile nanotube in its p... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-6270 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Atomic structures of a bactericidal contractile nanotube in its pre- and post-contraction states | |||||||||
 Map data Map data | Helical reconstruction of pyocin trunk | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | pyocin / bacteriocin / sheath / tube | |||||||||
| Function / homology |  Function and homology information Function and homology information | |||||||||
| Biological species |  | |||||||||
| Method | helical reconstruction / cryo EM / Resolution: 3.5 Å | |||||||||
 Authors Authors | Ge P / Scholl D / Leiman PG / Yu X / Miller JF / Zhou ZH | |||||||||
 Citation Citation |  Journal: Nat Struct Mol Biol / Year: 2015 Journal: Nat Struct Mol Biol / Year: 2015Title: Atomic structures of a bactericidal contractile nanotube in its pre- and postcontraction states. Authors: Peng Ge / Dean Scholl / Petr G Leiman / Xuekui Yu / Jeff F Miller / Z Hong Zhou /   Abstract: R-type pyocins are representatives of contractile ejection systems, a class of biological nanomachines that includes, among others, the bacterial type VI secretion system (T6SS) and contractile ...R-type pyocins are representatives of contractile ejection systems, a class of biological nanomachines that includes, among others, the bacterial type VI secretion system (T6SS) and contractile bacteriophage tails. We report atomic models of the Pseudomonas aeruginosa precontraction pyocin sheath and tube, and the postcontraction sheath, obtained by cryo-EM at 3.5-Å and 3.9-Å resolutions, respectively. The central channel of the tube is negatively charged, in contrast to the neutral and positive counterparts in T6SSs and phage tails. The sheath is interwoven by long N- and C-terminal extension arms emanating from each subunit, which create an extensive two-dimensional mesh that has the same connectivity in the extended and contracted state of the sheath. We propose that the contraction process draws energy from electrostatic and shape complementarities to insert the inner tube through bacterial cell membranes to eventually kill the bacteria. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_6270.map.gz emd_6270.map.gz | 267 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-6270-v30.xml emd-6270-v30.xml emd-6270.xml emd-6270.xml | 10.1 KB 10.1 KB | Display Display |  EMDB header EMDB header |
| Images |  400_6270.gif 400_6270.gif 80_6270.gif 80_6270.gif | 66.8 KB 3.6 KB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6270 http://ftp.pdbj.org/pub/emdb/structures/EMD-6270 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6270 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6270 | HTTPS FTP |
-Related structure data
| Related structure data |  3j9qMC  5w5eM  6271C  3j9rC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_6270.map.gz / Format: CCP4 / Size: 276 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_6270.map.gz / Format: CCP4 / Size: 276 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Helical reconstruction of pyocin trunk | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.104 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
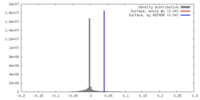
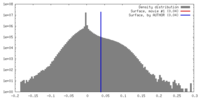
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
- Sample components
Sample components
-Entire : Pyocin (pre-contraction)
| Entire | Name: Pyocin (pre-contraction) |
|---|---|
| Components |
|
-Supramolecule #1000: Pyocin (pre-contraction)
| Supramolecule | Name: Pyocin (pre-contraction) / type: sample / ID: 1000 / Oligomeric state: helical / Number unique components: 2 |
|---|
-Macromolecule #1: pyocin tail sheath
| Macromolecule | Name: pyocin tail sheath / type: protein_or_peptide / ID: 1 / Name.synonym: sheath / Oligomeric state: helix of hexameric rings / Recombinant expression: No / Database: NCBI |
|---|---|
| Source (natural) | Organism:  |
| Sequence | UniProtKB: Similar to FI genes of P2, phiCTX, and PS17: tail sheath |
-Macromolecule #2: pyocin tube
| Macromolecule | Name: pyocin tube / type: protein_or_peptide / ID: 2 / Oligomeric state: helix of hexameric rings / Recombinant expression: No / Database: NCBI |
|---|---|
| Source (natural) | Organism:  |
| Sequence | UniProtKB: FIIR2 protein |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | helical reconstruction |
| Aggregation state | helical array |
- Sample preparation
Sample preparation
| Buffer | pH: 7.4 / Details: 1x PBS |
|---|---|
| Grid | Details: baked 1.2/1.3 Quantifoil |
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 90 K / Instrument: FEI VITROBOT MARK IV / Method: blot time 4 seconds, blot force 1 |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Temperature | Average: 80 K |
| Details | Scanned with 9200 ED |
| Date | Oct 7, 2009 |
| Image recording | Category: FILM / Film or detector model: KODAK SO-163 FILM / Digitization - Scanner: NIKON SUPER COOLSCAN 9000 / Digitization - Sampling interval: 6.35 µm / Number real images: 307 / Average electron dose: 25 e/Å2 / Bits/pixel: 16 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.5 µm / Nominal defocus min: 1.6 µm / Nominal magnification: 59000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
- Image processing
Image processing
| Details | Relion-based IHRSR |
|---|---|
| Final reconstruction | Applied symmetry - Helical parameters - Δz: 38.4 Å Applied symmetry - Helical parameters - Δ&Phi: 18.3 ° Applied symmetry - Helical parameters - Axial symmetry: C6 (6 fold cyclic) Algorithm: OTHER / Resolution.type: BY AUTHOR / Resolution: 3.5 Å / Resolution method: OTHER / Software - Name: Relion, IHRSR |
| CTF correction | Details: Each particle |
 Movie
Movie Controller
Controller









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)