-検索条件
-検索結果
検索 (著者・登録者: hagen & wj)の結果124件中、1から50件目までを表示しています

EMDB-16454: 
Electron cryo-tomography of HeLa cells expressing untagged Cidec

EMDB-16455: 
Electron cryo-tomography of HeLa cells expressing Cidec-EGFP

EMDB-14325: 
Cytoplasmic ring of the human nuclear pore complex from isolated HeLa nuclear envelopes

EMDB-14326: 
Structure of nuclear pore complex from HEK cells with GP210 knockout

EMDB-14327: 
Nuclear pore complex from intact HeLa cells
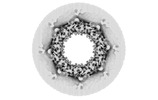
EMDB-14328: 
Inner/spoke ring of the human nuclear pore complex from isolated HeLa nuclear envelopes.

EMDB-14330: 
Nuclear ring of the human nuclear pore complex from isolated HeLa nuclear envelopes

EMDB-14321: 
Human nuclear pore complex (dilated)

EMDB-14322: 
Human nuclear pore complex (constricted)

EMDB-14243: 
cryoEM structure of human Nup155 (residues 19-981)

EMDB-14032: 
Structure of the EIAV CA-SP2 hexamer (C2), acquired on Krios G4, Selectris/Falcon4, processed with Relion/M

EMDB-14034: 
Structure of the EIAV CA-SP hexamer (C2), acquired on Krios 3Gi/Gatan Bioquantum K3, processed with Relion/M

EMDB-14033: 
Structure of the EIAV CA-SP hexamer (C6), acquired on Krios 3Gi/Bioquantum K3, processed with Relion/M

EMDB-14035: 
Structure of the EIAV CA-SP hexamer (C6), acquired on Krios4, Selectris/Falcon4, processed with AV3

EMDB-14036: 
Cryo-electron tomogram of EIAV CASPNC VLPs, acquired on Krios G4, Selectris/Falcon4

EMDB-14037: 
Cryo-electron tomogram of EIAV CASPNC VLPs, acquired on Krios 3Gi, Bioquantum K3

EMDB-14031: 
Structure of the EIAV CA-SP hexamer (C6), acquired on a Krios 4, Selectris/Falcon4, processed with Relion/M

EMDB-11222: 
Structure of SARS-CoV-2 spike glycoprotein (S) trimer determined by sub-tomogram averaging

EMDB-11223: 
Structure of SARS-CoV-2 spike glycoprotein (S) monomer in a closed conformation determined by sub-tomogram averaging

EMDB-22190: 
Subtomogram averaging map of yeast polysome

EMDB-22196: 
Cryo-EM Structure of K63R Ubiquitin Mutant Ribosome under Oxidative Stress

EMDB-22197: 
Cryo-EM Structure of K63R Ubiquitin Mutant Consensus Ribosome under Oxidative Stress

EMDB-22198: 
Cryo-EM Structure of K63 Ubiquitinated Yeast Translocating Ribosome under Oxidative Stress

EMDB-10677: 
in-cell 70S ribosome from Mycoplasma pneumoniae

EMDB-10678: 
in-cell elongating expressome from Mycoplasma pneumoniae with focused refinement on RNAP

EMDB-10679: 
in-cell elongating expressome from Mycoplasma pneumoniae

EMDB-10680: 
Multibody refinement of RNA polymerase-NusA body of Mycoplasma pneumoniae in-cell expressome

EMDB-10681: 
Multibody refinement of 30S ribosome body of Mycoplasma pneumoniae in-cell expressome

EMDB-10682: 
Multibody refinement of 50S ribosome body of Mycoplasma pneumoniae in-cell expressome

EMDB-10683: 
70S ribosome from chloramphenicol-treated Mycoplasma pneumoniae cells

EMDB-10684: 
in-cell stalled expressome from pseudouridimycin-treated Mycoplasma pneumoniae

EMDB-10685: 
Multibody refinement of RNA polymerase body of pseudouridimycin-stalled Mycoplasma pneumoniae in-cell expressome

EMDB-10686: 
Multibody refinement of 30S ribosome body of pseudouridimycin-stalled Mycoplasma pneumoniae in-cell expressome

EMDB-10687: 
Multibody refinement of 50S ribosome body of pseudouridimycin-stalled Mycoplasma pneumoniae in-cell expressome

EMDB-10207: 
A 4.2 Angstrom structure of HIV-1 CA-SP1

EMDB-10595: 
Cryo-EM structure of the RNA Polymerase III-Maf1 complex

EMDB-10500: 
Cryo-EM structure of AtNBR1-PB1 filament (S-type)

EMDB-10499: 
Cryo-EM structure of AtNBR1-PB1 filament (L-type)

EMDB-10501: 
Cryo-EM structure of p62-PB1 filament (L-type)

EMDB-10502: 
Cryo-EM structure of p62-PB1 filament (L-type)

EMDB-0153: 
RELION-3.0 reconstruction of beta-galactosidase particles in EMPIAR-10061

EMDB-0263: 
Horse spleen apo-ferritin with beam tilt correction in RELION-3.0

EMDB-0307: 
Representative tomogram of the COPII in vitro reconstitution used for subtomogram averaging

EMDB-0144: 
Human apo-ferritin reconstructed in RELION-3.0

EMDB-0152: 
RELION-3.0 reconstruction of influenza hemagglutinin (HA) trimer using particles from micrographs tilted at 40 degrees in EMPIAR-10097

EMDB-4184: 
RNA Polymerase III pre-initiation complex

EMDB-4180: 
RNA Polymerase III open complex

EMDB-4181: 
RNA Polymerase III initially transcribing complex

EMDB-4182: 
RNA Polymerase III closed complex CC1

EMDB-4183: 
RNA Polymerase III closed complex CC2
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
