[English] 日本語
 Yorodumi
Yorodumi- PDB-5zpq: Copper amine oxidase from Arthrobacter globiformis anaerobically ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5zpq | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Copper amine oxidase from Arthrobacter globiformis anaerobically reduced by phenylethylamine at pH 9 at 288 K (1) | |||||||||
 Components Components | Phenylethylamine oxidase | |||||||||
 Keywords Keywords | OXIDOREDUCTASE / COPPER AMINE OXIDASE / TOPAQUINONE / TPQ | |||||||||
| Function / homology |  Function and homology information Function and homology informationprimary-amine oxidase / primary methylamine oxidase activity / amine metabolic process / quinone binding / copper ion binding Similarity search - Function | |||||||||
| Biological species |  Arthrobacter globiformis (bacteria) Arthrobacter globiformis (bacteria) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.849 Å MOLECULAR REPLACEMENT / Resolution: 1.849 Å | |||||||||
 Authors Authors | Murakawa, T. / Baba, S. / Kawano, Y. / Hayashi, H. / Yano, T. / Tanizawa, K. / Kumasaka, T. / Yamamoto, M. / Okajima, T. | |||||||||
| Funding support |  Japan, 2items Japan, 2items
| |||||||||
 Citation Citation |  Journal: Proc. Natl. Acad. Sci. U.S.A. / Year: 2019 Journal: Proc. Natl. Acad. Sci. U.S.A. / Year: 2019Title: In crystallothermodynamic analysis of conformational change of the topaquinone cofactor in bacterial copper amine oxidase. Authors: Murakawa, T. / Baba, S. / Kawano, Y. / Hayashi, H. / Yano, T. / Kumasaka, T. / Yamamoto, M. / Tanizawa, K. / Okajima, T. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5zpq.cif.gz 5zpq.cif.gz | 514.7 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5zpq.ent.gz pdb5zpq.ent.gz | 416.8 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5zpq.json.gz 5zpq.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/zp/5zpq https://data.pdbj.org/pub/pdb/validation_reports/zp/5zpq ftp://data.pdbj.org/pub/pdb/validation_reports/zp/5zpq ftp://data.pdbj.org/pub/pdb/validation_reports/zp/5zpq | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5zouC  5zowC  5zoxC  5zoyC  5zozC  5zp0C  5zp1C  5zp2C  5zp3C  5zp4C  5zp5C  5zp6C  5zp7C  5zp8C  5zp9C  5zpaC  5zpbC  5zpcC  5zpdC  5zpeC  5zpfC  5zpgC  5zphC  5zpiC  5zpjC  5zpkC  5zplC  5zpmC  5zpnC  5zpoC  5zppC  5zprC  5zpsC  5zptC  1iu7S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||
| Unit cell |
| ||||||||||||||||||||||||
| Components on special symmetry positions |
|
- Components
Components
-Protein , 1 types, 2 molecules AB
| #1: Protein | Mass: 68913.750 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Arthrobacter globiformis (bacteria) / Plasmid: pepo-2 / Production host: Arthrobacter globiformis (bacteria) / Plasmid: pepo-2 / Production host:  |
|---|
-Non-polymers , 5 types, 1018 molecules 

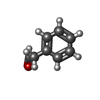






| #2: Chemical | | #3: Chemical | #4: Chemical | #5: Chemical | #6: Water | ChemComp-HOH / | |
|---|
-Details
| Has protein modification | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.18 Å3/Da / Density % sol: 61.29 % |
|---|---|
| Crystal grow | Temperature: 289 K / Method: microdialysis / pH: 7.4 / Details: 1.05M potassium-sodium tartrate, 25mM HEPES |
-Data collection
| Diffraction | Mean temperature: 288 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SPring-8 SPring-8  / Beamline: BL38B1 / Wavelength: 1 Å / Beamline: BL38B1 / Wavelength: 1 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: RAYONIX MX225HE / Detector: CCD / Date: Nov 8, 2015 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 1.85→50 Å / Num. obs: 148084 / % possible obs: 99.9 % / Redundancy: 3.5 % / Biso Wilson estimate: 19.88 Å2 / Rmerge(I) obs: 0.096 / Rpim(I) all: 0.06 / Rrim(I) all: 0.113 / Χ2: 1.83 / Net I/σ(I): 7.3 / Num. measured all: 521039 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 1IU7 Resolution: 1.849→39.673 Å / SU ML: 0.17 / Cross valid method: THROUGHOUT / σ(F): 1.96 / Phase error: 17.49 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 171.04 Å2 / Biso mean: 24.753 Å2 / Biso min: 6.82 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.849→39.673 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0 / Total num. of bins used: 30
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller






















 PDBj
PDBj



