-検索条件
-検索結果
検索 (著者・登録者: ni & f)の結果15,291件中、1から50件目までを表示しています

EMDB-43706: 
Human DNA polymerase theta helicase domain in complex with ssDNA, dimer form

EMDB-43816: 
Human DNA polymerase theta helicase domain in complex with AMP-PNP, dimer form

EMDB-43817: 
Human DNA polymerase theta helicase domain dimer, apo-form

EMDB-43818: 
Human DNA polymerase theta helicase domain tetramer, apo-form

EMDB-45217: 
Human DNA polymerase theta helicase domain in microhomology annealed state 1, dimer form

EMDB-45218: 
Human DNA polymerase theta helicase domain in microhomology annealed state 2, dimer form

PDB-8w0a: 
Human DNA polymerase theta helicase domain in complex with ssDNA, dimer form

PDB-9asj: 
Human DNA polymerase theta helicase domain in complex with AMP-PNP, dimer form

PDB-9ask: 
Human DNA polymerase theta helicase domain dimer, apo-form

PDB-9asl: 
Human DNA polymerase theta helicase domain tetramer, apo-form

PDB-9c5q: 
Human DNA polymerase theta helicase domain in microhomology annealed state 2, dimer form

EMDB-18881: 
AL amyloid fibril from the FOR010 light chain

EMDB-19818: 
AL amyloid fibril from the FOR103 light chain

PDB-8r47: 
AL amyloid fibril from the FOR010 light chain

PDB-9eme: 
AL amyloid fibril from the FOR103 light chain

EMDB-43088: 
Cryogenic electron microscopy structure of human serum albumin in complex with teniposide

EMDB-43089: 
Cryogenic electron microscopy structure of human serum albumin in complex with salicylic acid

EMDB-43090: 
Cryogenic electron microscopy structure of apo human serum albumin

PDB-8vac: 
Cryogenic electron microscopy structure of human serum albumin in complex with teniposide

PDB-8vae: 
Cryogenic electron microscopy structure of human serum albumin in complex with salicylic acid

PDB-8vaf: 
Cryogenic electron microscopy structure of apo human serum albumin

EMDB-50296: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

PDB-9fbv: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

EMDB-50358: 
In vitro-induced genome-releasing intermediate of Rhodobacter microvirus Ebor computed with C5 symmetry

EMDB-19548: 
cryoEM structure of Acs1 filament determined by FilamentID

EMDB-19549: 
cryoEM structure of the central Ald4 filament determined by FilamentID

EMDB-19550: 
cryoEM structure of purified Acs1 filament from meiotic yeast cells
EMDB-19551: 
cryo sub-tomogram average of Acs1 filament from spread meiotic yeast spheroplasts
EMDB-19552: 
cryo sub-tomogram average of Ald4 filaments from purified meiotic yeast mitochondria
EMDB-19553: 
cryo-tomogram of FIB-milled meiotic yeast cell containing filaments in mitochondria

EMDB-19554: 
cryo-tomogram of FIB-milled meiotic yeast cell containing filaments in the nucleus
EMDB-19555: 
cryo-tomogram of FIB-milled meiotic yeast cell containing filaments in the cytoplasm
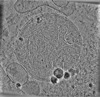
EMDB-19556: 
cryo-tomogram of purified meiotic yeast mitochondrion containing Ald4 filaments
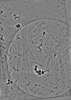
EMDB-19557: 
cryo-tomogram of spread meiotic yeast spheroplast containing Acs1 filaments

EMDB-19558: 
cryo-tomogram of spread starved yeast spheroplast containing filaments

PDB-8rwj: 
cryoEM structure of Acs1 filament determined by FilamentID

PDB-8rwk: 
cryoEM structure of the central Ald4 filament determined by FilamentID

EMDB-36635: 
Structure of arginine oxidase from Pseudomonas sp. TRU 7192

PDB-8jt7: 
Structure of arginine oxidase from Pseudomonas sp. TRU 7192

EMDB-17582: 
Cryo-EM structure of Caenorhabditis elegans DPF-3 (apo)

PDB-8pba: 
Cryo-EM structure of Caenorhabditis elegans DPF-3 (apo)

EMDB-35116: 
Cryo-EM structure of 6-subunit Smc5/6

EMDB-35128: 
Cryo-EM structure of 6-subunit Smc5/6 arm region

EMDB-35187: 
Cryo-EM structure of 5-subunit Smc5/6

EMDB-60245: 
Smc5/6-5mer consensus map

EMDB-60246: 
Smc5/6-6mer consensus map

PDB-8i21: 
Cryo-EM structure of 6-subunit Smc5/6 arm region

EMDB-50090: 
Vibrio cholerae DdmD apo complex

PDB-9ezx: 
Vibrio cholerae DdmD apo complex

EMDB-19395: 
CryoEM structure of recombinant human Bri2 BRICHOS oligomers
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
