-検索条件
-検索結果
検索 (著者・登録者: rosenbaum & e)の結果全45件を表示しています

EMDB-50518: 
LGTV with TBEV prME

EMDB-50624: 
LGTV TP21. Langat virus, strain TP21

EMDB-28164: 
Structure of FFAR1-Gq complex bound to DHA

EMDB-28177: 
Structure of FFAR1-Gq complex bound to TAK-875

EMDB-28185: 
Structure of FFAR1-Gq complex bound to TAK-875 in a lipid nanodisc

PDB-8eit: 
Structure of FFAR1-Gq complex bound to DHA

PDB-8ejc: 
Structure of FFAR1-Gq complex bound to TAK-875

PDB-8ejk: 
Structure of FFAR1-Gq complex bound to TAK-875 in a lipid nanodisc

EMDB-25399: 
Molecular mechanism of the the wake-promoting agent TAK-925

PDB-7sr8: 
Molecular mechanism of the the wake-promoting agent TAK-925

EMDB-25389: 
Structure of the orexin-2 receptor (OX2R) bound to TAK-925, Gi and scFv16

PDB-7sqo: 
Structure of the orexin-2 receptor(OX2R) bound to TAK-925, Gi and scFv16

EMDB-23405: 
WT Chicken Scap L1-L7 / Fab 4G10 complex focused refinement

EMDB-23406: 
Map of WT cScap/Fab complex

EMDB-23407: 
Map of full mutant cScap/Fab complex

EMDB-23408: 
Chicken Scap D435V L1-L7 domain / Fab complex focused map

PDB-7lkf: 
WT Chicken Scap L1-L7 / Fab 4G10 complex focused refinement

PDB-7lkh: 
Chicken Scap D435V L1-L7 domain / Fab complex focused map

EMDB-30392: 
Cryo-EM structure of Fenoldopam bound dopamine receptor DRD1-Gs signaling complex

EMDB-30393: 
Cryo-EM structure of A77636 bound dopamine receptor DRD1-Gs signaling complex

EMDB-30394: 
Cryo-EM structure of PW0464 bound dopamine receptor DRD1-Gs signaling complex

EMDB-30395: 
Cryo-EM structure of Dopamine and LY3154207 bound dopamine receptor DRD1-Gs signaling complex

EMDB-30452: 
Cryo-EM structure of SKF83959 bound dopamine receptor DRD1-Gs signaling complex

PDB-7ckw: 
Cryo-EM structure of Fenoldopam bound dopamine receptor DRD1-Gs signaling complex

PDB-7ckx: 
Cryo-EM structure of A77636 bound dopamine receptor DRD1-Gs signaling complex

PDB-7cky: 
Cryo-EM structure of PW0464 bound dopamine receptor DRD1-Gs signaling complex

PDB-7ckz: 
Cryo-EM structure of Dopamine and LY3154207 bound dopamine receptor DRD1-Gs signaling complex

PDB-7crh: 
Cryo-EM structure of SKF83959 bound dopamine receptor DRD1-Gs signaling complex

EMDB-23211: 
Cryo-EM structure of human ACE2 receptor bound to protein encoded by vaccine candidate BNT162b1

EMDB-23215: 
Cryo-EM structure of protein encoded by vaccine candidate BNT162b2

PDB-7l7f: 
Cryo-EM structure of human ACE2 receptor bound to protein encoded by vaccine candidate BNT162b1

PDB-7l7k: 
Cryo-EM structure of protein encoded by vaccine candidate BNT162b2

EMDB-21243: 
Structure of a D2 dopamine receptor-G-protein complex in a lipid membrane

EMDB-21244: 
Structure of a D2 dopamine receptor-G-protein complex in a lipid membrane

EMDB-21245: 
Structure of a D2 dopamine receptor-G-protein complex in a lipid membrane

PDB-6vms: 
Structure of a D2 dopamine receptor-G-protein complex in a lipid membrane

EMDB-4584: 
Structure and assembly of the mitochondrial membrane remodelling GTPase Mgm1
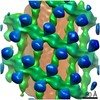
EMDB-10062: 
Structure of s-Mgm1 decorating the outer surface of tubulated lipid membranes
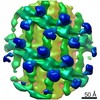
EMDB-10063: 
Structure of s-Mgm1 decorating the outer surface of tubulated lipid membranes in the GTPgammaS bound state

EMDB-10064: 
Structure of s-Mgm1 decorating the inner surface of tubulated lipid membranes

EMDB-10065: 
Structure of s-Mgm1 decorating the inner surface of tubulated lipid membranes in the GTPgammaS bound state

PDB-6rzt: 
Structure of s-Mgm1 decorating the outer surface of tubulated lipid membranes

PDB-6rzu: 
Structure of s-Mgm1 decorating the outer surface of tubulated lipid membranes in the GTPgammaS bound state

PDB-6rzv: 
Structure of s-Mgm1 decorating the inner surface of tubulated lipid membranes

PDB-6rzw: 
Structure of s-Mgm1 decorating the inner surface of tubulated lipid membranes in the GTPgammaS bound state
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
