-検索条件
-検索結果
検索 (著者・登録者: lie & tj)の結果71件中、1から50件目までを表示しています
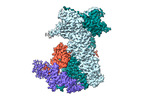
EMDB-18498: 
Cryo-EM structure of the benzo[a]pyrene-bound Hsp90-XAP2-AHR complex

EMDB-28850: 
SARS-CoV-2 Gamma 6P Mut7 S + COVA309-3 Fab

EMDB-28851: 
SARS-CoV-2 Gamma 6P Mut7 S + COVA309-10 Fab

EMDB-28852: 
SARS-CoV-2 Omicron 6P S + COVA309-35 Fab

EMDB-28853: 
SARS-CoV-2 Gamma 6P Mut7 + S COVA309-38 Fab

EMDB-15786: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli (Apo form)

EMDB-15787: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with PE
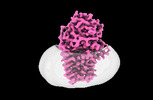
EMDB-15788: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with PE (C387S mutant)

EMDB-15789: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with Lyso-PE

EMDB-15790: 
Cryo-EM structure apolipoprotein N-acyltransferase Lnt from E.coli in complex with FP3
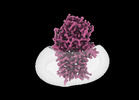
EMDB-15791: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with Pam3

PDB-8b0k: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli (Apo form)

PDB-8b0l: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with PE

PDB-8b0m: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with PE (C387S mutant)

PDB-8b0n: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with Lyso-PE

PDB-8b0o: 
Cryo-EM structure apolipoprotein N-acyltransferase Lnt from E.coli in complex with FP3

PDB-8b0p: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with Pam3

EMDB-25100: 
Unmethylated Mtb Ribosome 50S with SEQ-9
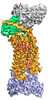
EMDB-25171: 
Cryo-EM structure of ACKR3 in complex with CXCL12, an intracellular Fab, and an extracellular Fab

EMDB-25172: 
Cryo-EM structure of ACKR3 in complex with chemokine N-terminal mutant CXCL12_LRHQ, an intracellular Fab, and an extracellular Fab
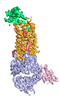
EMDB-25173: 
Cryo-EM structure of ACKR3 in complex with CXCL12 and an intracellular Fab
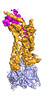
EMDB-25174: 
Cryo-EM structure of human ACKR3 in complex with chemokine N-terminal mutant CXCL12_LRHQ and an intracellular Fab
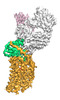
EMDB-25175: 
Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, and an extracellular Fab
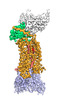
EMDB-25176: 
Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, an extracellular Fab, and an intracellular Fab
EMDB-25177: 
Cryo-EM structure of human ACKR3 in complex with a small molecule partial agonist CCX662, and an intracellular Fab
EMDB-26217: 
Negative stain EM map of COVA1-07 mAb bound to the S2 domain of SARS-CoV-2 S
EMDB-26218: 
Negative stain EM map of COVA2-14 mAb bound to the S2 domain of SARS-CoV-2 S

EMDB-26219: 
Negative stain EM map of COVA2-18 mAb bound to the S2 domain of SARS-CoV-2 S
EMDB-26220: 
Negative stain EM map of the S2 domain of SARS-CoV-2 S

EMDB-22865: 
CryoEM structure of A2296-methylated Mycobacterium tuberculosis ribosome bound with SEQ-9

EMDB-25792: 
Cryo-EM structure of the spike of SARS-CoV-2 Omicron variant of concern

EMDB-23914: 
Cryo-EM structure of SARS-CoV-2 spike in complex with neutralizing antibody B1-182.1 that targets the receptor-binding domain

EMDB-23915: 
Cryo-EM structure of SARS-CoV-2 spike in complex with neutralizing antibody B1-182.1 that targets the receptor-binding domain

EMDB-23498: 
Cryo-EM structure of SARS-CoV-2 spike in complex with neutralizing antibody A23-58.1 that targets the receptor-binding domain

EMDB-23499: 
Cryo-EM structure of SARS-CoV-2 spike in complex with neutralizing antibody A23-58.1 that targets the receptor-binding domain

EMDB-10223: 
Cryo-EM Structure of T. kodakarensis 70S ribosome

EMDB-10224: 
Cryo-EM Structure of T. kodakarensis 70S ribosome in TkNat10 deleted strain

EMDB-10503: 
Cryo-EM Structure of T. kodakarensis 70S ribosome
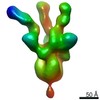
EMDB-22061: 
negative stain EM map of SARS-CoV-2 spike in complex with COVA2-15 Fab

EMDB-22062: 
negative stain EM map of SARS-CoV-2 spike in complex with COVA1-22

EMDB-22063: 
negative stain EM map of SARS-CoV-2 spike in complex with COVA2-07

EMDB-22064: 
negative stain EM map of SARS-CoV-2 spike in complex with COVA2-39 Fab

EMDB-22065: 
negative stain EM map of SARS-CoV-2 spike in complex with COVA2-04 Fab

EMDB-22066: 
negative stain EM map of SARS-CoV-2 spike in complex with COVA1-12 Fab

EMDB-4584: 
Structure and assembly of the mitochondrial membrane remodelling GTPase Mgm1
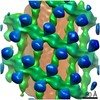
EMDB-10062: 
Structure of s-Mgm1 decorating the outer surface of tubulated lipid membranes
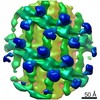
EMDB-10063: 
Structure of s-Mgm1 decorating the outer surface of tubulated lipid membranes in the GTPgammaS bound state

EMDB-10064: 
Structure of s-Mgm1 decorating the inner surface of tubulated lipid membranes

EMDB-10065: 
Structure of s-Mgm1 decorating the inner surface of tubulated lipid membranes in the GTPgammaS bound state

PDB-6rzt: 
Structure of s-Mgm1 decorating the outer surface of tubulated lipid membranes
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
