-検索条件
-検索結果
検索 (著者・登録者: kelley & l)の結果53件中、1から50件目までを表示しています

EMDB-16537: 
Optimizing Cryo-FIB Lamellas for sub-5 Angstrom in situ Structural Biology : Subtomogram average of the Large Subunit of S.Cerevisiae 80S Ribosome
EMDB-41617: 
CryoEM structure of PI3Kalpha

PDB-8tu6: 
CryoEM structure of PI3Kalpha
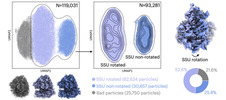
EMDB-18231: 
CryoDRGN Reconstruction of 80S Ribosome in Rotated State from S.cerevisiae Prepared using Cryo-plasmaFIB Milling
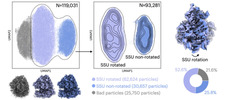
EMDB-18232: 
CryoDRGN Reconstruction of 80S Ribosome in Unrotated State from S.cerevisiae Prepared using Cryo-plasmaFIB Milling
EMDB-18197: 
Subtomogram average of Ribosome from S.cerevisiae prepared using cryo-plasmaFIB milling

EMDB-27847: 
Mouse norovirus strain CR6, attenuated

EMDB-27849: 
Mouse norovirus strain CR6 at pH 5.0

EMDB-27319: 
Mouse Norovirus strain WU23

EMDB-27321: 
Mouse norovirus strain WU23 + 10mM GCDCA

EMDB-27322: 
Murine norovirus strain WU23 at pH 5

EMDB-25035: 
Cryo-EM 3D map of the human Exostosin-1 and Exostosin-2 heterodimer

EMDB-25036: 
Cryo-EM 3D map of the human Exostosin-1 and Exostosin-2 heterodimer in complex with a 4-sugar oligosaccharide acceptor analog

EMDB-25037: 
Cryo-EM 3D map of the human Exostosin-1 and Exostosin-2 heterodimer in complex with a 7-sugar oligosaccharide acceptor analog

EMDB-26701: 
Cryo-EM structure of the human Exostosin-1 and Exostosin-2 heterodimer in complex with UDP-GlcNAc

EMDB-26702: 
Cryo-EM structure of the human Exostosin-1 and Exostosin-2 heterodimer in complex with UDP-GlcA

PDB-7sch: 
Cryo-EM structure of the human Exostosin-1 and Exostosin-2 heterodimer

PDB-7scj: 
Cryo-EM structure of the human Exostosin-1 and Exostosin-2 heterodimer in complex with a 4-sugar oligosaccharide acceptor analog

PDB-7sck: 
Cryo-EM structure of the human Exostosin-1 and Exostosin-2 heterodimer in complex with a 7-sugar oligosaccharide acceptor analog

PDB-7uqx: 
Cryo-EM structure of the human Exostosin-1 and Exostosin-2 heterodimer in complex with UDP-GlcNAc

PDB-7uqy: 
Cryo-EM structure of the human Exostosin-1 and Exostosin-2 heterodimer in complex with UDP-GlcA
EMDB-26396: 
CryoET of E. coli prepared with the Waffle Method

EMDB-23766: 
Insulin receptor ectodomain dimer complexed with two IRPA-3 partial agonists

EMDB-23767: 
Insulin receptor ectodomain dimer complexed with two IRPA-9 partial agonists

PDB-7md4: 
Insulin receptor ectodomain dimer complexed with two IRPA-3 partial agonists

PDB-7md5: 
Insulin receptor ectodomain dimer complexed with two IRPA-9 partial agonists

EMDB-24181: 
The structure of bovine thyroglobulin with iodinated tyrosines

PDB-7n4y: 
The structure of bovine thyroglobulin with iodinated tyrosines

EMDB-23122: 
Subtomogram average of the hexameric fungal plasma membrane proton pump Pma1 from protoplast membrane of Candida glabrata CBS138 strain

EMDB-23123: 
Subtomogram average of the hexameric fungal plasma membrane proton pump Pma1 from protoplast membrane of Candida glabrata KH238 strain

EMDB-22052: 
Apoferritin short exposure 3D reconstruction with 10% total dose

EMDB-22053: 
Apoferritin short exposure 3D reconstruction with 25% total dose

EMDB-22056: 
Apoferritin short exposure 3D reconstruction with 50% total dose

EMDB-22057: 
Apoferritin short exposure 3D reconstruction with 75% total dose

EMDB-22058: 
Apoferritin short exposure 3D reconstruction with 100% total dose

EMDB-22059: 
Clustered protocadherin partially open conformation

EMDB-22060: 
Clustered protocadherin closed conformation

EMDB-4772: 
Cryo-EM structure of autoinhibited human talin-1

PDB-6r9t: 
Cryo-EM structure of autoinhibited human talin-1

EMDB-20521: 
Horse spleen apoferritin light chain

PDB-6pxm: 
Horse spleen apoferritin light chain

EMDB-9233: 
T20S proteasome

EMDB-9203: 
bovine glutamate dehydrogenase (18apr21a)

EMDB-8156: 
Autoinhibited F-BAR protein Nervous Wreck (non-membrane-bound)

EMDB-8161: 
Nervous wreck (Nwk aa 1-731) on lipid monolayer

EMDB-2715: 
Electron cryo-microscopy of ABCG2 from two-dimensional crystals

PDB-4v8y: 
Cryo-EM reconstruction of the 80S-eIF5B-Met-itRNAMet Eukaryotic Translation Initiation Complex

PDB-4v8z: 
Cryo-EM reconstruction of the 80S-eIF5B-Met-itRNAMet Eukaryotic Translation Initiation Complex

PDB-4v68: 
T. thermophilus 70S ribosome in complex with mRNA, tRNAs and EF-Tu.GDP.kirromycin ternary complex, fitted to a 6.4 A Cryo-EM map.

EMDB-2421: 
Molecular architecture of the 80S-eIF5B-Met-itRNAMet Eukaryotic Translation Initiation Complex
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
