-検索条件
-検索結果
検索 (著者・登録者: howe & a)の結果203件中、1から50件目までを表示しています
EMDB-40601: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa
EMDB-40625: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa
EMDB-40626: 
cytochrome bc1 complex from Pseudomonas aeruginosa

EMDB-40627: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (Locally refined cytochrome cbb3)
EMDB-40637: 
cytochrome bc1 complex from Pseudomonas aeruginosa (Locally refined bc1 monomer with Rieske head domain in b state)
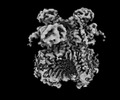
EMDB-40638: 
cytochrome bc1 complex from Pseudomonas aeruginosa (Locally refined bc1 monomer with Rieske head domain in c state)

EMDB-40643: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (Locally refined cytochrome cbb3 with CcoP1 isoform)
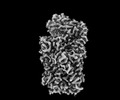
EMDB-40645: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (Locally refined cytochrome cbb3 with CcoP2 isoform)

PDB-8smr: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa

PDB-8snh: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa

EMDB-17265: 
ATM(Q2971A) activated by oxidative stress in complex with Mg AMP-PNP and p53 peptide

EMDB-17266: 
ATM(Q2971A) dimeric C-terminal region activated by oxidative stress in complex with Mg AMP-PNP and p53 peptide

EMDB-17267: 
ATM(Q2971A) in complex with Mg AMP-PNP

EMDB-17268: 
ATM(Q2971A) dimeric C-terminal region in complex with Mg AMP-PNP

PDB-8oxm: 
ATM(Q2971A) activated by oxidative stress in complex with Mg AMP-PNP and p53 peptide

PDB-8oxo: 
ATM(Q2971A) dimeric C-terminal region activated by oxidative stress in complex with Mg AMP-PNP and p53 peptide

PDB-8oxp: 
ATM(Q2971A) in complex with Mg AMP-PNP

PDB-8oxq: 
ATM(Q2971A) dimeric C-terminal region in complex with Mg AMP-PNP

EMDB-40983: 
Negative stain EM assembly of MYC, JAZ, and NINJA complex

PDB-8t2i: 
Negative stain EM assembly of MYC, JAZ, and NINJA complex

EMDB-29365: 
Co-structure of the Respiratory Syncytial Virus RNA-dependent RNA polymerase with MRK-1

EMDB-29366: 
Co-structure of the Human Metapneunomovirus RNA-dependent RNA polymerase with MRK-1

PDB-8fpi: 
Co-structure of the Respiratory Syncytial Virus RNA-dependent RNA polymerase with MRK-1

PDB-8fpj: 
Co-structure of the Human Metapneunomovirus RNA-dependent RNA polymerase with MRK-1

EMDB-16772: 
Subtomogram average of Immature Rotavirus TLP penton

EMDB-16773: 
In situ STA of rotavirus TLP (icos)

PDB-8co6: 
Subtomogram average of Immature Rotavirus TLP penton

EMDB-16762: 
In situ map of Rotavirus SLP
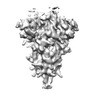
EMDB-16403: 
Subtomogram averaging of a coronavirus spike (ChAdOx1 19E6) in C3 symmetry
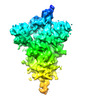
EMDB-16404: 
Subtomogram averaging of a coronavirus spike (ChAdOx1 19E6) in C1 symmetry

EMDB-16405: 
Subtomogram averaging of a coronavirus spike (ChAdOx1 19E) in C3 symmetry

EMDB-16406: 
Subtomogram averaging of a coronavirus spike in situ (ChAdOx1 19E) in C1 symmetry

EMDB-16697: 
Subtomogram averaging of a coronavirus spike (ChAdOx1 19E6) in C1 symmetry after 3D classification

EMDB-16146: 
SPA of Trypsin untreated Rotavirus TLP spike

EMDB-16769: 
In situ STA of rotavirus DLP (penton)

EMDB-16771: 
In situ STA of Rotavirus enveloped DLP (icos)

EMDB-16774: 
in situ Subtomogram average of Immature Rotavirus TLP spike

PDB-8bp8: 
SPA of Trypsin untreated Rotavirus TLP spike

PDB-8coa: 
in situ Subtomogram average of Immature Rotavirus TLP spike

EMDB-16767: 
In situ STA of rotavirus enveloped DLP (penton)

EMDB-16511: 
HERV-K Gag immature lattice

PDB-8c9m: 
HERV-K Gag immature lattice

EMDB-15870: 
Subtomogram average of the human Sec61-TRAP-OSTA-translocon

EMDB-15871: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Decoding-Sampling State

EMDB-15872: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Classical Pre+ State

EMDB-15873: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Classical Pre State

EMDB-15874: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Rotated-1 Pre+ State
EMDB-15875: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Rotated-1 Pre State
EMDB-15876: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Rotated-2 Pre State

EMDB-15877: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Translocation State
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
