-検索条件
-検索結果
検索 (著者・登録者: christine & b)の結果407件中、1から50件目までを表示しています

EMDB-43017: 
60S ribosome biogenesis intermediate (Dbp10 pre-catalytic structure - Overall map)

EMDB-43018: 
60S ribosome biogenesis intermediate (Dbp10 pre-catalytic structure - PTC Local map)

EMDB-43019: 
60S ribosome biogenesis intermediate (Dbp10 pre-catalytic structure - Local map L1 region)

EMDB-43020: 
60S ribosome biogenesis intermediate (Dbp10 pre-catalytic structure - Local map Rrp14/Rrp15/Ssf1 region)

EMDB-43021: 
60S ribosome biogenesis intermediate (Dbp10 catalytic structure - Overall map)

EMDB-43022: 
60S ribosome biogenesis intermediate (Dbp10 catalytic structure - Dbp10 Local map)

EMDB-43023: 
60S ribosome biogenesis intermediate (Dbp10 catalytic structure - Low-pass filtered locally refined map)

EMDB-43024: 
60S ribosome biogenesis intermediate (Dbp10 catalytic structure - L1 local map

EMDB-43026: 
60S ribosome biogenesis intermediate (Dbp10 catalytic intermediate - Rrp14/Rrp15/Ssf1 local map)

EMDB-43027: 
60S ribosome biogenesis intermediate (Dbp10 post-catalytic structure - Overall map)

EMDB-43028: 
60S ribosome biogenesis intermediate (Dbp10 post-catalytic structure - Dbp10 Local map)

EMDB-43029: 
60S ribosome biogenesis intermediate (Dbp10 post-catalytic structure - H64 Local map)

PDB-8v83: 
60S ribosome biogenesis intermediate (Dbp10 pre-catalytic structure - Overall map)

PDB-8v84: 
60S ribosome biogenesis intermediate (Dbp10 catalytic structure - Overall map)

PDB-8v85: 
60S ribosome biogenesis intermediate (Dbp10 catalytic structure - Low-pass filtered locally refined map)

PDB-8v87: 
60S ribosome biogenesis intermediate (Dbp10 post-catalytic structure - Overall map)

EMDB-19405: 
Tomograms of spoIVB Bacillus subtilis sporangia
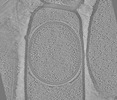
EMDB-19411: 
Tomograms of cotE Bacillus subtilis sporangia

EMDB-18245: 
Plunge-frozen (control) map of beta-galactosidase

EMDB-18244: 
ESIBD structure of beta-galactosidase

PDB-8q7y: 
ESIBD structure of beta-galactosidase

EMDB-17705: 
Structure of Chelator-GIDSR4 - Fbp1 - phospho-Ubc8~ubiquitin - class I

EMDB-17706: 
Structure of Chelator-GIDSR4 - Fbp1 - phospho-Ubc8~ubiquitin - class II

EMDB-17707: 
Chelator-GIDSR4 - Fbp1 - phospho-Ubc8~ubiquitin - class III

EMDB-17709: 
Structure of Chelator-GIDSR4 - Fbp1 - phospho-Ubc8~ubiquitin - class V

EMDB-17710: 
Structure of Chelator-GIDSR4 - Fbp1 - phospho-Ubc8~ubiquitin - class IV

EMDB-17713: 
Catalytic module of human CTLH E3 ligase bound to multiphosphorylated UBE2H~ubiquitin

EMDB-17715: 
SRS and Cat modules of human CTLHSR4 bound to multiphosphorylated UBE2H~ubiquitin

EMDB-17716: 
Structure of CTLHSR4 - phospho-UBE2H~ubiquitin bound to engineered VH
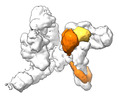
EMDB-17717: 
SRS and Cat modules of yeast Chelator-GIDSR4 bound to multiphosphorylated Ubc8~ubiquitin
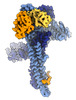
EMDB-17764: 
Catalytic module of yeast GID E3 ligase bound to multiphosphorylated Ubc8~ubiquitin

PDB-8pjn: 
Catalytic module of human CTLH E3 ligase bound to multiphosphorylated UBE2H~ubiquitin

PDB-8pmq: 
Catalytic module of yeast GID E3 ligase bound to multiphosphorylated Ubc8~ubiquitin

EMDB-18323: 
Chimeric Adenovirus-derived dodecamer

PDB-8qbx: 
Chimeric Adenovirus-derived dodecamer

EMDB-41370: 
Structure of a class A GPCR/Fab complex

EMDB-41827: 
Structure of a class A GPCR/agonist complex (focused map2)

EMDB-41828: 
Structure of a class A GPCR/agonist complex (focused map1)

EMDB-41829: 
Structure of a class A GPCR/agonist complex

EMDB-41850: 
Structure of a class A GPCR/agonist complex (Consensus map)

PDB-8tlm: 
Structure of a class A GPCR/Fab complex

PDB-8u1u: 
Structure of a class A GPCR/agonist complex

EMDB-42399: 
Cryo-EM structure of T4 Bacteriophage Clamp Loader with Sliding Clamp and DNA

EMDB-42402: 
Cryo-EM structure of T4 Bacteriophage Clamp Loader with Sliding Clamp

PDB-8unf: 
Cryo-EM structure of T4 Bacteriophage Clamp Loader with Sliding Clamp and DNA

PDB-8unh: 
Cryo-EM structure of T4 Bacteriophage Clamp Loader with Sliding Clamp

EMDB-16242: 
Cryo-EM structure of RANBP10-CTLH SR4 complex

EMDB-16243: 
Cryo-EM map of ARMC8-specific nanobody bound to CTLH-SR4

EMDB-29281: 
Cryo-EM structure of STING oligomer bound to cGAMP and NVS-STG2

EMDB-29282: 
Cryo-EM structure of STING oligomer bound to cGAMP, NVS-STG2 and C53
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
