-検索条件
-検索結果
検索 (著者・登録者: castro & h)の結果308件中、1から50件目までを表示しています

EMDB-18180: 
cryoEM structure of SARS-CoV2 Spike trimer in complex with Fab23

PDB-8q5y: 
cryoEM structure of SARS-CoV2 Spike trimer in complex with Fab23

EMDB-16375: 
SARS-CoV2 Omicron BA.1 RBD in complex with CAB-A17 antibody

PDB-8c0y: 
SARS-CoV2 Omicron BA.1 RBD in complex with CAB-A17 antibody

EMDB-42970: 
Model and map from local refinement of a CAB-A17 - Omicron Ba.1 spike complex

PDB-8v4f: 
Model and map from local refinement of a CAB-A17 - Omicron Ba.1 spike complex

EMDB-17125: 
Knockout of GMC-oxidoreductase genes reveals that functional redundancy preserves mimivirus essential functions

EMDB-17131: 
Knockout of GMC-oxidoreductase genes reveals that functional redundancy preserves mimivirus essential functions

PDB-8orh: 
Knockout of GMC-oxidoreductase genes reveals that functional redundancy preserves mimivirus essential functions

PDB-8ors: 
Knockout of GMC-oxidoreductase genes reveals that functional redundancy preserves mimivirus essential functions

EMDB-15270: 
SARS Cov2 Spike RBD in complex with Fab47

PDB-8a95: 
SARS Cov2 Spike RBD in complex with Fab47

EMDB-27500: 
EBNA1 DNA binding domain (DBD) (458-617)+2 repeats of family repeat (FR) region

PDB-8dlf: 
EBNA1 DNA binding domain (DBD) (458-617)+2 repeats of family repeat (FR) region

EMDB-15273: 
SARS Cov2 Spike in 1-up conformation complex with Fab47

PDB-8a99: 
SARS Cov2 Spike in 1-up conformation complex with Fab47

EMDB-15269: 
SARS CoV2 Spike in the 2-up state in complex with Fab47

EMDB-15271: 
SARS Cov2 Spike RBD in complex with Fab47

PDB-8a94: 
SARS CoV2 Spike in the 2-up state in complex with Fab47.

PDB-8a96: 
SARS Cov2 Spike RBD in complex with Fab47

EMDB-26873: 
Subtomogram averages of the 8-nm repeats of the conoid fibers from the cryo-FIB milled Neospora caninum

EMDB-28231: 
Subtomogram averages of the pre-conoid ring P1 and P2 from the cryo-FIB milled Neospora caninum

EMDB-28234: 
Subtomogram averages of the pre-conoid ring P3 from the cryo-FIB milled Neospora caninum

EMDB-28246: 
Composite map of the pre-conoid ring P1, P2 and P3 from the cryo-FIB milled Neospora caninum

EMDB-28247: 
Subtomogram averages of the 8-nm repeats of the conoid fibers in the protruded states from the cryo-FIB milled Neospora caninum

EMDB-28249: 
Subtomogram averages of the 8-nm repeats of the conoid fibers in the retracted states from the cryo-FIB milled Neospora caninum

EMDB-26610: 
Cryo-EM structure of rabbit RyR1 in the presence of high Mg2+ and AMP-PCP in nanodisc
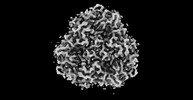
EMDB-32588: 
The Cryo-EM structure of siphonaxanthin chlorophyll a/b type light-harvesting complex II

PDB-7wlm: 
The Cryo-EM structure of siphonaxanthin chlorophyll a/b type light-harvesting complex II

EMDB-14633: 
PucA-LH2 complex from Rps. palustris

PDB-7zcu: 
PucA-LH2 complex from Rps. palustris

EMDB-14650: 
PucB-LH2 complex from Rps. palustris

EMDB-14682: 
PucD-LH2 complex from Rps. palustris

EMDB-14685: 
PucE-LH2 complex from Rps. palustris

PDB-7zdi: 
PucB-LH2 complex from Rps. palustris

PDB-7ze3: 
PucD-LH2 complex from Rps. palustris

PDB-7ze8: 
PucE-LH2 complex from Rps. palustris

EMDB-24762: 
in situ cryo-electron tomogram of wild-type yeast lipid droplet in 2% glucose-fed log growth conditions

EMDB-24764: 
in situ cryo-electron tomogram of wild-type yeast lipid droplet after 4-hour exposure to acute glucose restriction (AGR, 0.001% glucose media)

EMDB-24766: 
in situ cryo-electron tomogram of wild-type yeast lipid droplet after 4-hour exposure to acute glucose restriction (AGR, 0.001% glucose media) with 0.1% oleic acid

EMDB-24767: 
in situ cryo-electron tomogram of tgl3,4,5-ko yeast lipid droplet after 4-hour exposure to acute glucose restriction (AGR, 0.001% glucose media)

EMDB-24781: 
in situ cryo-electron tomogram of wild-type yeast lipid droplet in 2% glucose-fed log growth conditions with 0.1% oleic acid

EMDB-24782: 
in situ cryo-electron tomogram of nvj1-ko yeast lipid droplet after 4-hour exposure to acute glucose restriction (AGR, 0.001% glucose media)

EMDB-13421: 
Cryo-EM maps of the RET/GDF15/GFRAL complex

EMDB-13688: 
Negative stain EM map of the extracellular domain of the RET(C634R) mutant dimer
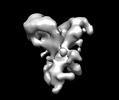
EMDB-13689: 
Negative stain EM map of the extracellular domain of the RET(C634R)/GDF15/GFRAL complex

EMDB-24210: 
The 3D structure and in situ arrangements of CatSper channel from the cryo-electron tomography and subtomographic average of mouse wild type sperm flagella

EMDB-26206: 
The 3D structure and in situ arrangements (Forward slash) of CatSper channel from the cryo-electron tomography and subtomographic average of mouse efcab9 mutant sperm flagella

EMDB-26207: 
The 3D structure and in situ arrangements (Backslash) of CatSper channel from the cryo-electron tomography and subtomographic average of mouse efcab9 mutant sperm flagella

EMDB-13416: 
Human voltage-gated potassium channel Kv3.1 (apo condition)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
