-検索条件
-検索結果
検索 (著者・登録者: ludtke & s)の結果177件中、1から50件目までを表示しています

EMDB-29696: 
In situ structure of GspD-beta-GspS complex on the bacterial outer membrane

EMDB-29697: 
In situ structure of GspD-beta-GspS complex on the bacterial outer membrane when GspD-beta is overexpressed solely

EMDB-29698: 
In situ structure of GspD-beta on the bacterial inner membrane

EMDB-29702: 
In situ structure of GspD-alpha on the bacterial inner membrane

EMDB-29703: 
In situ structure of GspD-alpha on the bacterial outer membrane
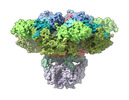
EMDB-27982: 
Structure of the full-length IP3R1 channel determined at high Ca2+
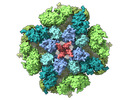
EMDB-27983: 
Structure of the full-length IP3R1 channel determined in the presence of Calcium/IP3/ATP

PDB-8eaq: 
Structure of the full-length IP3R1 channel determined at high Ca2+

PDB-8ear: 
Structure of the full-length IP3R1 channel determined in the presence of Calcium/IP3/ATP

EMDB-24609: 
Subnanometer in situ structure of the AcrAB-TolC efflux pump bound with MBX

EMDB-26018: 
The symmetry-released subpellicular microtubule map from detergent-extracted Toxoplasma cells

EMDB-26019: 
Subpellicular microtubule from detergent-extract Toxoplasma gondii cells

EMDB-26020: 
The tubulin-based conoid from detergent-extract Toxoplasma gondii cells

PDB-7tnq: 
The symmetry-released subpellicular microtubule map from detergent-extracted Toxoplasma cells

PDB-7tns: 
Subpellicular microtubule from detergent-extract Toxoplasma gondii cells

PDB-7tnt: 
The tubulin-based conoid from detergent-extract Toxoplasma gondii cells

EMDB-26010: 
The symmetrized subpellicular microtubule map from intact Toxoplasma gondii cells

EMDB-26006: 
3D reconstruction of detergent-extract Toxoplasma gondii cells at the apical end

EMDB-26007: 
Three-dimensional organization of the apical complex in Toxoplasma tachyzoites

EMDB-26008: 
In situ average map of conoid with PCR from intact Toxoplasma gondii cells

EMDB-26009: 
The conoid segment from intact Toxoplasma gondii cells

EMDB-24287: 
Cryo-ET platelet of WT mouse

EMDB-24288: 
Cryo-ET platelet of AML mouse

EMDB-24289: 
Cryo-ET platelet of pre-AML mouse

EMDB-24291: 
Cryo-ET platelet of control mouse (1 week)

EMDB-24298: 
Cryo-ET platelet of control mouse (3 week)

EMDB-24224: 
Combined 3D structure of the isolated yeast NPC

EMDB-24225: 
Low resolution, multi-part 3D structure of the isolated yeast NPC

EMDB-24231: 
Double nuclear outer ring from the isolated yeast NPC

EMDB-24232: 
Inner ring spoke from the isolated yeast NPC

EMDB-24258: 
Structure of the in situ yeast NPC
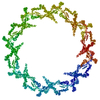
PDB-7n84: 
Double nuclear outer ring from the isolated yeast NPC
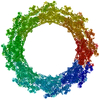
PDB-7n85: 
Inner ring spoke from the isolated yeast NPC

PDB-7n9f: 
Structure of the in situ yeast NPC

EMDB-24129: 
Classification of ribosome assembly intermediates

EMDB-24130: 
Exploration of subtle structural variability of ribosome assembly intermediates

EMDB-24131: 
Continuous movement of the central protuberance domain of 50S ribosome.

EMDB-24092: 
Continuous movement of the pre-catalytic spliceosome, mode 1.

EMDB-24093: 
Continuous movement of the pre-catalytic spliceosome, mode 2.

EMDB-24094: 
Continuous movement of the pre-catalytic spliceosome, mode 1-2.

EMDB-24096: 
Continuous movement of the pre-catalytic spliceosome, mode 1+2.

EMDB-24118: 
Continuous motion of SARS-COV-2 spike protein receptor binding domain. Movement model 1.

EMDB-24119: 
Continuous motion of SARS-COV-2 spike protein receptor binding domain. Movement model 2.

EMDB-23122: 
Subtomogram average of the hexameric fungal plasma membrane proton pump Pma1 from protoplast membrane of Candida glabrata CBS138 strain

EMDB-23123: 
Subtomogram average of the hexameric fungal plasma membrane proton pump Pma1 from protoplast membrane of Candida glabrata KH238 strain

EMDB-22079: 
EM Structure of Full-Length Androgen Receptor and DNA Complex

EMDB-22080: 
EM Structure of Full-Length Androgen Receptor Coactivator Complex

EMDB-20416: 
Cryo-EM structure of the full-length Bacillus subtilis glyQS T-box riboswitch in complex with tRNA-Gly
PDB-6pom: 
Cryo-EM structure of the full-length Bacillus subtilis glyQS T-box riboswitch in complex with tRNA-Gly

EMDB-0529: 
Purified ribosome - A complete data processing workflow for CryoET and subtomogram averaging
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
