-検索条件
-検索結果
検索 (著者・登録者: yan & jh)の結果170件中、1から50件目までを表示しています
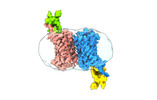
EMDB-36488: 
Structure of Duffy Antigen Receptor for Chemokines (DARC)/ACKR1 in complex with the chemokine, CCL7 (Composite map)

EMDB-37212: 
Structure of Duffy Antigen Receptor for Chemokines (DARC)/ACKR1 in complex with the chemokine, CCL7 (Receptor original map)
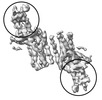
EMDB-37214: 
Structure of Duffy Antigen Receptor for Chemokines (DARC)/ACKR1 in complex with the chemokine, CCL7 (Ligand/CCL7 focused map)

PDB-8jps: 
Structure of Duffy Antigen Receptor for Chemokines (DARC)/ACKR1 in complex with the chemokine, CCL7 (Composite map)

EMDB-19929: 
Structural basis of D9-THC analog activity at the Cannabinoid 1 receptor

PDB-9erx: 
Structural basis of D9-THC analog activity at the Cannabinoid 1 receptor

EMDB-38216: 
Cryo-EM structure of SARS-CoV-2 S-BQ.1 in complex with antibody O5C2
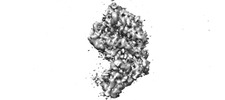
EMDB-36892: 
Structure of BtKY72 spike receptor-binding domain (RBD) complexed with bat ACE2

EMDB-35827: 
Structure of CbCas9 bound to 20-nucleotide complementary DNA substrate

EMDB-37652: 
Structure of CbCas9 bound to 6-nucleotide complementary DNA substrate

EMDB-37656: 
Structure of CbCas9-PcrIIC1 complex bound to 28-bp DNA substrate (20-nt complementary)

EMDB-37657: 
Structure of CbCas9-PcrIIC1 complex bound to 62-bp DNA substrate (symmetric 20-nt complementary)

EMDB-37762: 
Structure of CbCas9-PcrIIC1 complex bound to 62-bp DNA substrate (non-targeting complex)

EMDB-40825: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10
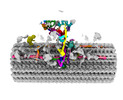
EMDB-41284: 
Combined linker domain of N-DRC and associated proteins Tetrahymena

PDB-8tid: 
Combined linker domain of N-DRC and associated proteins Tetrahymena

EMDB-41189: 
Baseplate of Nexin-dynein regulatory complex from Tetrahymena thermophila
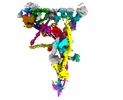
EMDB-41251: 
Linker domain of Nexin-dynein regulatory complex from Tetrahymena thermophila

EMDB-41270: 
Focused refinement of the N-DRC from the Tetrahymena WT subtomo

EMDB-41375: 
Linker domain of N-DRC complex from Tetrahymena thermophila

EMDB-41376: 
96nm repeat of Doublet microtubule from Tetrahymena thermophila

EMDB-41504: 
DRC9/10 baseplate from Tetrahymena thermophila

PDB-8tek: 
Baseplate of Nexin-dynein regulatory complex from Tetrahymena thermophila

PDB-8th8: 
Linker domain of Nexin-dynein regulatory complex from Tetrahymena thermophila

EMDB-28692: 
30S_delta_ksgA_h44_inactive_conformation
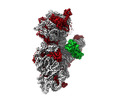
EMDB-28720: 
30S_delta_ksgA+KsgA complex

EMDB-35670: 
Structure of Full-Length AsfvPrimPol in Apo-Form

EMDB-35671: 
Structure of Full-Length AsfvPrimPol in Complex-Form

EMDB-33734: 
Cryo-EM structure of SARS-CoV-2 spike in complex with K202.B bispecific antibody

EMDB-28178: 
Structure of lineage IV Lassa virus glycoprotein complex (strain Josiah)

EMDB-28179: 
Structure of lineage II Lassa virus glycoprotein complex (strain NIG08-A41)

EMDB-28180: 
Structure of lineage V Lassa virus glycoprotein complex (strain Soromba-R)

EMDB-28181: 
Structure of lineage VII Lassa virus glycoprotein complex (strain Togo/2016/7082)

EMDB-28182: 
Lassa virus glycoprotein complex (Josiah) bound to 12.1F Fab

EMDB-28183: 
Lassa virus glycoprotein complex (Josiah) bound to 19.7E Fab

EMDB-28184: 
Lassa virus glycoprotein complex (Josiah) bound to S370.7 Fab

EMDB-28254: 
Composite 70S ribosome structure for "Atomistic simulations of the E. coli ribosome provide selection criteria for translationally active substrates

EMDB-28255: 
70S map for: "Atomistic simulations of the E. coli ribosome provide selection criteria for translationally active substrates"

EMDB-28256: 
30S-focused map for: "Atomistic simulations of the E. coli ribosome provide selection criteria for translationally active substrates"

EMDB-28257: 
50S-focused map for: "Atomistic simulations of the E. coli ribosome provide selection criteria for translationally active substrates"

EMDB-29397: 
Structure of Mycobacterium smegmatis Rsh bound to a 70S translation initiation complex

PDB-8fr8: 
Structure of Mycobacterium smegmatis Rsh bound to a 70S translation initiation complex

EMDB-27973: 
Erwinia amylovora 70S Ribosome

EMDB-27993: 
Erwinia amlyavora 50S ribosome

EMDB-28003: 
Erwinia phage vB_EamM_RAY (RAY) Capsid Vertex

EMDB-28004: 
Erwinia phage vB_EamM_RAY (RAY) Capsid Collar

EMDB-28005: 
Erwinia phage vB_EamM_RAY (RAY) Tail Sheath

EMDB-28006: 
Erwinia phage vB_EamM_RAY (RAY) Baseplate

EMDB-28007: 
Erwinia phage vB_EamM_RAY (RAY) Chimallin

EMDB-28008: 
Erwinia phage vB_EamM_RAY (RAY) Putative PhuZ Filament
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
