-検索条件
-検索結果
検索 (著者・登録者: qu & c)の結果3,784件中、1から50件目までを表示しています

EMDB-17164: 
Structure of human terminal uridylyltransferase 4 (TUT4, ZCCHC11) in complex with pre-let7g miRNA and Lin28A

EMDB-50860: 
Cryo EM map of the type 2A polymorph of alpha-synuclein at pH 7.0.

EMDB-50888: 
Cryo EM structure of the type 3B polymorph of alpha-synuclein at low pH.

EMDB-38580: 
Structure of human class T GPCR TAS2R14-miniGs/gust complex with Aristolochic acid A.

EMDB-38582: 
Structure of human class T GPCR TAS2R14-DNGi complex with Aristolochic acid A.

EMDB-38583: 
Structure of human class T GPCR TAS2R14-Gi complex with Aristolochic acid A.

EMDB-38584: 
Structure of human class T GPCR TAS2R14-Gustducin complex with Aristolochic acid A.

EMDB-38586: 
Structure 2 of human class T GPCR TAS2R14-miniGs/gust complex with Flufenamic acid.

EMDB-38587: 
Structure of human class T GPCR TAS2R14-DNGi complex with Flufenamic acid.

EMDB-38588: 
Structure of human class T GPCR TAS2R14-Gi complex.

EMDB-39376: 
Structure of human class T GPCR TAS2R14-Ggustducin complex with agonist 28.1

PDB-8xql: 
Structure of human class T GPCR TAS2R14-miniGs/gust complex with Aristolochic acid A.

PDB-8xqn: 
Structure of human class T GPCR TAS2R14-DNGi complex with Aristolochic acid A.

PDB-8xqo: 
Structure of human class T GPCR TAS2R14-Gi complex with Aristolochic acid A.

PDB-8xqp: 
Structure of human class T GPCR TAS2R14-Gustducin complex with Aristolochic acid A.

PDB-8xqr: 
Structure 2 of human class T GPCR TAS2R14-miniGs/gust complex with Flufenamic acid.

PDB-8xqs: 
Structure of human class T GPCR TAS2R14-DNGi complex with Flufenamic acid.

PDB-8xqt: 
Structure of human class T GPCR TAS2R14-Gi complex.

PDB-8yky: 
Structure of human class T GPCR TAS2R14-Ggustducin complex with agonist 28.1

EMDB-43097: 
Simulation-driven design of prefusion stabilized SARS-CoV-2 spike S2 antigen

EMDB-50815: 
Rea1 delta AAA2H2alpha ATPgS structure

EMDB-50816: 
Rea1 D2915A-R2976A-D3042A mutant ATP conformation I

EMDB-50817: 
Rea1 D2915A-R2976A-D3042A ATP conformation II

EMDB-50818: 
Rea1 D2915A-R2976A-D3042A mutant ATP conformation III

EMDB-50296: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

PDB-9fbv: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

EMDB-37210: 
Prefusion RSV F Bound to Lonafarnib and D25 Fab

PDB-8kg5: 
Prefusion RSV F Bound to Lonafarnib and D25 Fab

EMDB-44839: 
Intact V-ATPase State 2 and synaptophysin complex in mouse brain isolated synaptic vesicles

EMDB-44840: 
Intact V-ATPase State 3 and synaptophysin complex in mouse brain isolated synaptic vesicles
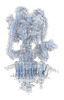
EMDB-44841: 
Intact V-ATPase State 3 in synaptophysin knock-out isolated synaptic vesicles

EMDB-44842: 
Intact V-ATPase State 2 in synaptophysin knock-out isolated synaptic vesicles

EMDB-44843: 
Intact V-ATPase State 1 and synaptophysin complex in mouse brain isolated synaptic vesicles

EMDB-44844: 
Intact V-ATPase State 1 in synaptophysin knock-out isolated synaptic vesicles
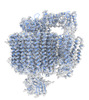
EMDB-44845: 
V0-only V-ATPase in synaptophysin gene knock-out mouse brain isolated synaptic vesicles

EMDB-44846: 
V0-only V-ATPase and synaptophysin complex in mouse brain isolated synaptic vesicles

EMDB-44847: 
Tomograms of isolated synaptic vesicles from Syp-/- mouse brain

EMDB-44848: 
Tomograms of isolated synaptic vesicles from wild-type mouse brain

EMDB-44855: 
Intact state1 V-ATPase of isolated synaptic vesicles from wild-type mouse brain by subtomogram averaging

EMDB-44856: 
Intact state2 V-ATPase of isolated synaptic vesicles from wild-type mouse brain by subtomogram averaging

EMDB-44857: 
Intact state3 V-ATPase of isolated synaptic vesicles from wild-type mouse brain by subtomogram averaging

EMDB-44858: 
V0-only V-ATPase of isolated synaptic vesicles from wild-type mouse brain by subtomogram averaging

PDB-9bra: 
Intact V-ATPase State 2 and synaptophysin complex in mouse brain isolated synaptic vesicles

PDB-9brq: 
Intact V-ATPase State 3 and synaptophysin complex in mouse brain isolated synaptic vesicles

PDB-9brr: 
Intact V-ATPase State 3 in synaptophysin knock-out isolated synaptic vesicles

PDB-9brs: 
Intact V-ATPase State 2 in synaptophysin knock-out isolated synaptic vesicles

PDB-9brt: 
Intact V-ATPase State 1 and synaptophysin complex in mouse brain isolated synaptic vesicles

PDB-9bru: 
Intact V-ATPase State 1 in synaptophysin knock-out isolated synaptic vesicles

PDB-9bry: 
V0-only V-ATPase in synaptophysin gene knock-out mouse brain isolated synaptic vesicles

PDB-9brz: 
V0-only V-ATPase and synaptophysin complex in mouse brain isolated synaptic vesicles
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
