-検索条件
-検索結果
検索 (著者・登録者: peters & n)の結果339件中、1から50件目までを表示しています

EMDB-41839: 
Cryo-EM structure of yeast SWR1C subunit Swc5 bound to the nucleosome, 3D class 0

EMDB-41851: 
Cryo-EM structure of yeast SWR1C subunit Swc5 bound to the nucleosome, 3D class 1

EMDB-41852: 
Cryo-EM structure of yeast SWR1C subunit Swc5 bound to the nucleosome, 3D class 2

EMDB-41853: 
Cryo-EM structure of yeast SWR1C subunit Swc5 bound to the nucleosome, 3D class 4

EMDB-18594: 
Cryo-EM structure of E. coli cytochrome bo3 quinol oxidase assembled in peptidiscs

PDB-8qqk: 
Cryo-EM structure of E. coli cytochrome bo3 quinol oxidase assembled in peptidiscs

EMDB-43736: 
Umb1 umbrella toxin particle

EMDB-43737: 
Umb1 umbrella toxin particle (local refinement of UmbB1 bound ALF of UmbC1 and UmbA1)

PDB-8w20: 
Umb1 umbrella toxin particle

PDB-8w22: 
Umb1 umbrella toxin particle (local refinement of UmbB1 bound ALF of UmbC1 and UmbA1)
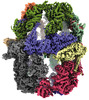
EMDB-19477: 
Saccharomyces cerevisiae FAS type I

EMDB-19489: 
Tobacco mosaic virus from scanning transmission electron microscopy at CSA=2.0 mrad

EMDB-16004: 
Structure of hexameric subcomplexes (Truncation Delta2-6) of the fractal citrate synthase from Synechococcus elongatus PCC7942
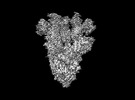
EMDB-28198: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike in complex with LLNL-199

EMDB-28199: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike in complex with 2130-1-0114-112

PDB-8ekd: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike in complex with 2130-1-0114-112

EMDB-15529: 
Structure of a first level Sierpinski triangle formed by a citrate synthase

EMDB-16510: 
AQP7_inhibitor

PDB-8c9h: 
AQP7_inhibitor

EMDB-43137: 
SARS-CoV-2 Frameshift Stimulatory Element with Upstream Multibranch Loop

PDB-8vci: 
SARS-CoV-2 Frameshift Stimulatory Element with Upstream Multibranch Loop

EMDB-41579: 
Structure of full-length LexA bound to a RecA filament

PDB-8trg: 
Structure of full-length LexA bound to a RecA filament

EMDB-27813: 
Structure of monomeric LRRK1

EMDB-27814: 
Local refinement around RCKW of LRRK1

EMDB-27815: 
Local refinement of LRRK1 around the ROC-COR-kinase domains

EMDB-27816: 
Local refinement around kinase and WD40 domains of LRRK1

EMDB-27817: 
Structure of dimeric LRRK1

EMDB-27818: 
Symmetry expansion of dimeric LRRK1

PDB-8e04: 
Structure of monomeric LRRK1

PDB-8e05: 
Structure of dimeric LRRK1

PDB-8e06: 
Symmetry expansion of dimeric LRRK1

EMDB-27810: 
Cryo-EM structure of chi dynein bound to Lis1

EMDB-27811: 
Symmetry expansion of yeast cytoplasmic dynein-1 bound to Lis1 in the chi conformation.

PDB-8dzz: 
Cryo-EM structure of chi dynein bound to Lis1

PDB-8e00: 
Symmetry expansion of yeast cytoplasmic dynein-1 bound to Lis1 in the chi conformation.

EMDB-18010: 
Charging of vitreous samples in cryogenic electron microscopy mitigated by graphene - BfrB - Dataset 4 - Quantifoil 300 mesh R1.2/1.3 with Graphene - Large Beam

EMDB-18028: 
Charging of vitreous samples in cryogenic electron microscopy mitigated by graphene - BfrB - Dataset 2 - Quantifoil 300 mesh R1.2/1.3 with Graphene - Small Beam

EMDB-18029: 
Charging of vitreous samples in cryogenic electron microscopy mitigated by graphene - BfrB - Dataset 1 - Quantifoil 300 mesh R1.2/1.3 - Small Beam

EMDB-18030: 
Charging of vitreous samples in cryogenic electron microscopy mitigated by graphene - BfrB - Dataset 3 - Quantifoil 300 mesh R1.2/1.3 - Large Beam

EMDB-26348: 
I-F3b Cascade-TniQ full R-loop complex

PDB-7u5d: 
I-F3b Cascade-TniQ full R-loop complex

EMDB-26349: 
I-F3b Cascade-TniQ full R-loop complex

PDB-7u5e: 
I-F3b Cascade-TniQ partial R-loop complex

EMDB-15853: 
Tetrameric structure of 47 N-terminally truncated human tryptophan hydroxylase 2 with dimerized regulatory domains

EMDB-16223: 
Cryo-EM structure of the catalytic domain tetramer of N-terminally truncated human tryptophan hydroxylase 2

EMDB-29657: 
Semi-synthetic CoA-alpha-Synuclein Constructs Trap N-terminal Acetyltransferase NatB for Binding Mechanism Studies

PDB-8g0l: 
Semi-synthetic CoA-alpha-Synuclein Constructs Trap N-terminal Acetyltransferase NatB for Binding Mechanism Studies

EMDB-29735: 
Structure of nucleosome-bound Sirtuin 6 deacetylase

PDB-8g57: 
Structure of nucleosome-bound Sirtuin 6 deacetylase
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
