-検索条件
-検索結果
検索 (著者・登録者: moore & m)の結果535件中、1から50件目までを表示しています

EMDB-41874: 
CryoEM structure of A/Solomon Islands/3/2006 H1 HA in complex with 05.GC.w2.3C10-H1_SI06

PDB-8u44: 
CryoEM structure of A/Solomon Islands/3/2006 H1 HA in complex with 05.GC.w2.3C10-H1_SI06

EMDB-40815: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies C3V5, V1V3, N611 and base from participant 017

EMDB-40816: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies gp41-N611/FP and base from participant 03

EMDB-40817: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies gp41-N611/FP and base from participant 07

EMDB-40818: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies gp120-GH and base from participant 09

EMDB-40819: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies C3V5, V1V3, gp41-GH/FP and base from participant 11
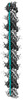
EMDB-19042: 
MAP7 MTBD (microtubule binding domain) decorated microtubule protofilament

EMDB-19043: 
MAP7 MTBD (microtubule binding domain) decorated Taxol stabilized microtubule (13pf) protofilament

EMDB-19044: 
MAP7 MTBD (microtubule binding domain) decorated Taxol stabilized microtubule (14pf) protofilament

PDB-8rc1: 
MAP7 MTBD (microtubule binding domain) decorated microtubule protofilament

EMDB-41888: 
Structure of Apo CXCR4/Gi complex

EMDB-41889: 
Structure of CXCL12-bound CXCR4/Gi complex

EMDB-41890: 
Structure of AMD3100-bound CXCR4/Gi complex

EMDB-41891: 
Structure of REGN7663 Fab-bound CXCR4/Gi complex

EMDB-41892: 
Structure of REGN7663-Fab bound CXCR4

EMDB-41893: 
Structure of trimeric CXCR4 in complex with REGN7663 Fab

EMDB-41894: 
Structure of tetrameric CXCR4 in complex with REGN7663 Fab

EMDB-17557: 
Cryo-EM structure of cortactin-stabilized Arp2/3-complex nucleated actin branches-Local refined map on mother filament

EMDB-17553: 
Cryo-EM structure of cortactin-stabilized Arp2/3 complex nucleated actin branches-Daughter filament consensus map

EMDB-17554: 
Cryo-EM structure of cortactin-stabilized Arp2/3 nucleated actin branches-Local refined map on Arp2/3 complex

EMDB-17555: 
Cryo-EM structure of cortactin-stabilized Arp2/3-complex nucleated actin branches-Local refined map on the daughter filament and cortactin density

EMDB-17556: 
Cryo-EM structure of cortactin-stabilized Arp2/3-complex nucleated actin branches-Local refined map on capping protein

EMDB-17558: 
Cryo-EM structure of cortactin stabilized Arp2/3-complex nucleated actin branches

PDB-8p94: 
Cryo-EM structure of cortactin stabilized Arp2/3-complex nucleated actin branches

EMDB-42124: 
Cryo-EM structure of human STEAP1 in complex with AMG 509 Fab

PDB-8ucd: 
Cryo-EM structure of human STEAP1 in complex with AMG 509 Fab

EMDB-40088: 
HIV-1 Env subtype C CZA97.12 SOSIP.664 in complex with 3BNC117 Fab

PDB-8gje: 
HIV-1 Env subtype C CZA97.12 SOSIP.664 in complex with 3BNC117 Fab

EMDB-27703: 
Structure of RBD directed antibody DH1047 in complex with SARS-CoV-2 spike: Local refinement of RBD-Fab interace

PDB-8dtk: 
Structure of RBD directed antibody DH1047 in complex with SARS-CoV-2 spike: Local refinement of RBD-Fab interace

EMDB-28570: 
CryoEM structure of PN45545 TCR-CD3 complex

EMDB-28571: 
CryoEM structure of PN45545 TCR-CD3 in complex with HLA-A2 MAGEA4 (230-239)

EMDB-28572: 
CryoEM structure of PN45428 TCR-CD3 in complex with HLA-A2 MAGEA4

EMDB-28573: 
CryoEM structure of HLA-A2 bound to MAGEA4 (230-239) peptide

EMDB-28574: 
CryoEM structure of HLA-A2 bound to MAGEA8 (232-241) peptide

PDB-8es7: 
CryoEM structure of PN45545 TCR-CD3 complex

PDB-8es8: 
CryoEM structure of PN45545 TCR-CD3 in complex with HLA-A2 MAGEA4 (230-239)

PDB-8es9: 
CryoEM structure of PN45428 TCR-CD3 in complex with HLA-A2 MAGEA4

PDB-8esa: 
CryoEM structure of HLA-A2 bound to MAGEA4 (230-239) peptide

PDB-8esb: 
CryoEM structure of HLA-A2 bound to MAGEA8 (232-241) peptide

PDB-8sbe: 
Structure of the rat vesicular glutamate transporter 2 determined by single-particle Cryo-EM

EMDB-27232: 
Subtomogram of Fluad vaccine hemagglutinin spiked nanodisc

EMDB-27233: 
Tomogram of Flublok vaccine hemagglutinin starfish complexes

EMDB-26574: 
KS-AT di-domain of mycobacterial Pks13 with endogenous KS ligand bound

EMDB-27002: 
ACP1-KS-AT domains of mycobacterial Pks13

EMDB-27003: 
KS-AT domains of mycobacterial Pks13 with inward AT conformation

EMDB-27004: 
KS-AT domains of mycobacterial Pks13 with outward AT conformation

EMDB-27005: 
ACP1-KS-AT domains of mycobacterial Pks13

PDB-7uk4: 
KS-AT di-domain of mycobacterial Pks13 with endogenous KS ligand bound
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
