-検索条件
-検索結果
検索 (著者・登録者: mikel & v)の結果105件中、1から50件目までを表示しています
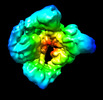
EMDB-15205: 
cryoEM structure of the catalytically inactive EndoS from S. pyogenes in complex with the Fc region of immunoglobulin G1

PDB-8a64: 
cryoEM structure of the catalytically inactive EndoS from S. pyogenes in complex with the Fc region of immunoglobulin G1.
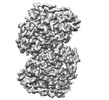
EMDB-15036: 
Cryo-EM structure of "CT-CT dimer" of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz6: 
Cryo-EM structure of "CT-CT dimer" of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15028: 
Cryo-EM structure of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15029: 
Cryo-EM structure of "CT oxa" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA
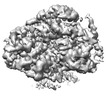
EMDB-15030: 
Cryo-EM structure of "CT empty" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA
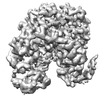
EMDB-15031: 
Cryo-EM structure of "CT react" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15032: 
Cryo-EM structure of "CT pyr" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15033: 
Cryo-EM structure of "BC react" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15034: 
Cryo-EM structure of "BC closed" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15035: 
Cryo-EM structure of "BC open" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15037: 
Cryo-EM structure of Lactococcus lactis pyruvate carboxylase with acetyl-CoA and cyclic di-AMP

PDB-7zyy: 
Cryo-EM structure of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zyz: 
Cryo-EM structure of "CT oxa" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz0: 
Cryo-EM structure of "CT empty" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz1: 
Cryo-EM structure of "CT react" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz2: 
Cryo-EM structure of "CT pyr" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz3: 
Cryo-EM structure of "BC react" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz4: 
Cryo-EM structure of "BC closed" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz5: 
Cryo-EM structure of "BC open" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz8: 
Cryo-EM structure of Lactococcus lactis pyruvate carboxylase with acetyl-CoA and cyclic di-AMP

EMDB-12636: 
CspA-27 cotranslational folding intermediate 1

EMDB-12928: 
CspA-27 cotranslational folding intermediate 2

EMDB-12929: 
CspA-27 cotranslational folding intermediate 3

EMDB-12930: 
CspA-70 cotranslational folding intermediate 2

EMDB-13055: 
CspA-70 cotranslational folding intermediate 1

PDB-7nww: 
CspA-27 cotranslational folding intermediate 1

PDB-7oif: 
CspA-27 cotranslational folding intermediate 2

PDB-7oig: 
CspA-27 cotranslational folding intermediate 3

PDB-7oii: 
CspA-70 cotranslational folding intermediate 2

PDB-7ot5: 
CspA-70 cotranslational folding intermediate 1

EMDB-11606: 
Cryo-EM map of the large glutamate dehydrogenase composed of 180 kDa subunits from Mycobacterium smegmatis (open conformation)

EMDB-11612: 
Cryo-EM map of the large glutamate dehydrogenase composed of 180 kDa subunits from Mycobacterium smegmatis (close conformation)

EMDB-11613: 
Cryo-EM map of the large glutamate dehydrogenase composed of 180 kDa subunits from Mycobacterium smegmatis (monomer)

PDB-7a1d: 
Cryo-EM map of the large glutamate dehydrogenase composed of 180 kDa subunits from Mycobacterium smegmatis (open conformation)

EMDB-10373: 
CryoEM structure for Turnip mosaic virus (TuMV)

EMDB-10374: 
CryoEM structure for viral like particles (VLPs) of Turnip mosaic virus (TuMV).

PDB-6t34: 
Atomic model for Turnip mosaic virus (TuMV)

EMDB-0172: 
Cryo-EM structure of the archaeal extremophilic internal membrane containing Haloarcula hispanica icosahedral virus 2 (HHIV-2) at 3.78 Angstroms resolution.

PDB-6h82: 
Cryo-EM structure of the archaeal extremophilic internal membrane containing Haloarcula hispanica icosahedral virus 2 (HHIV-2) at 3.78 Angstroms resolution.

EMDB-0050: 
DNA-devoid HCIV-1 virus particle

EMDB-0072: 
Localized reconstructed spike HCIV-1

EMDB-0073: 
Localized reconstructed spike HCIV-1

EMDB-0131: 
HHIV-2 pentameric vertex complex

EMDB-0174: 
Cryo-EM structure of archaeal extremophilic internal membrane-containing Haloarcula californiae icosahedral virus 1 (HCIV-1) at 3.74 Angstroms resolution.

PDB-6h9c: 
Cryo-EM structure of archaeal extremophilic internal membrane-containing Haloarcula californiae icosahedral virus 1 (HCIV-1) at 3.74 Angstroms resolution.

EMDB-3785: 
Structure of Watermelon mosaic virus potyvirus.

PDB-5odv: 
Structure of Watermelon mosaic virus potyvirus.

EMDB-3618: 
Bypassing 70S ribosome
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
