-検索条件
-検索結果
検索 (著者・登録者: kim & dy)の結果83件中、1から50件目までを表示しています
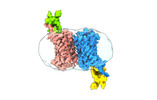
EMDB-36488: 
Structure of Duffy Antigen Receptor for Chemokines (DARC)/ACKR1 in complex with the chemokine, CCL7 (Composite map)

EMDB-37212: 
Structure of Duffy Antigen Receptor for Chemokines (DARC)/ACKR1 in complex with the chemokine, CCL7 (Receptor original map)
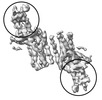
EMDB-37214: 
Structure of Duffy Antigen Receptor for Chemokines (DARC)/ACKR1 in complex with the chemokine, CCL7 (Ligand/CCL7 focused map)

PDB-8jps: 
Structure of Duffy Antigen Receptor for Chemokines (DARC)/ACKR1 in complex with the chemokine, CCL7 (Composite map)
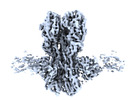
EMDB-41874: 
CryoEM structure of A/Solomon Islands/3/2006 H1 HA in complex with 05.GC.w2.3C10-H1_SI06

EMDB-16375: 
SARS-CoV2 Omicron BA.1 RBD in complex with CAB-A17 antibody

PDB-8c0y: 
SARS-CoV2 Omicron BA.1 RBD in complex with CAB-A17 antibody

EMDB-42970: 
Model and map from local refinement of a CAB-A17 - Omicron Ba.1 spike complex

PDB-8v4f: 
Model and map from local refinement of a CAB-A17 - Omicron Ba.1 spike complex
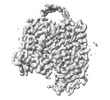
EMDB-35377: 
Cryo-EM structure of GPR156 of GPR156-miniGo-scFv16 complex (local refine)

EMDB-35378: 
Cryo-EM structure of miniGo-scFv16 of GPR156-miniGo-scFv16 complex (local refine)

EMDB-35380: 
Cryo-EM structure of GPR156-miniGo-scFv16 complex

EMDB-35382: 
Cryo-EM structure of GPR156A/B of G-protein free GPR156 (local refine)

EMDB-35389: 
Cryo-EM structure of GPR156C/D of G-protein free GPR156 (local refine)

EMDB-35390: 
Cryo-EM structure of G-protein free GPR156

PDB-8ieb: 
Cryo-EM structure of GPR156 of GPR156-miniGo-scFv16 complex (local refine)

PDB-8iec: 
Cryo-EM structure of miniGo-scFv16 of GPR156-miniGo-scFv16 complex (local refine)

PDB-8ied: 
Cryo-EM structure of GPR156-miniGo-scFv16 complex

PDB-8iei: 
Cryo-EM structure of GPR156A/B of G-protein free GPR156 (local refine)

PDB-8iep: 
Cryo-EM structure of GPR156C/D of G-protein free GPR156 (local refine)

PDB-8ieq: 
Cryo-EM structure of G-protein free GPR156

EMDB-29858: 
Hepatitis B virus capsid bound to importin alpha1

EMDB-29756: 
Empty capsid of Hepatitis B virus

EMDB-29785: 
Hepatitis B virus capsid bound to importin alpha1/beta heterodimer

EMDB-28728: 
Structure of 3A10 Fab in complex with A/Moscow/10/1999 (H3N2) influenza virus neuraminidase

EMDB-28729: 
Structure of 1F04 Fab in complex with A/Moscow/10/1999 (H3N2) influenza virus neuraminidase

EMDB-28730: 
Structure of 3C08 Fab in complex with A/Moscow/10/1999 (H3N2) influenza virus neuraminidase

PDB-8ez3: 
Structure of 3A10 Fab in complex with A/Moscow/10/1999 (H3N2) influenza virus neuraminidase

PDB-8ez7: 
Structure of 1F04 Fab in complex with A/Moscow/10/1999 (H3N2) influenza virus neuraminidase

PDB-8ez8: 
Structure of 3C08 Fab in complex with A/Moscow/10/1999 (H3N2) influenza virus neuraminidase

EMDB-28156: 
Cryo-EM structure of human DNMT3B homo-tetramer (form I)

EMDB-28157: 
Cryo-EM structure of human DNMT3B homo-tetramer (form II)

EMDB-28158: 
Cryo-EM structure of human DNMT3B homo-trimer

EMDB-28159: 
Cryo-EM structure of human DNMT3B homo-hexamer

PDB-8eih: 
Cryo-EM structure of human DNMT3B homo-tetramer (form I)

PDB-8eii: 
Cryo-EM structure of human DNMT3B homo-tetramer (form II)

PDB-8eij: 
Cryo-EM structure of human DNMT3B homo-trimer

PDB-8eik: 
Cryo-EM structure of human DNMT3B homo-hexamer

EMDB-35828: 
Cryo-EPty SPA at CSA of 1.03 mrad

EMDB-35916: 
Cryo-EPty SPA at CSA of 3.26 mrad

EMDB-35917: 
Cryo-EPty SPA at CSA of 4.83 mrad

EMDB-28163: 
Cryo-EM map of octopus sensory receptor CRT1

EMDB-28167: 
Cryo-EM map of squid sensory receptor CRB1

PDB-8eis: 
Cryo-EM structure of octopus sensory receptor CRT1

PDB-8eiz: 
Cryo-EM structure of squid sensory receptor CRB1

EMDB-26735: 
Hantavirus ANDV Gn(H) protein in complex with 2 Fabs ANDV-5 and ANDV-34
EMDB-26736: 
Hantavirus MAPV Gn(H)/Gc protein in complex with 2 Fabs SNV-24 and SNV-53

EMDB-27318: 
CryoEM structure of Hantavirus ANDV Gn(H) protein complex with 2Fabs ANDV-5 and ANDV-34

PDB-8dbz: 
CryoEM structure of Hantavirus ANDV Gn(H) protein complex with 2Fabs ANDV-5 and ANDV-34

EMDB-28523: 
Structure of interleukin receptor common gamma chain (IL2Rgamma) in complex with two antibodies
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
