-検索条件
-検索結果
検索 (著者・登録者: kay & h)の結果1,059件中、1から50件目までを表示しています

EMDBエントリ 画像なし
EMDB-39212: 
Cryo-EM structure of Dragon Grouper nervous necrosis virus-like particle at pH8.0 (3.23A)

EMDBエントリ 画像なし
EMDB-39213: 
Cryo-EM structure of Dragon Grouper nervous necrosis virus-like particle at pH6.5 (2.82A)

EMDBエントリ 画像なし
EMDB-39214: 
Cryo-EM structure of Dragon Grouper nervous necrosis virus-like particle at pH5.0 (3.52A)

EMDBエントリ 画像なし
EMDB-39215: 
Cryo-EM structure of Dragon Grouper nervous necrosis virion at pH6.5 (3.12A)

EMDBエントリ 画像なし
EMDB-39217: 
Cryo-EM structure of Dragon Grouper nervous necrosis virion at pH5.0 (4.36A)

PDB-8yf6: 
Cryo-EM structure of Dragon Grouper nervous necrosis virus-like particle at pH8.0 (3.23A)

PDB-8yf7: 
Cryo-EM structure of Dragon Grouper nervous necrosis virus-like particle at pH6.5 (2.82A)

PDB-8yf8: 
Cryo-EM structure of Dragon Grouper nervous necrosis virus-like particle at pH5.0 (3.52A)

PDB-8yf9: 
Cryo-EM structure of Dragon Grouper nervous necrosis virion at pH6.5 (3.12A)

EMDBエントリ 画像なし
EMDB-16860: 
Ivabradine bound to HCN4 channel

PDB-8ofi: 
Ivabradine bound to HCN4 channel

EMDB-45078: 
E.coli GroEL + PBZ1587 inhibitor

EMDB-45079: 
E.coli GroEL apoenzyme

EMDB-45080: 
E.Faecium GroEL

EMDB-45098: 
E.coli GroEL + PBZ1587 inhibitor C1 reconstruction

PDB-9c0b: 
E.coli GroEL + PBZ1587 inhibitor

PDB-9c0c: 
E.coli GroEL apoenzyme

PDB-9c0d: 
E.Faecium GroEL

EMDB-18135: 
Cryo-EM structure of the methanogenic Na+ translocating N5-methyl-H4MPT:CoM methyltransferase complex

PDB-8q3v: 
Cryo-EM structure of the methanogenic Na+ translocating N5-methyl-H4MPT:CoM methyltransferase complex

EMDB-17762: 
Cryo-EM structure of active Phthaloyl-CoA decarboxylase (Pcd) complex with prFMN bound

PDB-8pmk: 
Cryo-EM structure of active Phthaloyl-CoA decarboxylase (Pcd) complex with prFMN bound

EMDB-41107: 
CryoEM structure of TR-TRAP

PDB-8t9d: 
CryoEM structure of TR-TRAP
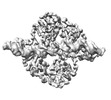
EMDB-18864: 
Central glycolytic genes regulator (CggR) bound to DNA operator

PDB-8r3g: 
Central glycolytic genes regulator (CggR) bound to DNA operator

EMDB-40968: 
Atomic model of the mammalian Mediator complex with MED26 subunit

EMDB-40972: 
CryoEM map of TR-TRAP

EMDB-40975: 
CryoEM map of mouse mediator complex with alternate conformation CKM module

PDB-8t1i: 
Atomic model of the mammalian Mediator complex with MED26 subunit
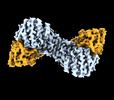
EMDB-44123: 
Cryo-EM density of GluK2 amino-terminal domain (GluK2-ATD) from the open-state structure of kainate receptor GluK2 in complex with agonist glutamate and positive allosteric modulator BPAM344 bound to ConA

EMDB-44126: 
Open state of kainate receptor GluK2 in complex with agonist glutamate and positive allosteric modulator BPAM344 bound to two concanavalin A dimers

EMDB-44127: 
Open state of kainate receptor GluK2 in complex with agonist glutamate and positive allosteric modulator BPAM344 bound to one concanavalin A dimer

EMDB-17298: 
Cryo-EM structure of Phthaloyl-CoA decarboxylase (Pcd) bound with substrate analog/inhibitor, 2-CN-benzoyl-CoA

PDB-8oz5: 
Cryo-EM structure of Phthaloyl-CoA decarboxylase (Pcd) bound with substrate analog/inhibitor, 2-CN-benzoyl-CoA

EMDB-17324: 
Cryo-EM structure of Phthaloyl-CoA decarboxylase (Pcd) bound with product, benzoyl-CoA

PDB-8p02: 
Cryo-EM structure of Phthaloyl-CoA decarboxylase (Pcd) bound with product, benzoyl-CoA

EMDB-19837: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state with 1-bp DNA mismatch

EMDB-19838: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state active site with 1-bp DNA mismatch

EMDB-19839: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state with 1-bp DNA mismatch consensus map
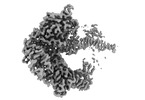
EMDB-19840: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state with 1-bp DNA mismatch catalytic core focused map

EMDB-19841: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state with 1-bp DNA mismatch processivity factor focused refinement

PDB-9enp: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state with 1-bp DNA mismatch

PDB-9enq: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state active site with 1-bp DNA mismatch

EMDB-19066: 
TREK2 in OGNG/CHS detergent micelle with biparatopic inhibitory nanobody Nb6158

EMDB-44124: 
Structure of concanavalin A (ConA) dimer from the open-state structure of kainate receptor GluK2 in complex with agonist glutamate and positive allosteric modulator BPAM344 bound to one ConA dimer. Type II interface between GluK2 ligand-binding domain and ConA

EMDB-44125: 
Structure of concanavalin A (ConA) dimer from the open-state structure of kainate receptor GluK2 in complex with agonist glutamate and positive allosteric modulator BPAM344 bound to two ConA dimers. Type I interface between GluK2 ligand-binding domain and ConA

EMDB-44128: 
Ligand-binding and transmembrane domains of kainate receptor GluK2 in the open state, a complex with agonist glutamate and positive allosteric modulator BPAM344

EMDB-44129: 
Open state of kainate receptor GluK2 in complex with agonist glutamate and positive allosteric modulator BPAM344 bound to two concanavalin A dimers. Composite map.

EMDB-44130: 
Open state of kainate receptor GluK2 in complex with agonist glutamate and positive allosteric modulator BPAM344 bound to one concanavalin A dimer. Composite map.
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
