-検索条件
-検索結果
検索 (著者・登録者: hirsch & e)の結果58件中、1から50件目までを表示しています

EMDB-16759: 
Cryo-EM structure of retinal-free proteoopsin bound to decanoate

EMDB-16795: 
Cryo-EM structure of pentameric proteorhodopsin A18L mutant

EMDB-16796: 
Cryo-EM structure of hexameric proteorhodopsin A18L mutant

PDB-8cnk: 
Cryo-EM structure of retinal-free proteoopsin bound to decanoate

PDB-8cqc: 
Cryo-EM structure of pentameric proteorhodopsin A18L mutant

PDB-8cqd: 
Cryo-EM structure of hexameric proteorhodopsin A18L mutant

EMDB-29070: 
Cryo-electron microscopy structure of human mt-SerRS in complex with mt-tRNA(UGA-TL)

PDB-8ffy: 
Cryo-electron microscopy structure of human mt-SerRS in complex with mt-tRNA(UGA-TL)
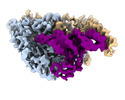
EMDB-26310: 
Cryo-electron microscopy structure of human mt-SerRS in complex with mt-tRNA (GCU)
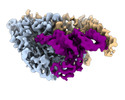
EMDB-26311: 
Cryo-electron microscopy structure of human mt-SerRS in complex with mt-tRNA(GCU-TL)

PDB-7u2a: 
Cryo-electron microscopy structure of human mt-SerRS in complex with mt-tRNA (GCU)

PDB-7u2b: 
Cryo-electron microscopy structure of human mt-SerRS in complex with mt-tRNA(GCU-TL)

EMDB-12191: 
Cryo-EM structure of the Phosphatidylinositol 3-kinase type 2a (PI3KC2a) of the class II PI3K family

EMDB-24795: 
Mouse heavy chain apoferritin determined in presence of 1.66% glycerol.

EMDB-24796: 
Mouse heavy chain apoferritin determined in presence of 20% glycerol (200keV)

EMDB-24797: 
Mouse heavy chain apoferritin determined in presence of 20% glycerol (300keV)

EMDB-24798: 
Rabbit muscle aldolase determined in presence of 20% v/v glycerol

EMDB-24799: 
Rabbit muscle aldolase determined in the presence of 20% v/v glycerol.

EMDB-11955: 
Cryo-EM structure of the green-light absorbing proteorhodopsin

PDB-7b03: 
Cryo-EM structure of the green-light absorbing proteorhodopsin

EMDB-11292: 
SARS-CoV-2 Nsp1 bound to the human LYAR-80S ribosome complex

EMDB-11299: 
SARS-CoV-2 Nsp1 bound to the human LYAR-80S-eEF1a ribosome complex

EMDB-11301: 
SARS-CoV-2 Nsp1 bound to a pre-40S-like ribosome complex

PDB-6zmi: 
SARS-CoV-2 Nsp1 bound to the human LYAR-80S ribosome complex

PDB-6zmo: 
SARS-CoV-2 Nsp1 bound to the human LYAR-80S-eEF1a ribosome complex

PDB-6zmt: 
SARS-CoV-2 Nsp1 bound to a pre-40S-like ribosome complex

EMDB-11289: 
SARS-CoV-2 Nsp1 bound to the human CCDC124-80S-eERF1 ribosome complex

PDB-6zme: 
SARS-CoV-2 Nsp1 bound to the human CCDC124-80S-eERF1 ribosome complex

EMDB-11276: 
SARS-CoV-2 Nsp1 bound to the human 40S ribosomal subunit

EMDB-11288: 
SARS-CoV-2 Nsp1 bound to the human CCDC124-80S-EBP1 ribosome complex

EMDB-11310: 
SARS-CoV-2 Nsp1 bound to a pre-40S-like ribosome complex - state 2

EMDB-11325: 
SARS-CoV-2 Nsp1 bound to a human 43S preinitiation ribosome complex - state 1

EMDB-11335: 
SARS-CoV-2 Nsp1 bound to a human 43S preinitiation ribosome complex - state 2

PDB-6zlw: 
SARS-CoV-2 Nsp1 bound to the human 40S ribosomal subunit

PDB-6zm7: 
SARS-CoV-2 Nsp1 bound to the human CCDC124-80S-EBP1 ribosome complex

PDB-6zn5: 
SARS-CoV-2 Nsp1 bound to a pre-40S-like ribosome complex - state 2

PDB-6zon: 
SARS-CoV-2 Nsp1 bound to a human 43S preinitiation ribosome complex - state 1

PDB-6zp4: 
SARS-CoV-2 Nsp1 bound to a human 43S preinitiation ribosome complex - state 2

EMDB-21516: 
Cryo-EM structure of anti-CRISPR AcrIF9, bound to the type I-F crRNA-guided CRISPR surveillance complex

EMDB-21517: 
Cryo-EM structure of anti-CRISPR AcrIF9 and dsDNA, bound to the type I-F crRNA-guided CRISPR surveillance complex

PDB-6w1x: 
Cryo-EM structure of anti-CRISPR AcrIF9, bound to the type I-F crRNA-guided CRISPR surveillance complex

PDB-6whi: 
Cryo-electron microscopy structure of the type I-F CRISPR RNA-guided surveillance complex bound to the anti-CRISPR AcrIF9

EMDB-9195: 
ADP-bound human mitochondrial Hsp60-Hsp10 football complex

EMDB-9196: 
ADP-bound human mitochondrial Hsp60-Hsp10 half-football complex

PDB-6mrc: 
ADP-bound human mitochondrial Hsp60-Hsp10 football complex

PDB-6mrd: 
ADP-bound human mitochondrial Hsp60-Hsp10 half-football complex

EMDB-9115: 
Structure of the human TRPV3 channel in the apo conformation

EMDB-9117: 
Structure of the human TRPV3 channel in a putative sensitized conformation

EMDB-9119: 
Structure of human TRPV3 in the presence of 2-APB in C4 symmetry

EMDB-9120: 
Structure of human TRPV3 in the presence of 2-APB in C2 symmetry (1)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
