-検索条件
-検索結果
検索 (著者・登録者: hill & jm)の結果55件中、1から50件目までを表示しています

EMDB-43746: 
Plasmodium falciparum 20S proteasome bound to an inhibitor

EMDB-27674: 
LRRC8A:C conformation 2 (oblong) top mask

EMDB-27675: 
LRRC8A:C conformation 2 (oblong) LRR mask

EMDB-27676: 
LRRC8A:C conformation 2 (oblong)

EMDB-27677: 
LRRC8A:C conformation 1 (round) top focus

EMDB-27678: 
LRRC8A:C conformation 1 (round) LRR focus 1

EMDB-27679: 
LRRC8A:C conformation 1 (round) LRR focus 2

EMDB-27681: 
LRRC8A:C conformation 1 (round) LRR focus 3

EMDB-27682: 
LRRC8A:C conformation 1 (round)

EMDB-27686: 
LRRC8A:C in MSPE3D1 nanodisc top focus

EMDB-27687: 
LRRC8A:C in MSP1E3D1 nanodisc

EMDB-28894: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-28895: 
LRRC8A(T48D):C conformation 2 LRR focus

EMDB-28897: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-28898: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-28901: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-28903: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-28904: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-28905: 
LRRC8A(T48D):C conformation 2 top focus
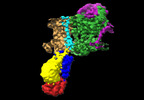
EMDB-14347: 
Cryo-EM map of the WT KdpFABC complex in the E2-P conformation, stabilised with the inhibitor orthovanadate
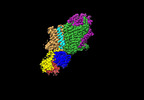
EMDB-14911: 
Cryo-EM map of the WT KdpFABC complex in the E1-P tight conformation, stabilised with the inhibitor orthovanadate
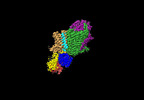
EMDB-14912: 
Cryo-EM map of the WT KdpFABC complex in the E1-P tight conformation, under turnover conditions
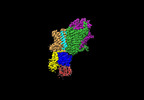
EMDB-14913: 
Cryo-EM map of the WT KdpFABC complex in the E1_ATPearly conformation, under turnover conditions
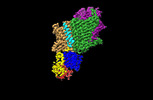
EMDB-14914: 
Cryo-EM structure of the KdpFABC complex in a nucleotide-free E1 conformation loaded with K+

EMDB-14915: 
Cryo-EM structure of the KdpFABC complex in a nucleotide-free E1 conformation loaded with K+
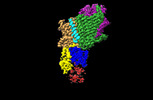
EMDB-14916: 
Cryo-EM structure of the KdpFABC complex in a nucleotide-free E1 conformation loaded with K+
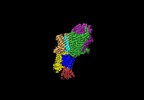
EMDB-14917: 
Cryo-EM map of the WT KdpFABC complex in the E1-P_ADP conformation, under turnover conditions

EMDB-14918: 
Cryo-EM map of the unphosphorylated KdpFABC complex in the E2-P conformation, under turnover conditions

EMDB-14919: 
Cryo-EM map of the unphosphorylated KdpFABC complex in the E1-P_ADP conformation, under turnover conditions

EMDB-26483: 
Actin organization during clathrin-mediated endocytosis

EMDB-26484: 
In situ map of the clathrin hub

EMDB-12478: 
Cryo-EM structure of the KdpFABC complex in an E1-ATP conformation loaded with K+

EMDB-12482: 
Rb-loaded cryo-EM structure of the E1-ATP KdpFABC complex.

EMDB-22829: 
Human Tom70 in complex with SARS CoV2 Orf9b

EMDB-22078: 
Characterization of the SARS-CoV-2 S Protein: Biophysical, Biochemical, Structural, and Antigenic Analysis

EMDB-10277: 
Cryo-EM structure of human SERINC5 in LMNG micelles, bound to Fab.

EMDB-10279: 
CryoEM structure of SERINC from Drosophila melanogaster

EMDB-10290: 
Structure of Fanconi anaemia core complex (consensus map)

EMDB-10291: 
Structure of the Fanconi Anaemia core complex (focussed map for top region)

EMDB-10292: 
Structure of the Fanconi Anaemia core complex (focussed map for middle region)

EMDB-10293: 
Structure of the Fanconi Anaemia core complex (focussed map for base region)

EMDB-10294: 
Structure of the Fanconi anaemia core subcomplex

EMDB-0443: 
Vps4 with Cyclic Peptide Bound in the Central Pore

EMDB-20139: 
Vps4 with Cyclic Peptide Bound in the Central Pore (Focused Classification of Subunit F, State1)

EMDB-20140: 
Vps4 with Cyclic Peptide Bound in the Central Pore (Focused Classification of Subunit F, State2)

EMDB-20141: 
Vps4 with Cyclic Peptide Bound in the Central Pore (Focused Classification of Subunit F, State3)

EMDB-20142: 
Vps4 with Cyclic Peptide Bound in the Central Pore

EMDB-20144: 
Vps4 with Cyclic Peptide Bound in the Central Pore (Map for Peptide cF30)

EMDB-20147: 
Vps4 with Cyclic Peptide Bound in the Central Pore (Map for Peptide cFF30)

EMDB-3753: 
Negative stain 3D single particle reconstruction of the isolated surface adhesin P140/P110 complex of Mycoplasma genitalium.
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
