-検索条件
-検索結果
検索 (著者・登録者: gil & d)の結果1,485件中、1から50件目までを表示しています

EMDB-41542: 
Polyclonal immune complex of Fab binding the H2 HA from serum of subject 3-3 at week 4
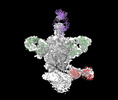
EMDB-44246: 
Cryo-EM structure of HIV-1 JRFL v6 Env in complex with vaccine-elicited, Membrane Proximal External Region (MPER) directed antibody DH1317.4.

EMDB-41578: 
mGluR3 class 1 in the presence of the antagonist LY 341495

EMDB-45242: 
mGluR3 in the presence of the antagonist LY 341495 and positive allosteric modulator VU6023326 class 2

PDB-8trd: 
mGluR3 class 1 in the presence of the antagonist LY 341495

EMDB-42400: 
RORC mRNA 3'UTR riboswitch A97G/G98A mutant class C

EMDB-42401: 
RORC mRNA 3'UTR riboswitch 77-GA mutant class A

EMDB-42403: 
RORC mRNA 3'UTR riboswitch 117-AC mutant class C

EMDB-42404: 
RORC mRNA 3'UTR riboswitch 117-AC mutant class B

EMDB-41501: 
mGluR3 in the presence of the agonist LY379268 and PAM VU6023326

EMDB-41567: 
Metabotropic glutamate receptor 3 class 3 bound to antagonist LY 341495

EMDB-41568: 
mGluR3 in the presence of the agonist LY379268

EMDB-41577: 
mGluR3 in the presence of the antagonist LY 341495 and positive allosteric modulator VU6023326

EMDB-44861: 
metabotropic glutamate receptor subtype three bound to the antagonist LY 341495, class two

PDB-8tqb: 
mGluR3 in the presence of the agonist LY379268 and PAM VU6023326

PDB-8tr0: 
Metabotropic glutamate receptor 3 class 3 bound to antagonist LY 341495

PDB-8tr2: 
mGluR3 in the presence of the agonist LY379268

PDB-8trc: 
mGluR3 in the presence of the antagonist LY 341495 and positive allosteric modulator VU6023326

EMDB-16825: 
Structure of human terminal uridylyltransferase 7 (hTUT7/ZCCHC6)

EMDB-17084: 
Structure of human terminal uridylyltransferase 7 (hTUT7/ZCCHC6) bound with pre-let7g miRNA and UTPalphaS
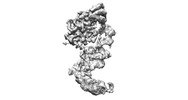
EMDB-17086: 
Human terminal uridylyltransferase 7 (TUT7/ZCCHC6) bound with pre-let7g miRNA and Lin28A - complex 1
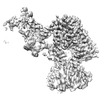
EMDB-17087: 
Human terminal uridylyltransferase 7 (TUT7/ZCCHC6) bound with pre-let7g miRNA and Lin28A - complex 2

PDB-8oef: 
Structure of human terminal uridylyltransferase 7 (hTUT7/ZCCHC6)

PDB-8opp: 
Structure of human terminal uridylyltransferase 7 (hTUT7/ZCCHC6) bound with pre-let7g miRNA and UTPalphaS

PDB-8ops: 
Human terminal uridylyltransferase 7 (TUT7/ZCCHC6) bound with pre-let7g miRNA and Lin28A - complex 1

PDB-8opt: 
Human terminal uridylyltransferase 7 (TUT7/ZCCHC6) bound with pre-let7g miRNA and Lin28A - complex 2

EMDB-18990: 
CryoEM map of tau PHF sarkosyl-extracted from a human AD patient (associated with in situ tomography)

EMDB-18180: 
cryoEM structure of SARS-CoV2 Spike trimer in complex with Fab23

PDB-8q5y: 
cryoEM structure of SARS-CoV2 Spike trimer in complex with Fab23
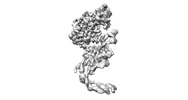
EMDB-17164: 
Structure of human terminal uridylyltransferase 4 (TUT4, ZCCHC11) in complex with pre-let7g miRNA and Lin28A

PDB-8ost: 
Structure of human terminal uridylyltransferase 4 (TUT4, ZCCHC11) in complex with pre-let7g miRNA and Lin28A
EMDB-43144: 
MicroED structure of SARS-CoV-2 main protease (MPro/3CLPro) with missing cone eliminated by suspended drop

PDB-8vd7: 
MicroED structure of SARS-CoV-2 main protease (MPro/3CLPro) with missing cone eliminated by suspended drop

EMDB-50148: 
Tau PHF subtomogram average relating to CS1 extended data Figure 9A

EMDB-50152: 
Tau PHF subtomogram average relating to CS2 Figure 3i-j.

EMDB-50153: 
Tau PHF subtomogram average relating to CS3 extended data Figure 9c

EMDB-50155: 
Tau PHF subtomogram average relating to CS4 extended data Figure 9d

EMDB-50156: 
Tau PHF subtomogram average relating to CS5 extended data Figure 9b

EMDB-50157: 
Tau PHF subtomogram average relating to CS6 extended data Figure 9e

EMDB-50159: 
Tau PHF subtomogram average relating to CS7 extended data Figure 9f

EMDB-50160: 
Tau PHF subtomogram average relating to LOL1_PHF Figure 4g-h

EMDB-50161: 
Tau SF subtomogram average relating to LOL1_SF Figure 4g-h

EMDB-50162: 
Tau SF subtomogram average relating to LOL2_SF Figure 4i-j
EMDB-42527: 
Pre-fusion Measles virus fusion protein complexed with Fab 77
EMDB-42539: 
Structure of the Measles virus Fusion protein in the post-fusion conformation
EMDB-42593: 
Structure of the Measles virus Fusion protein in the pre-fusion conformation

EMDB-42595: 
Structure of the Measles virus Fusion protein in the pre-fusion conformation with bound [FIP-HRC]2-PEG11
EMDB-43827: 
Fab 77-stabilized MeV F ectodomain fragment

PDB-8ut2: 
Pre-fusion Measles virus fusion protein complexed with Fab 77

PDB-8utf: 
Structure of the Measles virus Fusion protein in the post-fusion conformation
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
