-検索条件
-検索結果
検索 (著者・登録者: becker & st)の結果339件中、1から50件目までを表示しています

EMDB-50296: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

PDB-9fbv: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

EMDB-19638: 
YlmH bound to PtRNA-50S

EMDB-19641: 
YlmH bound to stalled 50S subunits with RqcH and PtRNA

PDB-8s1p: 
YlmH bound to PtRNA-50S

PDB-8s1u: 
YlmH bound to stalled 50S subunits with RqcH and PtRNA

EMDB-17218: 
Lipidic amyloid-beta(1-40) fibril - polymorph L1

EMDB-17223: 
Lipidic amyloid-beta(1-40) fibril - polymorph L2

EMDB-17234: 
Lipidic amyloid-beta(1-40) fibril - polymorph L3

EMDB-17235: 
Lipidic amyloid-beta(1-40) fibril - polymorph L2-L3

EMDB-17238: 
Lipidic amyloid-beta(1-40) fibril - polymorph L2-L2

EMDB-17239: 
Lipidic amyloid-beta(1-40) fibril - polymorph L3-L3

PDB-8ovk: 
Lipidic amyloid-beta(1-40) fibril - polymorph L1

PDB-8ovm: 
Lipidic amyloid-beta(1-40) fibril - polymorph L2

PDB-8owd: 
Lipidic amyloid-beta(1-40) fibril - polymorph L3

PDB-8owe: 
Lipidic amyloid-beta(1-40) fibril - polymorph L2-L3

PDB-8owj: 
Lipidic amyloid-beta(1-40) fibril - polymorph L2-L2

PDB-8owk: 
Lipidic amyloid-beta(1-40) fibril - polymorph L3-L3

EMDB-43137: 
SARS-CoV-2 Frameshift Stimulatory Element with Upstream Multibranch Loop

EMDB-16552: 
Structure of the RQT-bound 80S ribosome from S. cerevisiae (C2) - composite map

EMDB-16553: 
Structure of RQT (C1) bound to the stalled ribosome in a disome unit from S. cerevisiae - focused Refinement 80S

EMDB-16554: 
Structure of RQT (C1) bound to the stalled ribosome in a disome unit from S. cerevisiae - focused Refinement RQT

EMDB-29725: 
Vaccine-elicited human antibody 2C06 in complex with HIV-1 envelope trimer BG505 DS-SOSIP

EMDB-29731: 
Vaccine-elicited human antibody 2C09 in complex with HIV-1 envelope trimer BG505 DS-SOSIP

PDB-8g4m: 
Vaccine-elicited human antibody 2C06 in complex with HIV-1 envelope trimer BG505 DS-SOSIP

PDB-8g4t: 
Vaccine-elicited human antibody 2C09 in complex with HIV-1 envelope trimer BG505 DS-SOSIP

EMDB-15280: 
RQT-bound 80S ribosome from S. cerevisiae (C1, raw consensus map RQT)

EMDB-15228: 
RQT-bound 80S ribosome from S. cerevisiae (C1, raw consensus map)

EMDB-17154: 
Cryo-EM structure of CLOCK-BMAL1 bound to a nucleosomal E-box at position SHL+5.8 (consensus and constituent map 1)

EMDB-17155: 
Cryo-EM structure of CLOCK-BMAL1 bound to a nucleosomal E-box at position SHL-6.2 (DNA conformation 1)

EMDB-17156: 
Cryo-EM structure of CLOCK-BMAL1 bound to a nucleosomal E-box at position SHL-6.2 (DNA conformation 2)

EMDB-17157: 
Cryo-EM structure of CLOCK-BMAL1 bound to a nucleosomal E-box at position SHL+5.8 (composite map)

EMDB-17158: 
Cryo-EM structure of CLOCK-BMAL1 bound to a nucleosomal E-box at position SHL+5.8 (constituent map 2 from additional focus classification on PAS domains)

EMDB-17159: 
Cryo-EM map of MYC-MAX-OCT4-LIN28 complex

EMDB-17160: 
Cryo-EM structure of CLOCK-BMAL1 bound to the native Por enhancer nucleosome (map 2, additional 3D classification and flexible refinement)

EMDB-17161: 
Cryo-EM structure of CLOCK-BMAL1 bound to the native Por enhancer nucleosome (map 1)

EMDB-17162: 
MAX-MAX bound to a nucleosome at SHL+5.1 and SHL-6.9.

EMDB-17183: 
OCT4 and MYC-MAX co-bound to a nucleosome

EMDB-17184: 
MYC-MAX bound to a nucleosome at SHL+5.8

PDB-8osj: 
Cryo-EM structure of CLOCK-BMAL1 bound to a nucleosomal E-box at position SHL-6.2 (DNA conformation 1)

PDB-8osk: 
Cryo-EM structure of CLOCK-BMAL1 bound to a nucleosomal E-box at position SHL+5.8 (composite map)

PDB-8osl: 
Cryo-EM structure of CLOCK-BMAL1 bound to the native Por enhancer nucleosome (map 2, additional 3D classification and flexible refinement)

PDB-8ots: 
OCT4 and MYC-MAX co-bound to a nucleosome

PDB-8ott: 
MYC-MAX bound to a nucleosome at SHL+5.8
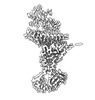
EMDB-15592: 
BA.4/5 SARS-CoV-2 Spike bound to mouse ACE2 (local)

PDB-8aqw: 
BA.4/5 SARS-CoV-2 Spike bound to mouse ACE2 (local)
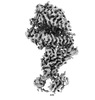
EMDB-15588: 
BA.4/5 SARS-CoV-2 Spike bound to human ACE2 (local)

EMDB-15589: 
Beta SARS-CoV-2 Spike bound to mouse ACE2 (local)

EMDB-15590: 
BA.1 SARS-CoV-2 Spike bound to mouse ACE2 (local)
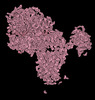
EMDB-15591: 
BA.2.12.1 SARS-CoV-2 Spike bound to mouse ACE2 (local)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
