-検索条件
-検索結果
検索 (著者・登録者: baker & d)の結果624件中、1から50件目までを表示しています

EMDB-42542: 
CryoEM Structure of Allosterically Switchable De Novo Protein sr322, In Closed State without Effector Peptide, off Target Multimeric State

EMDB-42442: 
CryoEM Structure of Allosterically Switchable De Novo Protein sr322, In Closed State without Effector Peptide

EMDB-42491: 
CryoEM Structure of Allosterically Switchable De Novo Protein sr312, in Open State with Effector Peptide

EMDB-45655: 
Cryo-EM structure of alpha5beta1 integrin in complex with NeoNectin

EMDB-28966: 
CryoEM map of de novo designed oligomeric protein C4-71_6x

EMDB-28967: 
CryoEM map of de novo designed oligomeric protein C4-71_8x
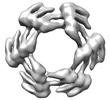
EMDB-28968: 
CryoEM map of de novo designed oligomeric protein C6-71

EMDB-28969: 
CryoEM map of de novo designed oligomeric protein C6-71_6x

EMDB-28970: 
CryoEM map of de novo designed oligomeric protein C6-71_8x

EMDB-28971: 
CryoEM map of de novo designed oligomeric protein C8-71_6x

EMDB-28972: 
CryoEM map of de novo designed oligomeric protein C8-71_8x
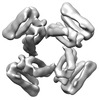
EMDB-28973: 
CryoEM map of de novo designed oligomeric protein C4-81

EMDB-28974: 
CryoEM map of designed oligomeric protein C4-71

EMDB-40602: 
cryoEM map for design HE0537, a D4 symmetric homo-oligomer designed with RFdiffusion.
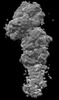
EMDB-44479: 
Cryo-EM structure of synthetic claudin-4 complex with Clostridium perfringens enterotoxin C-terminal domain, sFab COP-2, and Nanobody

PDB-9bei: 
Cryo-EM structure of synthetic claudin-4 complex with Clostridium perfringens enterotoxin C-terminal domain, sFab COP-2, and Nanobody

EMDB-40260: 
CryoEM map of a de novo designed T=4 icosahedral nanocage hierarchically built from pseudosymmetric trimers; design Ico(T=4)-4

EMDB-29939: 
EM map of de novo designed protein nanopore RNR_C6_3 using a reinforcement learning approach

EMDB-42070: 
DpHF18 filament

EMDB-42075: 
DpHF7 filament

EMDB-42088: 
DpHF19 filament

PDB-8uao: 
DpHF18 filament

PDB-8ub3: 
DpHF7 filament

PDB-8ubg: 
DpHF19 filament

EMDB-40267: 
CryoEM map of a T=1 off-target state of design Ico(T=4)-4

EMDB-40268: 
CryoEM map of a de novo designed T=4 octahedral nanocage hierarchically built from pseudosymmetric trimers; design Oct(T=4)-3

EMDB-40269: 
CryoEM map of a T=1 off-target state of design Oct(T=4)-3

EMDB-40208: 
Backbone model of de novo-designed chlorophyll-binding nanocage O32-15

EMDB-40209: 
Chlorophyll-binding region of de novo-designed nanocage O32-15

PDB-8glt: 
Backbone model of de novo-designed chlorophyll-binding nanocage O32-15

EMDB-41907: 
Computationally Designed, Expandable O4 Octahedral Handshake Nanocage
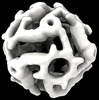
EMDB-42031: 
Computational Designed Nanocage O43_129_+8

EMDB-43318: 
Twistless helix 12 repeat ring design R12B

EMDB-29974: 
Cryo-EM structure of synthetic tetrameric building block sC4

EMDB-41364: 
CryoEM Structure of a Computationally Designed T3 Tetrahedral Nanocage

EMDB-42906: 
Computational Designed Nanocage O43_129

EMDB-42944: 
Computational Designed Nanocage O43_129_+4

PDB-8gel: 
Cryo-EM structure of synthetic tetrameric building block sC4

PDB-8tl7: 
CryoEM Structure of a Computationally Designed T3 Tetrahedral Nanocage

PDB-8v2d: 
Computational Designed Nanocage O43_129

PDB-8v3b: 
Computational Designed Nanocage O43_129_+4

EMDB-42601: 
CryoEM structure of Kappa Opioid Receptor bound to a semi-peptide and Gi1

EMDB-18245: 
Plunge-frozen (control) map of beta-galactosidase

EMDB-29602: 
Three component pH responsive antibody octahedra escape lysosomal degradation

EMDB-18387: 
FtsH1 protease from P.aeruginosa clone C in negative stain

EMDB-18388: 
FtsH2 protease from P.aeruginosa clone C in negative stain

EMDB-18389: 
P.aeruginosa clone C construct PaFtsH2-H1-link32 in negative stain

EMDB-18244: 
ESIBD structure of beta-galactosidase

PDB-8q7y: 
ESIBD structure of beta-galactosidase

EMDB-28958: 
CryoEM structure of designed modular protein oligomer C4-131
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
