[wwPDB] PDBエントリーの検証レポートを更新しました
現在PDBアーカイブで公開されている、X線やNMR、電子顕微鏡法によって解析された全てのエントリーについて、検証レポートを更新しました。
レポート中の統計グラフは2019年12月31日時点のアーカイブ情報を反映したものとなり、また検証レポートの作成過程で計算された電子密度マップ係数ファイルを追加しました。
更新後のレポートは、以下のFTPサイトからご利用頂けます。
- ftp://ftp.wwpdb.org/pub/pdb/validation_reports/ (wwPDB)
- ftp://ftp.rcsb.org/pub/pdb/validation_reports/ (RCSB PDB)
- ftp://ftp.ebi.ac.uk/pub/databases/pdb/validation_reports/ (PDBe)
- ftp://ftp.pdbj.org/pub/pdb/validation_reports/ (PDBj)
更新前の6月10日時点の検証レポートは、RCSB PDBとPDBjのスナップショットにてご確認頂けます。
更新された検証レポートでは、 検証に関する専門委員会(Validation Task Force)の意見を取り入れ、 広く受け入れられている評価基準を使って、構造の品質が評価されています。
更新されたレポートには、 最近導入された糖鎖に関するセクション及び糖鎖データの表現改善プロジェクトで導入された記号による糖鎖表示法(Symbol Nomenclature For Glycan:SNFG)に加えて、 X線構造中のリガンドの幾何学的な検証情報とリガンドとその周辺のモデル構造と電子密度マップの重なりを視覚化した図が追加されています。 リガンドの幾何学的な検証を強調表示した2次元平面図と、結晶解析構造の場合には、電子密度の3次元表示図が含まれます。
さらに、EMマップの解析、PDB構造モデルとEMDBマップの重なり具合の解析に加えて、 ハーフマップ又はFSCデータが登録された場合には、登録者による分解能と検証ソフトが提供する分解能を比較するために、レポートにFSC曲線も表示されます。
検証レポートは、OneDep(wwPDBによる構造データの検証・登録・キュレーションシステム)を通して、登録者に提供されます。 wwPDBは、スタンドアロンの検証サーバーや、 ウェブサービスAPIを使って、 登録者が登録前に構造を検証することを推奨しています。登録者は、データを登録する過程で、レポートの内容を確認し、承認することが求められます。 今後も、X線やNMR、電子顕微鏡法の専門委員会(VTF)の意見を取り入れ、 また、登録者やユーザからのフィードバックを集めながら、更に検証レポートを開発し、改善していく予定です。
wwPDBパートナーは、学術誌の編集者や査読者に、論文の投稿・査読過程の中で、著者に検証レポートの提出を求めるよう、推奨しています。 すでに、複数の学術誌 (Nature, eLife、 The Journal of Biological Chemistry、the International Union of Crystallography (IUCr) journals、 FEBS journals, Journal of Immunology、Angew Chem Int Ed Engl)では、論文投稿の過程で、wwPDB検証レポートの提出が求められます。 レポートには、日付とwwPDBのロゴが表示されており、その構造がどこのwwPDB拠点で処理されたかにかかわらず、同じ情報が含まれています。
さらに詳細な情報や検証レポートの例はwwPDBサイトからご覧いただけます。
コメントやご質問等は、validation@mail.wwpdb.orgまで、英語でご連絡ください。
[ wwPDB News ]
[wwPDB] PDBアーカイブとEMDBアーカイブのスナップショットを公開しました
2020年1月1日現在のPDBアーカイブ (wwPDB:ftp://ftp.wwpdb.org、 PDBj:ftp://ftp.pdbj.org)のスナップショットを、スナップショットサイト (wwPDB:ftp://snapshots.wwpdb.org、PDBj:ftp://snapshots.pdbj.org)に追加しました。 2005年1月以来、毎年、スナップショットをアーカイブし、PDBアーカイブに関する研究に役立つデータセットを提供し続けています。
ディレクトリ 20200101は、159,140件の実験的に決定された座標ファイルと、関連する実験データを含んでいます。 座標及び関連データのファイルは、PDBx/mmCIFフォーマット、PDBフォーマット、XMLフォーマットファイルで、それぞれご利用頂けます。 各ファイルの日付とタイムスタンプは、そのファイルが最後に更新された日付を表しています。 上記ディレクトリ内のPDBアーカイブのスナップショットのサイズは、575GBとなっています。
2020年1月1日現在のEMDBアーカイブ(ftp://ftp.ebi.ac.uk/pub/databases/emdb)のスナップショットは、 ftp://ftp.ebi.ac.uk/pub/databases/emdb_vault/20200101/と、ftp://snapshots.pdbj.org/20200101/からご利用頂けます。 公開済みエントリー(10370件)と廃止されたエントリー(130件)に対する、マップファイルとメタデータのXMLファイルを含んでおり、スナップショットのサイズは、1.7TBとなっています。
[ wwPDB News ]
[wwPDB] NMR制限情報の検証がOneDepから利用可能に
OneDepシステム上で提供されるNMRエントリーのwwPDBの検証レポートに、NMR制限情報の検証結果が追加され、登録者が登録データに含まれる潜在的な不一致を特定することが容易になりました。
wwPDBのNMRデータ検証に関する専門委員会(Validation Task Force , VTF)は、制限情報の検証の進め方に関する提言を公表しています(10.1016/j.str.2013.07.021)。それ以来、wwPDBはNMRの専門家と協力して、制限情報の検証を可能にするために、NEF(NMR Exchange Format)ディクショナリ(10.1038/nsmb.3041)の開発を行ってきました。
2020年3月より、OneDepシステム上で、NMR-STARまたはNEF形式の単一ファイルでNMR実験データ(化学シフトと制限情報)の受付けを開始しました。 今回、制限情報の検証機能がwwPDB検証パッケージに組み込まれ、NEFまたはNMR-STAR形式で制限情報が与えられた場合、OneDepで生成される検証レポートに制限情報の解析結果が含まれるようになりました。
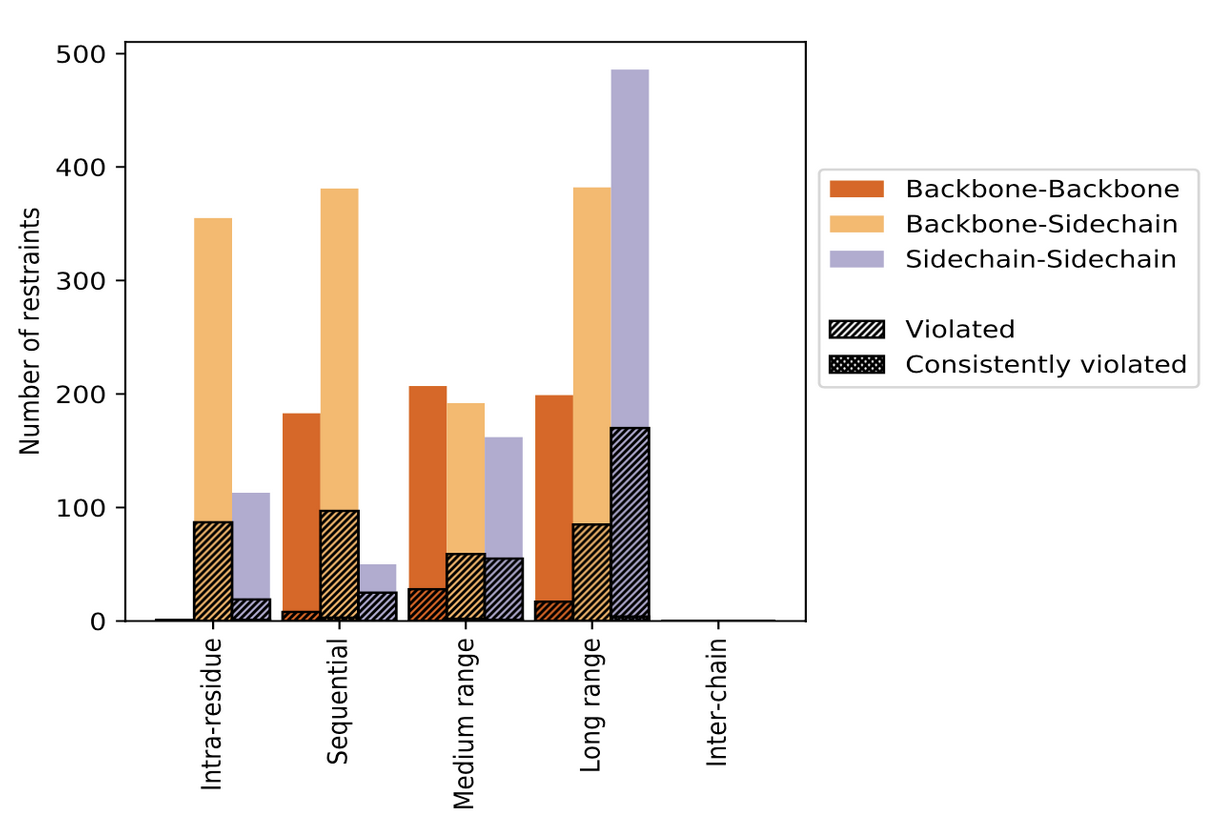
wwPDB検証レポートの制限情報解析は、構造的な束縛を伴う制限情報の統計、制限情報と原子座標モデルの間の違反、各モデルの距離制限及び二面角制限違反の平均出現数、各モデル及び各アンサンブルに対する距離制限及び二面角制限違反の分布解析を含み、グラフまたは表形式で表現されます。
今回追加された機能は、登録者やユーザがNMRデータの潜在的なエラーを特定することに役立つはずです。
NMRエントリーの検証レポートについて、詳しくはこちらをご覧ください。
wwPDBの検証レポートについてご質問などございましたら、validation@mail.wwpdb.orgまで英語でご連絡ください。
 |
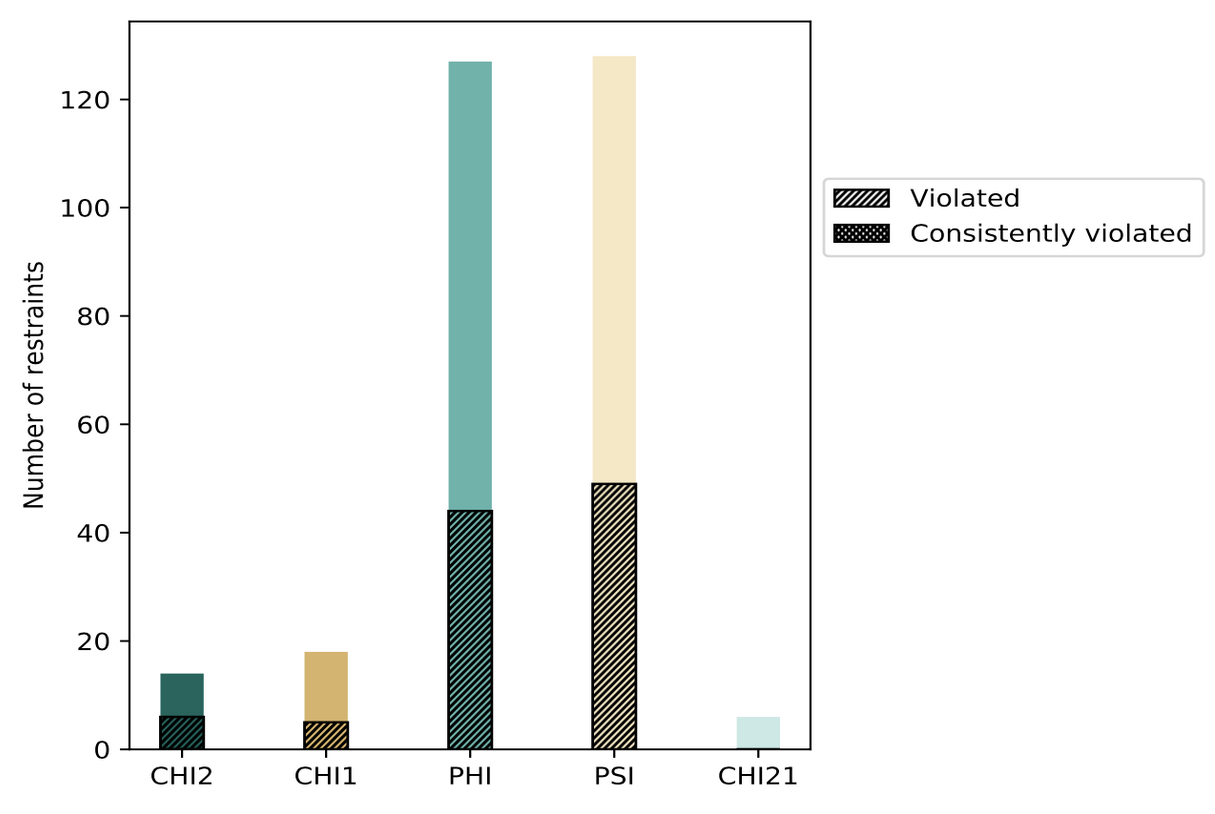 |
| 距離制限と違反の分布(Distribution of distance restraints and violations) | 二面角と違反の分布(Distribution of dihedral-angles and violations) |
[ wwPDB News ]
[wwPDB] 新型コロナウイルスプロテアーゼの構造が公開されました
PDBデータが、蛋白質構造に基づいた新薬発見の出発点となります。
新型コロナウイルス(2019-nCoVコロナウイルス)由来の3CL加水分解酵素(Mpro)の高分解能の結晶構造を、上海科技大学のRao Zihe氏とYang Haitao氏が率いる研究チームが決定しました。 このウイルスのメインプロテアーゼ構造(PDB 6lu7)が直ちに一般に公開されることで、新たに認識されたヒト病原体に関する研究が進むことが期待されます。
新型コロナウイルス(2019-nCoV)の最近の出現により、WHOは公衆衛生上の緊急事態を宣言し、国際的な懸念事項となっています。 この特定のウイルスをより深く理解し、さらなる拡散を防ぐための治療法とワクチンを開発するために、世界中で多大な研究努力がなされています。
この特定のコロナウイルス由来の酵素で決定された構造は、現在のところ6LU7のみですが、PDBには、他のコロナウイルス由来の酵素の構造が多数存在します。 2003年に、関係が深い重症急性呼吸器症候群コロナウイルス(SARS)が出現し、このウイルスについて最初の構造がPDBに登録されましたが、 現在は、SARS蛋白質の構造について200を超えるPDBエントリーが公開されています。 これら関連する蛋白質の構造情報は、コロナウイルスを更に理解するために、又、ウイルスをこれ以上発生させないための新しい治療法とワクチンの開発に不可欠です。
メインプロテアーゼとしても知られるコロナウイルス由来の3CL加水分解酵素(Mpro)は、 ウイルスの蛋白質分解の成熟に不可欠です。 ウイルスのポリ蛋白質の切断を阻害し、感染の拡大を防ぐ低分子薬の発見にとって有望なターゲットであると考えられています。
2019-nCoVコロナウイルス由来の3CL加水分解酵素(Mpro)のアミノ酸配列を、PDBアーカイブのエントリーと比較すると、 少なくとも90%の配列相同性を持つ蛋白質が95件のPDBエントリーで同定されました。 さらに、これらの関連蛋白質構造には、約30個の異なる低分子阻害剤が含まれており、新薬の発見に役立つ可能性があります。 2019-nCoVコロナウイルスの3CL加水分解酵素(Mpro)とSARSウイルスのメインプロテアーゼ(PDB 1q2w)の間で、アミノ酸配列の相同性が96%という 非常によく一致しているのは、創薬において特に重要です。 これら密接に関連するPDB構造についての要約データが利用でき、 (CSVファイル) 、容易にこの情報を見つけることができます。 さらに、PDBには、2番目のウイルスプロテアーゼ、RNAポリメラーゼ、ウイルススパイク蛋白質、ウイルスRNA、その他の蛋白質など、 200を超えるPDBエントリー中に20を超える固有のSARS蛋白質の3次元構造データが保管されています (CSVファイル)。
2019-nCoVコロナウイルス由来の3CL加水分解酵素(Mpro)の公開により、この情報が最も重要で価値があると証明できる時期に、 科学データがオープンでタイムリーに利用できることの重要性が強調されます。 wwPDBは、可能な限り包括的かつ正確なアーカイブを維持する一方で、高分子の構造データを誰もが自由に利用できるように努めています。 この新しい構造と関連ウイルスの構造により、研究者や臨床医が2019-nCoVコロナウイルスによる世界規模の緊急事態へ対処するのに役立つことを願っています。
[ wwPDB News ]
日本で解析された最初のCOVID-19関連エントリーが公開されました
|
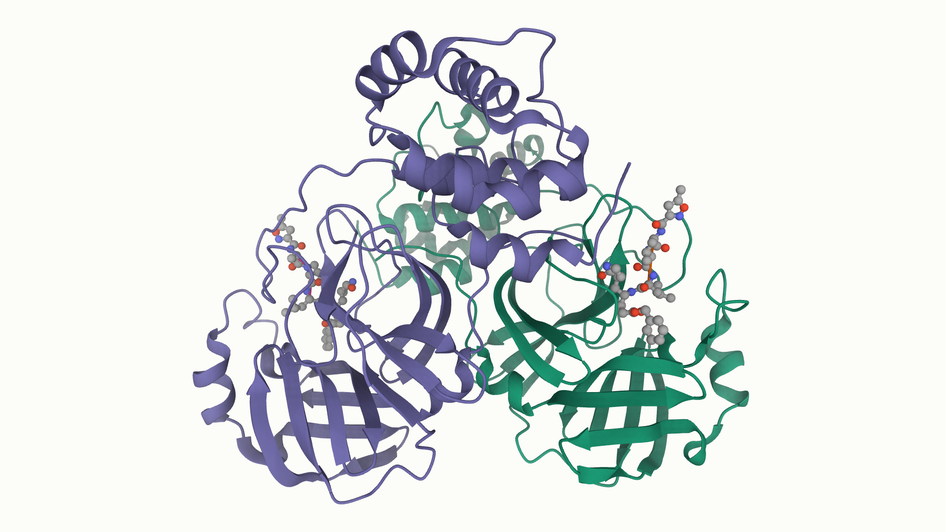
PDBデータが医薬品開発を加速することは広く知られており,現在猛威をふるう新型コロナウイルス感染症(COVID-19)に対しても, 世界中で積極的な研究が進められています。 PDBは地域分担で運営されているので,アジア発の蛋白質構造は全て大阪大学にあるPDBjで編集された後に世界共通のPDBアーカイブへ登録されます。 COVID-19に関連する最初の構造は,中国・上海科技大学のRao Zihe氏とYang Haitao氏が率いる共同グループが解析したメインプロテアーゼの構造でした(PDB ID: 6lu7)。 その後,沢山のCOVID-19関連構造が決定されてきましたが,この度,日本で最初のCOVID-19関連構造が大阪大学蛋白質研究所の高木淳一教授のグループからデータ登録され,論文発表を待たずに公開されました(右図,PDB ID: 7dmu)。 新型コロナウイルス(SARS-CoV-2)がヒトに感染するとき,スパイク蛋白質と呼ばれる表面に突き出た突起部分がACE2と呼ばれる受容体を認識します 。 高木教授のグループが着目したのはスパイク蛋白質の受容体結合部位(RBD: receptor binding domain)で,グループで開発したRBD親和性の向上したACE2受容体改変体(オレンジ色)が,スパイク蛋白質のRBD(緑色)に結合した構造を決定しました。SARS-CoV-2のヒトへの感染をブロックする中和抗体に代わるアプローチとして注目されます。 |

|
[wwPDB] PDBの50年
PDBは1971年10月20日に、 Crystallography: Protein Data Bank Nature New Biology 233: 223 (1971) doi: 10.1038/newbio233223b0において発表されました。
50年後の今日、PDBアーカイブには17万件を超える蛋白質や核酸、複合体の構造が登録されており、学生や研究者がタンパク質合成から健康や疾病まで、生体臨床医学と農業のあらゆる側面を理解する手助けとなっています。 国際蛋白質構造データバンク(wwPDB, Worldwide PDB)組織が、PDBを世界中の人に自由に公開できるよう運営管理しています。
PDBアーカイブ50周年を記念した新しいカレンダーは、自由にダウンロード頂けます。各月の画像は、バーチャルミーティングの背景としてもご利用頂けます。
wwPDBは2021年を通してシンポジウムやイベントを行い、この50周年記念を祝う予定です。
5月4日、5日に開催されるバーチャルシンポジウムの参加申し込みは、今月後半に開始する予定です。 イベントでは、構造生物学とバイオインフォマティクスで驚異的な発展を進めた世界中の講演者からのプレゼンテーションも予定されています。 学生や研究者は特に参加することが奨励されており、ポスター賞の対象となります。
PDB 50周年を迎えるにあたり、PDBの公共性、協調、教育の精神を支援して、wwPDB財団,もしくはPDBj支援基金へのご寄付もご検討ください。
wwPDB財団はアウトリーチ活動を支援する目的で、2010年に米国に設立されPDB40周年を祝うシンポジウムPDB40やワークショップ、教育出版物を支援した実績があります。
wwPDB財団は、米国の501(c)(3)団体として設立を認められています。
[ wwPDB News ]
[wwPDB] 糖鎖分子の表現の改善について(英語)
An archival-level carbohydrate remediation project that led to the re-release of over 14,000 PDB structures in July 2020. This update includes diverse oligosaccharides: glycosylation; metabolites such as maltose, sucrose, cellulose fragments; glycosaminoglycans, such as fragments of heparin and heparan sulfate; epitope patterns such as A/B blood group antigens and the H-type or Lewis-type stems; and many artificial carbohydrates mimicking or countering natural products (see documentation).
Starting in 2017, this PDB remediation aimed to standardize the biochemical nomenclature of the carbohydrate components following the IUPAC-IUBMB recommendations established by the carbohydrate community (PDF), and to provide uniform representation of oligosaccharides to improve the identification and searchability of oligosaccharides modeled in the PDB structures. During the remediation planning, wwPDB consulted community users and the PDBx/mmCIF Working Group and made data files available on GitHub in early 2020 for community feedback. wwPDB has collaborated with Robert Woods at University of Georgia in US, researchers at The Noguchi Institute and Soka University in Japan, and Thomas Lutteke in Germany to generate uniform linear descriptors for the oligosaccharide sequences.
To achieve these community goals, each oligosaccharide is represented as a branched entity with complete biochemical description and each glycosidic linkage specified. The full representation of carbohydrates is provided in the mmCIF format file, but this is not possible in legacy PDB format files (as the format has been frozen since 2012).
Proper indexing is necessary for branched entity representation and for generation of linear descriptors, hence the ordering (numbering) starts at the reducing end (#1), where the glycosylation occurs, to the non-reducing end in ascending order. Unique chain IDs are assigned to branched entities (oligosaccharides) to avoid residue numbering overlapped with protein residues and to enable consistent numbering for every oligosaccharide. For example, in PDB ID 6WPS, there are 5 oligosaccharides associated with the same protein chain A, the consistent ordering and numbering can only be retained with unique chain ID for each oligosaccharide in both PDBx/mmCIF and PDB format files
For archival consistency, a single-monosaccharide is defined as a non-polymer and treated consistently with other non-polymer ligands in the PDB. A single-monosaccharide occurring at a glycosylation site has a unique chain ID in the PDBx/mmCIF file (_atom_site.label_asym_id) but not in the PDB format file.
Using PDB ID 6WPS as an example, the PDBx/mmCIF data item _atom_site.label_asym_id corresponds to the column #7 in the atom_site coordinates section has an asym ID ‘Y’ for the 1st instance of single-monosaccharide, NAG bound to ASN 61 of protein chain ‘A’. The ‘Y’ value is unique for this monosaccharide. The additional chain ID (_atom_site.auth_asym_id) in the PDBx/mmCIF file that mapped to the PDB format file for this NAG is chain ‘A’, which is consistently represented as any other non-polymer ligands associated with the protein chain A.
#
loop_
_atom_site.group_PDB
_atom_site.id
_atom_site.type_symbol
_atom_site.label_atom_id
_atom_site.label_alt_id
_atom_site.label_comp_id
_atom_site.label_asym_id
_atom_site.label_entity_id
_atom_site.label_seq_id
_atom_site.pdbx_PDB_ins_code
_atom_site.Cartn_x
_atom_site.Cartn_y
_atom_site.Cartn_z
_atom_site.occupancy
_atom_site.B_iso_or_equiv
_atom_site.pdbx_formal_charge
_atom_site.auth_seq_id
_atom_site.auth_comp_id
_atom_site.auth_asym_id
_atom_site.auth_atom_id
_atom_site.pdbx_PDB_model_num
...
HETATM 27655 C C1 . NAG Y 6 . ? 191.103 162.375 206.665 1.00 47.28 ? 1301 NAG A C1 1
HETATM 27656 C C2 . NAG Y 6 . ? 191.067 161.665 208.065 1.00 47.22 ? 1301 NAG A C2 1
HETATM 27657 C C3 . NAG Y 6 . ? 190.138 160.434 207.960 1.00 47.42 ? 1301 NAG A C3 1
HETATM 27658 C C4 . NAG Y 6 . ? 188.730 160.906 207.541 1.00 48.73 ? 1301 NAG A C4 1
HETATM 27659 C C5 . NAG Y 6 . ? 188.838 161.622 206.176 1.00 48.66 ? 1301 NAG A C5 1
HETATM 27660 C C6 . NAG Y 6 . ? 187.494 162.153 205.709 1.00 48.17 ? 1301 NAG A C6 1
HETATM 27661 C C7 . NAG Y 6 . ? 193.233 161.885 209.217 1.00 47.40 ? 1301 NAG A C7 1
HETATM 27662 C C8 . NAG Y 6 . ? 194.594 161.311 209.471 1.00 47.45 ? 1301 NAG A C8 1
HETATM 27663 N N2 . NAG Y 6 . ? 192.418 161.218 208.414 1.00 47.36 ? 1301 NAG A N2 1
HETATM 27664 O O3 . NAG Y 6 . ? 190.069 159.774 209.231 1.00 47.22 ? 1301 NAG A O3 1
HETATM 27665 O O4 . NAG Y 6 . ? 187.867 159.778 207.435 1.00 48.89 ? 1301 NAG A O4 1
HETATM 27666 O O5 . NAG Y 6 . ? 189.760 162.757 206.285 1.00 47.83 ? 1301 NAG A O5 1
HETATM 27667 O O6 . NAG Y 6 . ? 186.953 163.102 206.622 1.00 49.06 ? 1301 NAG A O6 1
HETATM 27668 O O7 . NAG Y 6 . ? 192.879 162.950 209.739 1.00 47.58 ? 1301 NAG A O7 1
...
Author-provided chain ID and residue numbering for oligosaccharides are retained in the PDBx/mmCIF file (_pdbx_branch_scheme.auth_mon_id and _pdbx_branch_scheme.auth_seq_num, respectively). Users can map how carbohydrates are described in the corresponding primary citation to the PDBx/mmCIF files using _pdbx_branch_scheme mapping category. wwPDB strongly encourages depositors to use the wwPDB-assigned chain ID and residue numbers in any publication material.
For example, in PDB entry 6WPS
loop_
_pdbx_branch_scheme.asym_id
_pdbx_branch_scheme.entity_id
_pdbx_branch_scheme.mon_id
_pdbx_branch_scheme.num
_pdbx_branch_scheme.pdb_asym_id
_pdbx_branch_scheme.pdb_mon_id
_pdbx_branch_scheme.pdb_seq_num
_pdbx_branch_scheme.auth_asym_id
_pdbx_branch_scheme.auth_mon_id
_pdbx_branch_scheme.auth_seq_num
_pdbx_branch_scheme.hetero
J 4 NAG 1 I NAG 1 A NAG 1310 n
J 4 NAG 2 I NAG 2 A NAG 1311 n
K 4 NAG 1 J NAG 1 A NAG 1312 n
K 4 NAG 2 J NAG 2 A NAG 1313 n
L 4 NAG 1 K NAG 1 A NAG 1315 n
L 4 NAG 2 K NAG 2 A NAG 1316 n
M 4 NAG 1 M NAG 1 A NAG 1317 n
M 4 NAG 2 M NAG 2 A NAG 1318 n
N 5 NAG 1 N NAG 1 A NAG 1321 n
N 5 NAG 2 N NAG 2 A NAG 1322 n
N 5 BMA 3 N BMA 3 A BMA 1323 n
N 5 MAN 4 N MAN 4 A MAN 1325 n
N 5 MAN 5 N MAN 5 A MAN 1324 n
N 5 FUC 6 N FUC 6 A FUC 1320 n
O 4 NAG 1 O NAG 1 B NAG 1310 n
O 4 NAG 2 O NAG 2 B NAG 1311 n
P 4 NAG 1 P NAG 1 B NAG 1312 n
P 4 NAG 2 P NAG 2 B NAG 1313 n
Q 4 NAG 1 Q NAG 1 B NAG 1315 n
Q 4 NAG 2 Q NAG 2 B NAG 1316 n
R 4 NAG 1 R NAG 1 B NAG 1317 n
R 4 NAG 2 R NAG 2 B NAG 1318 n
S 5 NAG 1 S NAG 1 B NAG 1321 n
S 5 NAG 2 S NAG 2 B NAG 1322 n
S 5 BMA 3 S BMA 3 B BMA 1323 n
S 5 MAN 4 S MAN 4 B MAN 1325 n
S 5 MAN 5 S MAN 5 B MAN 1324 n
S 5 FUC 6 S FUC 6 B FUC 1320 n
...
As some users pointed out, single NAG could be just a part of the glycan that the author chose to build, as most natural N-glycans must have stem of a common core of 5 monosaccharides or its fucosylated version, such as those modeled in the PDB ID 6WPS. However, the PDB is a 3D-atomic coordinate archive in which the model coordinates are built based on supporting experimental data. Therefore, carbohydrates are described as-is in the modeled structures without reference to missing components of the presumed oligosaccharide sequence. If the author only builds a monosaccharide, then this monosaccharide is described as a non-polymer ligand.
Glycosylation annotation has been provided to facilitate searches of all glycosylation sites. A total of 45,000 glycosylation sites have been annotated in _struct_conn.pdbx_role in over 7500 PDB structures to identify all glycosylation sites and the monosaccharides bound at such sites. The annotation specifies the glycosylation sites, the monosaccharide identity and chain IDs in either PDB format or mmCIF format. In PDB ID 6WPS, a user can search N-Glycosylation in ‘_struct_conn.pdbx_role’ and find 16 glycosylation sites between ASN and NAG at chain A alone.
In addition, a total of 1040 carbohydrate ligands were reviewed and their nomenclature has been standardized in the PDB to follow the 1996 IUPAC recommendations. The updates of monosaccharides in the Chemical Component Dictionary include chemical names, synonyms, atom labels, modification vs common sugars tags, chemical types (e.g., isomers), structure feature types (e.g., anomers), and symbol identifiers (e.g., IUPAC condensed symbols). The chemical names provided in PDBx/mmCIF data item _chem_comp.name have been updated uniformly to include stereo- and ring- specific systematic names as described in the IUPAC recommendations. Trivial or common names are annotated in the new PDBx/mmCIF category, _pdbx_chem_comp_synonyms, with one name per row. Both the IUPAC extended-form symbol as described in the section 2-Carb-38.3 of the 1996 recommendation and the condensed-form symbol in 2-Carb-38.4 are provided in the identifier of an PDBx/mmCIF data item _pdbx_chem_comp_identifier.identifier. Carbohydrate features such as isomer, ring size, anomer, and aldose/ketose classification as described in the PDBx/mmCIF category _pdbx_chem_comp_feature.
The wwPDB encourages the community to use PDB/mmCIF format files rather than the frozen legacy PDB file format. The legacy format cannot support large structures. Currently, PDB format-files are not available for large structures that have either more than 62 chains or 99,999 atoms. In addition, the legacy format cannot support ligand ID codes beyond 3-characters, which will be needed in the coming years.
The wwPDB is committed to improving data representation in the PDB archive. Please do not hesitate to contact us at info@wwpdb.org.
[ wwPDB News ]
1月20日(水)、BINDS-PDBj講習会・蛋白研セミナー「データベース登録ノウハウ講習会」を開催します
- 開催趣旨
- 科学研究で得られたデータを、公開データベースに登録することは、科学の健全な発展に大変重要です。 構造生物学のデータの登録・配布については、PDBをはじめとする様々なデータベースが整備されています。 しかし、近年、登録に必要なデータが複雑化してきたため、初心者にはわかりにくくなってきていることも確かです。 そこで、本講習会では、データ登録作業を円滑に行っていただくためのコツやノウハウを、登録経験が豊富な方やデータベースの開発・管理を 行っている方を講師に招き、説明していただきます。
- 主催
- 日本蛋白質構造データバンクPDBj、創薬等先端技術支援基盤プラットフォーム(BINDS)、大阪大学蛋白質研究所
- 日時
- 2021年 1月20日(水)14:00-17:30
- 開催方法
- ZOOMによるウェビナー形式での開催
- 定員
- 100名程度 (先着順)
- 参加費
- 無料 (事前登録必要)
- 参加申込
- 参加申込フォームよりお申込みください。(申込期限:1/13(水) 17:00まで)
参加申込後にリモート会議のアドレスをメールで送付いたします。 - お問い合わせ
- PDBjのお問い合わせページ をご利用ください。
<プログラム>
| 14:00-14:05 | 「開会の挨拶」(大阪大学 蛋白質研究所) | ||
| 14:05-14:30 | 「最近のPDBの動向」栗栖 源嗣 (大阪大学 蛋白質研究所) | |
|
| 14:30 – 15:00 | 「X線結晶構造の登録」白井 剛 (長浜バイオ大学) | [講義資料] | |
| 15:00 – 15:30 | 「NMR構造・データの登録」横地 政志 (大阪大学 蛋白質研究所) | [講義資料] | |
| 休憩(15分) | |||
| 15:45 – 16:00 | 「X線画像アーカイブ XRDa」栗栖 源嗣 (大阪大学 蛋白質研究所) | [講義資料] | |
| 16:00 – 16:15 | 「モデル構造アーカイブ BSM-Arc」 Gert-jan Bekker (大阪大学 蛋白質研究所) | [講義資料] | |
| 16:15 – 16:30 | 「電顕2D画像アーカイブ EMPIAR」 川端 猛 (大阪大学 蛋白質研究所) | [講義資料] | |
| 16:30-17:00 | 「電子顕微鏡の構造・マップの登録」山下 恵太郎 (MRC分子生物学研究所) | [講義資料] | |
| 17:00 – 17:25 | 全体を通じた質問 | ||
| 17:25 – 17:30 | 「閉会の挨拶」栗栖 源嗣 (大阪大学 蛋白質研究所) |
1月18日(月)・19日(火)、CiCLE講習会・蛋白研セミナー「単粒子解析リモート講習会」を開催します
- 開催趣旨
- 近年のクライオ電子顕微鏡の単粒子解析の進展は著しく、X線結晶解析に迫る分解能の生体高分子像が得られるようになってきています。 本リモート講習会では、電顕の初心者の方を対象に、電子顕微鏡の原理と実験手続き、単粒子解析の原理、画像処理、三次元再構成、 原子モデリングについての講義と講習を行います。遠隔による講義のため、本格的な実習はできませんが、動画や実データの配布を行うことで、できるだけ体感的な講習を行います。
- 主催
- 医療研究開発革新基盤創成事業(CiCLE)「タンパク質構造解析のハイスループット化へ向けた装置開発」、大阪大学蛋白質研究所
- 日程
- 2021年 1月18日(月)、19日(火)
- 開催方法
- ZOOMによるウェビナー形式での開催
- 定員
- 100名程度 (先着順)
- 参加費
- 無料 (事前登録必要)
- 参加申込
- 参加申込フォームよりお申込みください。(申込期限:1/12(火) 17:00まで)
参加申込後にリモート会議のアドレスをメールで送付いたします。 - ソフトウエアの事前インストールのお願い
あらかじめ、以下のソフトをお使いのPCに インストールしておいてください。
1) Comment Screen (1/18の講義中に使用します)
2) UCSF Chimera (1/19の計算機演習で使用します)
3) Coot (1/19の計算機演習で使用します)
*インストール手続きは以下にご案内します。- Comment Screenのインストール方法
- https://commentscreen.com/#downloadからWindows版、もしくはMacintosh版を ダウンロードしてインストールを行ってください。
- UCSF Chimeraのインストール方法
- https://www.cgl.ucsf.edu/chimera/download.htmlのサイトから、ダウンロードしインストールを行ってください。 Windows, MacOS X, Linux版があります。最新版は1.15です。非商用目的での使用のみ無料で用いることができます。 Windows版のダウンロード手続き、および Mac版のダウンロード手続きを参考にしてください。
- Cootのインストール方法
-
0.8.9か0.9.2の版をインストールしてください。いくつかのインストール法があります。
- Windowsの場合
- WinCootのページ(https://bernhardcl.github.io/coot/wincoot-download.html) からダウンロードして、インストールできます。 [Download WinCoot 0.8.9]をクリックすると、0.9.2の版がインストールできます。
- https://www2.mrc-lmb.cam.ac.uk/personal/pemsley/cootのページ からCoot 0.8.9のWindows版をダウンロードできます。
- CCP4のページ(http://www.ccp4.ac.uk/download/#os=windows)から、 CCP4をインストールすると、Coot 0.8.9もいっしょにインストールされます。
- MacOS Xの場合 http://www.ccp4.ac.uk/download/#os=macのサイトからCCP4をインストールすると、 Coot 0.8.9も同時にインストールされます。
- 講義・講習の担当
- Christoph Gerle(大阪大学 蛋白質研究所)、牧野 文信(日本電子)、加藤 貴之(大阪大学 蛋白質研究所)、
川端 猛(大阪大学 蛋白質研究所)、田中 秀明(大阪大学 蛋白質研究所) - お問い合わせ
- PDBjのお問い合わせページ をご利用ください。
<スケジュール>
2021年1月18日(月)| 13:00-13:05 | 「開会の挨拶」中川 敦史 | |
| 13:05-15:00 | 「低温電子顕微鏡入門」Christoph Gerle | [講義資料] |
| 15:10 – 16:00 | 「JEOLの低温電子顕微鏡の紹介」牧野 文信 | [講義資料] |
| 16:10 – 18:00 | 「単粒子解析の原理」加藤 貴之 |
| 13:00-15:00 | 「Relionによる電顕画像処理」川端 猛、加藤 貴之 | [講習資料] | 講習会の様子 [YouTube] |
|
講習会用電顕画像データ: EMPIAR-10291_50mic.tar
パソコン用の講習会データ |
 講習ビデオ:Innexin-6を題材にしたRelion 3.1の講習 -- 画像の前処理 -- [YouTube] |
||
| 15:10 – 16:00 | 「Relionによる三次元再構成」川端 猛、加藤 貴之 | [講習資料] | 講習会の様子 [YouTube] |
|
[補足資料:Relionの背景にあるベイズ理論] [補足資料:「UCSF Chimeraによる原子モデルのフィッティング」 |
 講習ビデオ:Innexin-6を題材としたRelion 3.1の講習 : 2D分類と3D分類 [YouTube] |
||
| 16:10 – 17:55 | 「Cootによる原子モデリング」田中 秀明 | [講習資料] | 講習会の様子 [YouTube] |
|
モデリング演習用のマップ・原子モデルデータ [モデリング用データ(EM_koushuukai.zip)(53 MB)] [モデリング用データ(EM_koushuukai.tar.gz)(53 MB)] |
講習ビデオ:Cootによる原子モデリング [YouTube] |
||
PDBjで処理したPDBエントリー数が40,000件に到達
PDBjでは2000年7月に大阪でのデータ処理を開始し,地域分担して処理したPDBエントリー数が累計で40,000件に到達しました(wwPDBサイトの統計ページ)。 wwPDB全体で約17万件のエントリー数ですので,全体の約24%をPDBjでデータ登録処理した計算になります。 2000年から継続して援助していただいているJSTに深く感謝いたします。
PDBj 20周年の記事については、ニュースレター(Vol.21 No.2)もご覧ください。
11月22日(日)、サイエンスアゴラ2020にてオンラインライブイベントを開催します
11月22日(日)、サイエンスアゴラ2020にてオンラインライブイベントを開催します。 視聴には概要ページ左下「このセッションに参加する(事前登録)」から事前参加登録が必要です(参加料無料)。ご参加お待ちしています。
- 日時
- 2020年11月22日(日)12:30-13:20
- タイトル
- ようこそ、タンパク質ワンダーランドへ!
- 出展者
- 大阪大学蛋白質研究所・PDBj
- 登壇者
- 栗栖 源嗣(大阪大学蛋白質研究所 教授、PDBj代表)
- 企画概要
- タンパク質と言えば栄養素の一種としてお肉や卵などを思い浮かべる人が多いかもしれません。 しかし、血液、だ液、ホルモン、抗体、分解酵素、光合成など私たちを含むあらゆる生物を動かしているものすべてにタンパク質が深く関わっています。 この身近で不思議な「タンパク質」のはたらきと形について解説します。対象は小学生高学年を想定しています。
- 講演資料
- こちらからダウンロードできます。
公開WEBシンポジウム「COVID-19パンデミックを契機として考える日本の結晶学の現状と今後」
公開WEBシンポジウム「COVID-19パンデミックを契機として考える日本の結晶学の現状と今後」にてPDBjからも講演します。ご参加お待ちしています。
- 日時
- 2020年11月29日(日)10:00~15:30
- 場所
- WebEXによるオンライン
- 対象
- どなたでもご参加いただけます
- 参加料金
- 無料
- 参加方法
- 登録ページから事前登録をお願いします。
- PDBjからの講演
-
- 時間
- 10:25~10:50
- 講演タイトル
- 新型コロナウイルス感染症と立体構造データベース-蛋白質科学の視点から
- 講演者
- 栗栖 源嗣(大阪大学蛋白質研究所 教授、PDBj代表)
[wwPDB] PDBは49周年を迎えます
PDBは1971年10月20日に、 Crystallography: Protein Data Bank Nature New Biology 233: 223 (1971) doi: 10.1038/newbio233223b0 において発表されました。
今日、PDBアーカイブには、約17万件の蛋白質や核酸、複合体の構造が登録されており、学生や研究者がタンパク質合成から健康や疾病まで、生体臨床医学と農業のあらゆる側面を理解する手助けとなっています。 国際蛋白質構造データバンク(wwPDB, Worldwide PDB)組織が、PDBを世界中の人に自由に公開できるよう運営管理しています。
wwPDBは、2021年に予定されている50周年記念シンポジウムとイベントを見据えており、PDB50周年のウェブサイトwwpdb.org/pdb50を定期的に更新する予定です。
PDB 50周年を迎えるにあたり、PDBの公共性、協調、教育の精神を支援して、wwPDB財団へのご寄付もご検討ください。
wwPDB財団は、wwPDBのアウトリーチ活動を支援する目的で、2010年に設立され、 PDB40周年を祝うシンポジウムPDB40やワークショップ、教育出版物を支援するために資金を集めました。
財団は、もっぱら科学、文学、慈善、教育的目的のためだけに501(c)(3)団体として設立を認められています。
[ wwPDB News ]
wwPDB基準で計算された電子密度マップ係数がPDBjの電子密度マップサービスからご利用できるようになりました
PDBjが以前EDMapサービスで提供していたX線構造の電子密度は、独自にCCP4を使って座標から計算した係数を使って提供していました。
2020年9月23日からは,wwPDBの検証レポートの計算でつかうBuster (Global Phasing社)
による係数で統一して提供しています。
詳しくは、過去のwwPDBニュースもご覧ください。
更新された電子密度マップ係数ファイルは検証レポートと同様、以下のPDBj FTPサイト(
ftp://ftp.pdbj.org/pub/pdb/validation_reports/)からご利用頂けます。
またPDBj Mineや電子密度マップサービスのウェブインターフェースからもそれらをダウンロードすることができます。
トーゴーの日シンポジウム2020にてPDBjからもポスター発表を行います
2020年10月5日(月)、オンラインで開催されるトーゴーの日シンポジウム2020にてPDBjからもポスター発表を行います。
[wwPDB] PDB-Devのウェブサイトを更新しました
PDB-Devウェブサイトを更新しました
新しく改善されたPDB-Devウェブサイトをご利用頂けるようになりました。 PDB-Devは、wwPDBが統合構造のアーカイブ要件を理解するために役立つプロトタイプシステムであり、 wwPDB 統合/ハイブリッド法専門委員会(wwPDB Integrative/Hybrid Methods (I/HM) Task Force)の提言に基づいて構築されました。 PDB-Devには現在、37件の生体高分子複合体の統合構造があります。
更新されたPDB-Devウェブインターフェイスは、動的で応答性の高いウェブページを提供し、2つの新機能が含まれています。
- PDB-Devにアーカイブされた構造の検索を容易にする新しい検索サービス。 現在の実装では、高分子名、エントリーの識別子、実験データ種別、著者名、引用文献、ソフトウェア、その他いくつかのキーワードによる検索をサポートしています。 ブール演算子とワイルドカードを使用した複雑なクエリをサポートしています。 検索結果はcsvまたはExcelファイルとしてダウンロードできます。
- 公開済みエントリーに対する個々のエントリーページ、強調表示した各構造の主要な詳細、ダウンロードリンク。
PDB-Devチームは、ユーザからのフィードバックを歓迎します。 コメントやご提案がございましたら、pdb-dev@mail.wwpdb.orgまで、英語にてお送りください。
[ wwPDB News ]
9/16~9/18に日本生物物理学会年会でPDBjオンライン展示会を開催します
PDBjは9/16(水)~9/18(金)にオンライン開催される第58回日本生物物理学会年会において オンライン展示会を開催いたします
日本蛋白質構造データバンク(PDBj: Protein Data Bank Japan)は、生体高分子の立体構造データのデータベースであり、 今年で設立20周年を迎えました。 米国RCSB PDBと欧州PDBeと共にwwPDBを組織し,アジア・中東地区からの構造データの登録を行っています。 本展示会では、20年を迎えたPDBjと、最近のPDBの動向に加え、 正式なファイルフォーマットとして採用されたmmCIFの概要、PDBjで開発された分子ビューアMolmilの使用法について、ご説明いたします。
学会の会期中に、以下のように説明資料と説明動画を公開いたしますので、お手すきの時間にご視聴ください。
| タイトル | 話者 | 説明資料 | 説明動画 |
| 20年を迎えたPDBj | 栗栖源嗣 | [資料] |  |
| PDBの最近の動向 | 栗栖源嗣 | [資料] |  |
| 分子ビューアMolmilの使い方 | 川端 猛 | [資料] |

|
| 旧PDBフォーマットとmmCIFフォーマット | 川端 猛 | [資料] |
|
また、学会の会期中の以下の時間に、学会参加者からのZOOMによる質問を受け付けます。 上記の資料・動画についての質問以外に、PDBのデータ・サービスの質問、データ登録、立体構造一般に関する質問にもできるだけ対応いたします。 ZOOMのアドレスは 学会の年会WEBページの オンライン展示会のページ でも公開しています。 皆様のお越しをお待ちしています。
| ZOOMによる質問受付時間 |
| 9月16日(水)12:00~13:30 : [ZOOMで接続] |
| 9月17日(木)12:00~13:30 : [ZOOMで接続] |
| 9月18日(金)12:00~13:30 : [ZOOMで接続] |
[wwPDB] 糖鎖分子の表現を改善したPDBデータを公開しました
PDBエントリー内の糖鎖に関する新たなデータ表現方法と、高分子構造中における糖鎖の検索性と相互運用性を向上させることを目的とした参照データをPDBデータに組み込みました。 PDBアーカイブにおける糖鎖表現を修正し改良するために、wwPDBでは以下のことを行いました。
- IUPAC-IUBMB勧告にしたがい化合物辞書の命名法を標準化
- オリゴ糖に対するばらつきのない表記法を提供
- 糖鎖科学のコミュニティのツールを使い、このコミュニティで一般的に使われている線形記述法を採用
- PDB構造における糖鎖修飾部位の説明を追加
このプロジェクトに関する詳細な情報はwwPDBのウェブサイトで確認できます。 2020年2月のお知らせ(wwPDB news/PDBj news)でも強調したように、PDBデータの生成、アクセス、視覚化を行うソフトウェアパッケージの開発者には、できるだけ早くこの情報を確認しソフトウェアに反映することを推奨します。
wwPDBでは多糖用として新たに「Branched」というエンティティ表現をつくり、多糖に含まれる個々の単糖要素すべてをPDBエントリーのデータに記述しました。この過程で化合物辞書(CCD)にある1000を超える単糖の原子命名法を標準化し、8000を超えるPDBエントリーにおいて分岐エンティティ表現をオリゴ糖に適用しました。影響を受けるPDBエントリーにおけるオリゴ糖の表記を明確なものにするため、オリゴ糖を構成する各単糖単位間の共有結合を明確に示した記述を含めています。さらに、wwPDB検証レポートでは、これらのオリゴ糖に関する一貫した表現と多糖記号命名法(SNFG)に基づく二次元表現図も記載しています。
糖鎖表現の修正を支援するため、線形記述法を提供するソフトウェアを糖鎖科学コミュニティと協力して開発し、PDBデータを糖鎖生物学者の間で一般的に用いられている別の表現法へと簡単に変換できるようにしました。 このツールには、米国ジョージア大学のGMMLからの短縮型IUPAC、日本の野口研究所のPDB2GlycanからのWURCS、ドイツのpdb-careからのLINUCSが含まれます。
wwPDBは、この機会を利用して、新しいデータカテゴリ_pdbx_chem_comp_synonymsを導入することにより、CCD内の化学的同義語の体系を改善しています。 これにより、PDB内の低分子の代替名称をより包括的にとらえることが可能になります。 ユーザーへの影響を最小限に抑えるために、移行期間をもうけ、2021年の間はレガシーデータ項目_chem_comp.pdbx_synonymsも保持されます。
糖鎖表現改良プロジェクトは、ニュージャージー州立ラトガース大学のRCSB PDBが主導して実施するwwPDBの共同プロジェクトであり、 ジョージア大学の複合糖質研究センターと共同でNIGMS助成金U01 CA221216から資金提供を受けています。
この変更に関するご質問などございましたら、wwPDBの糖鎖表現の改善に関するウェブページ をご覧頂くか、deposit-help@mail.wwpdb.orgまで、英語でお問合せください。
[ wwPDB News ]
PDBj FTPサイトから、PDBj Mineの旧RDBデータファイル群の提供を終了します
PDBjは2020年7月に20周年を迎えました

日本蛋白質構造データバンク(PDBj: Protein Data Bank Japan)は2020年7月で設立20周年を迎えました。JST-BIRD(当時)の支援を受けて最初のPDBエントリーの登録処理を行ったのが2000年7月6日です。2000年には,わずか157件であったPDBjでのデータ登録処理数は,RCSB PDBの協力も得て軌道に乗り,2001年には376件,2002年には602件の登録を行いました。2003年にRCSB PDBとPDBeと共にwwPDBを組織し,アジア・オセアニア地区の全データ登録に責任を持つまでに成長しました。その後,中国からのデータ登録数が急増したことにより,2016年からは責任地域をアジア・中東地区に修正して現在に至っています。この間,eF-siteやProModeなどの2次データベース構築や,PDBMLplus,RDFなど新規フォーマットの導入,MolmilやMagROなど関連ツール類の開発を進めてまいりました。これは一重に,登録者,利用者の皆様やJSTやAMEDをはじめとする関係機関による継続的なご支援の賜物です。PDBjは,これからも引き続き蛋白質構造データバンクのアジア・中東地区拠点として頑張ってまいりますので,変わらぬご支援を賜りますようお願い申し上げます。
[wwPDB] 7月29日に糖鎖分子の表現を改善したPDBデータを公開します
PDBエントリー内の糖鎖に関する新たなデータ表現方法と、高分子構造中における糖鎖の検索性と相互運用性を向上させることを目的とした参照データをPDBデータに組み込みます。PDBアーカイブにおける糖鎖表現を修正し改良するために、wwPDBでは以下のことを行いました。
- IUPAC-IUBMB勧告にしたがい化合物辞書の命名法を標準化
- オリゴ糖に対するばらつきのない表記法を提供
- 糖鎖科学のコミュニティのツールを使い、このコミュニティで一般的に使われている線形記述法を採用
- PDB構造における糖鎖修飾部位の説明を追加
改良されたデータには2020年7月29日よりFTPやwwPDBメンバーのウェブサイトからアクセスできます。 2020年2月のお知らせ(wwPDB news/PDBj news)でも強調したように、PDBデータの生成、アクセス、視覚化を行うソフトウェアパッケージの開発者には、できるだけ早くこの情報を確認しソフトウェアに反映することを推奨します。 このプロジェクトに関する詳細な情報はwwPDBのウェブサイトで確認できます。 影響を受けるエントリーと化合物の一覧は、データを公開する7月29日以降先述のページにて掲載します。
wwPDBでは多糖用として新たに「Branched」(分岐した)というエンティティ表現をつくり、多糖に含まれる個々の単糖要素すべてをPDBエントリーのデータに記述しました。この過程で化合物辞書(CCD)にある1000を超える単糖の原子命名法を標準化し、8000を超えるPDBエントリーにおいて分岐エンティティ表現をオリゴ糖に適用しました。影響を受けるPDBエントリーにおけるオリゴ糖の表記を明確なものにするため、オリゴ糖を構成する各単糖単位間の共有結合を明確に示した記述を含めています。さらに、wwPDB検証レポートでは、これらのオリゴ糖に関する一貫した表現と多糖記号命名法(SNFG)に基づく二次元表現図も記載しています。
糖鎖表現の修正を支援するため、線形記述法を提供するソフトウェアを糖鎖科学コミュニティと協力して開発し、PDBデータを糖鎖生物学者の間で一般的に用いられている別の表現法へと簡単に変換できるようにしました。 このツールには、米国ジョージア大学のGMMLからの短縮型IUPAC、日本の野口研究所のPDB2GlycanからのWURCS、ドイツのpdb-careからのLINUCSが含まれます。
さらに、一般的に用いられているオリゴ糖(例:sucrose(ショ糖), Lewis Y antigen(ルイスY抗原))118種について引き続きその名称で検索できるようにするため、BIRD(Biologically Interesting molecule Reference Dictionary:ペプチド様抗生物質・阻害剤分子の辞書)に共有結合情報と一般的な別名を追加しました。
wwPDBは、この機会を利用して、新しいデータカテゴリ_pdbx_chem_comp_synonymsを導入することにより、CCD内の化学的同義語の体系を改善しています。 これにより、PDB内の低分子の代替名称をより包括的にとらえることが可能になります。 ユーザーへの影響を最小限に抑えるために、移行期間をもうけ、2021年の間はレガシーデータ項目_chem_comp.pdbx_synonymsも保持されます。
糖鎖表現改良プロジェクトは、ニュージャージー州立ラトガース大学のRCSB PDBが主導して実施するwwPDBの共同プロジェクトであり、 ジョージア大学の複合糖質研究センターと共同でNIGMS助成金U01 CA221216から資金提供を受けています。
この変更に関するご質問などございましたら、wwPDBの糖鎖表現の改善に関するウェブページ をご覧頂くか、deposit-help@mail.wwpdb.orgまで、英語でお問合せください。
[ wwPDB News ]
令和2年度科学技術分野の文部科学大臣表彰科学技術賞(振興部門)の受賞
大阪大学蛋白質研究所では、2000年からPDBjの活動を開始し、欧米と協調して国際的蛋白質構造データバンクを推進し、基礎から応用まで幅広い研究分野に対して蛋白質構造データを国内から発信・供与してまいりました。その業績「国際的蛋白質構造データバンク構築への貢献と統合利用の振興」に対し、2020年4月7日付にて、PDBjの前統括責任者の中村 春木(国立大学法人大阪大学名誉教授)と現統括責任者の栗栖 源嗣(国立大学法人大阪大学蛋白質研究所教授)が、令和2年度科学技術分野の文部科学大臣表彰科学技術賞(振興部門)を受賞することが発表されました。
[wwPDB] EMDBのマップエントリー公開過程の合理化について
2020年4月15日から、電子顕微鏡データバンク(EMDB)は、一次マップと関連ファイルの公開の前にエントリーのメタデータ (いわゆるXMLヘッダファイル)を公開する手続きを終了します。
EMDBマップの新しい登録に対しては、マップが公開されるまで、メタデータはEMDBのFTPアーカイブに公開されません。 すでにメタデータが公開済みの未公開エントリーについては、マップが公開されるまで、状態に変更はありません。 EMDBは、登録者に、できるだけマップを早く公開するよう求めます。
EMDBヘッダファイルの事前公開を廃止することは、EMDBとwwPDBのポリシーと処理手順に合致させる過程の一環です。 EMDBポリシーの詳細については、EMDBサイトの文書をご覧ください。
[ wwPDB News ]
NHKと毎日放送で、PDBjの活動が紹介されました
NHK
”新型コロナ” データベース化 (3/17放送)
毎日放送
『タンパク質』のデータバンクが"研究成果"を先駆け公開...新型コロナ治療薬開発には『タンパク質』がカギ!
(3/17放送)
新型コロナウイルスの特集ページを公開しました
PDBjでは,猛威をふるう新型コロナウイルス感染症(COVID-19)に直接関連する構造情報をまとめて公開しました。 新型コロナウイルス(SevereAcute Respiratory Syndrome Coronavirus 2: SARS-CoV-2)がもつ蛋白質の情報を、 何処よりも早く正確に公開します。
【COVID-19特集ページ】
https://pdbj.org/featured/covid-19
【今月の分子2020年2月:コロナウイルスプロテーアーゼ】
https://numon.pdbj.org/mom/242
[wwPDB] 2020年に、PDBデータ中の糖鎖分子の表現を改良します
2020年7月に、wwPDBは糖鎖分子の標準化された表現を使って、PDB構造ファイルと参照データファイルを更新し、PDBデータの発見可能性(Findability)と相互運用可能性(Interoperability)を改善します。 この改善作業に関する詳細情報とPDBx/mmCIF辞書の拡張情報、500件以上のサンプルファイルは、 wwPDBサイトからご利用いただけます 。 PDBデータを作成・アクセスしたり、または視覚化するソフトウェアパッケージの開発者は、変更情報を確認して、必要に応じてソフトウェアを更新することをお勧めします。
糖質科学のコミュニティと協力して、化合物辞書(CCD)内の800近い単糖類の原子命名法を標準化するソフトウェアツールを開発しました。 このツールは、PDBアーカイブ内のオリゴ糖や多糖を、分岐ポリマー表現(branched polymeric representation)で表すことができるほか、糖鎖生物学でよく用いられる他の分子表現に変換することもできます。 影響を受けるPDBエントリーおよそ12,000件のそれぞれにおいて、オリゴ糖/多糖の明確な化学的記述を保証するために、単量体単位間の共有結合情報の明示的な記述を含めました。 一般的なオリゴ糖(例えば、ショ糖、ルイスX因子)を継続的に確実に発見するために、 BIRD(Biologically Interesting molecule Reference Dictionary; ペプチド様 の抗生物質・阻害剤分子の辞書)を拡張しました。 これには、共有結合情報や分子の一般的な別名が含められます。
wwPDBは、この機会を利用して、新しいデータカテゴリ_pdbx_chem_comp_synonymsを導入することにより、CCD内の化学的同義語の体系を改善しています。 これにより、PDB内の低分子の代替名称をより包括的にとらえることが可能になります。 ユーザーへの影響を最小限に抑えるために、最初の移行期間をもうけ、その間はレガシーデータ項目_chem_comp.pdbx_synonymsも保持されます。
.png) 上図の構造は: 1b5f
上図の構造は: 1b5f
[ wwPDB News ]
[wwPDB] PDB IDを変更せずに、公開済みのPDBエントリーの座標を改善できます(第2段階)
2020年2月18日から、全ての公開済みのPDBエントリーの登録者(PI)は、同じPDBアクセッションコードを保持したままモデル座標を更新でき、そのPDB IDの論文とのリンクを維持できるようになります。 バージョニングプロジェクトの最終段階では、現在のwwPDB OneDepシステムが開始される前に登録された構造についても、バージョン管理機能を拡張しました。
OneDepで登録されたエントリーについては、登録者はまずOneDep登録サイトにログインし、コミュニケーションパネルから、変更したい旨を連絡をする必要があります。 OneDep以前の古いシステムから登録されたエントリーについては、最初に deposit-help@mail.wwpdb.org宛のメールで希望を連絡し、件名と本文にPDB IDを記載する必要があります。 新しい座標の登録後、wwPDBのバイオキュレータが再処理を行い、登録者の確認後、直ちに新しいバージョンが公開されます。 PDBエントリーのバージョニングは、座標等の変更に限定され、登録済みの実験データ(構造因子)の差替え・変更は行えません。 座標の変更は、年間に1エントリー当たり1回まで、一人の研究責任者(PI)に対して3件のエントリーまでに制限しています。 通常の登録作業とのバランスを検証した後、この制限は2021年に見直す予定です。
各エントリーの最新のバージョンは、メインPDBアーカイブのFTPサイト(ftp.pdbj.org [PDBj]/ ftp.wwpdb.org [wwPDB])で公開されます。全てのメジャーバージョンのPDB構造ファイルは、バージョン化アーカイブ(ftp-versioned.pdbj.org [PDBj]/ ftp-versioned.wwpdb.org [wwPDB])に保管されます。詳細は、wwPDBウェブサイトをご覧ください。バージョン化アーカイブの構造は、将来のPDBアクセションコードの拡張を想定したものとなっています。そのためPDBエントリー1abcのファイルは、pdb_00001abcというフォルダ内に配置されています。
バージョニング管理の中でエントリーに行われる変更は、「メジャー」又は「マイナー」のどちらかに分類されます。 原子座標やポリマー配列、化合物等の化学的な表現に更新があると、メジャーバージョンが一つ増えることになります。他の変更は「マイナー」バージョンの更新になります。バージョン化の中で行われた変更はすべて、PDBx/mmCIFファイルのauditカテゴリに記録されます。
PDBバージョニングについて何かご質問がございましたら、wwPDB(deposit-help@mail.wwpdb.org)まで、英語でお問い合わせください。
[ wwPDB News ]
新しいEDMapサービスを開始しました
新エントリーに対しては、wwPDBから提供されているマップ係数 (2fo-fc と fo-fc)の両方、またはどちらかを選べるようになっています。 一方、既存のエントリーに対しては、PDBjが作成した(2fo-fc)のオリジナルのマップ係数だけがご利用できます。 さらに、ダウンロードについて、新しいwwPDBのマップに対しては、構造因子ファイル、mmCIFフォーマットファイル、私達が作成したMTZおよび フルマップのCCP4ファイルをご利用頂けます。一方、既存のエントリーについては、これまでのパイプラインで生成されてきた精密化ファイルとともに、 構造因子ファイルとCCP4ファイルだけがご利用頂けます。 これまでのように、CCP4フォーマットで作成されたローカルマップは、両方のタイプで、ダウンロードしてご利用頂けます。
FTPサイトに保管されているデータについては、全ての新しいwwPDBマップ係数が更新されるまでの間は、我々の既存のデータは、 これまで通り、ftp://ftp.pdbj.org/edmap/から、ご利用頂けます。 wwPDBのマップデータについては、FTPサイトの"validation_reports"フォルダから公開されています。
- /pub/pdb/validation_reports/*/*/*_validation_fo-fc_map_coef.cif.gz
- /pub/pdb/validation_reports/*/*/*_validation_2fo-fc_map_coef.cif.gz
一方、MTZファイルと、CCP4ファイルにつきましては、以下の新しい"edmap2"フォルダからご利用頂けます。
- /edmap2/mtz/*_validation_fo-fc_map_coef.mtz
- /edmap2/ccp4/*_validation_fo-fc_map_coef.ccp4
リニューアルされたEDmapサービスについて、ご質問等ございましたら、PDBjお問い合わせページ より、ご連絡ください。
[wwPDB] PDBアーカイブから、NMR実験データを統合フォーマットで提供します
2020年3月から、OneDepシステムは、NMR-STARまたはNEF形式の単一ファイルでNMRデータのアップロードの受付けを開始します。現在は、化学シフト、距離制限、およびピークリストのような各種NMR実験データが個別にアップロードされていますが、この状況が変わることになります。
NMR-STARは、NMRのデータを記述する公式なwwPDBのフォーマットであり、広範なディクショナリ [GitHub; Ulrich, E. L. et al. (2019) NMR-STAR: comprehensive ontology for representing, archiving and exchanging data from nuclear magnetic resonance spectroscopic experiments Journal of Biomolecular NMR, 73: 5–9. doi: 10.1007/s10858-018-0220-3] でサポートされています。 一方、NEF (NMR exchange format; Gutmanas et al. (2015) NMR Exchange Format: a unified and open standard for representation of NMR restraint data Nature Structural & Molecular Biology 22: 433–434 doi: 10.1038/nsmb.3041) は、軽量なフォーマット及びディクショナリで定義され、NMR構造決定において主流のソフトウェアがサポートしています。 これら2つの相互変換可能な標準フォーマットを一つのデータファイルとして利用することにより、データの保管や配布が簡単になるだけでなく、登録過程が容易になります。
統合データファイルを伴って登録される新しいエントリーについては、NMR-STARフォーマットで単一のファイルとしてNMRの実験データをPDB FTPアーカイブ上に配置します。NEFフォーマットへ最大限変換したファイルも提供します。 これら統合されたNMRデータファイルは、アーカイブ上で、既存のnmr_restraintsやnmr_chemical_shiftsと並んで作成される、新しいディレクトリ「nmr_data」内に追加されます。 加えて、これまでに利用ユーザに対応するため、統合ファイルは、既存のディレクトリ「nmr_restraints」 や「nmr_chemical_shifts」内にもコピーされます。
関連するNMRデータに容易にアクセスできるよう、NMR統合データファイルの標準化した命名規則も開発しています。ファイル名はPDBアクセションコードで始まり、nmr_dataが続き、その後にフォーマットタイプの拡張子が続きます。 例えば、「2lcb_nmr_data.nef」や 「2lcb_nmr_data.str」となります。 統合ファイルでのNMRデータの受け付け・配布の開始は、2020年3月頃を予定しています。
[ wwPDB News ]
1/10(金)、韓国生体分子科学連合学会にて、ランチョンセミナーを開催します
- 学会名
- 韓国生体分子科学連合学会
- プログラム・日程表
- ここから
- 日程
- 2020年1月10日(金)
- 会場
- 韓国・光州市・光州科学技術院(GIST)
- 定員
- 200名
- 参加費
- 無料
- 参加登録
- 当日整理券を配布します
<演者・演題>
- 講演1:
- OneDep: Unified System for Deposition, Biocuration, and Validation of Macromolecular Structures
中川敦史 (大阪大学 蛋白質研究所) - 講演2:
- Mandatory submission of PDBx/mmCIF format files for crystallographic depositions to the Protein Data Bank (PDB)
伊藤暢聡(東京医科歯科大学)
[wwPDB] EMDBのポリシーと処理手順に関する文書を公開しました
EMDBデータに対する首尾一貫したルールを確認するため、EMDBチームは、アーカイブに対する包括的なポリシーと処理手順に関する文書を作成しました。 この文書は、EMDBウェブサイトから公開されています。
2002年の設立以来、電子顕微鏡データバンク(EMDB; https://emdb-empiar.org/)は、公開アーカイブとして、生体高分子、その複合体、 細胞構造の3次元低温(クライオ)電子顕微鏡(cryo-EM)マップと断層像を保管しています。 2002年に最初の8つのEMDBエントリーが公開されて以来、EMDBアーカイブのエントリー数は着実に増大し続け、現在はほぼ1万件のマップが公開されています。 2016年からは、EMDBエントリーは、wwPDBのOneDepシステムを介して登録及び処理が行われ、 登録者の所属地域によって、EMDB、PDBe、RCSB PDB及びPDBjの各サイトが分担してバイオキュレーション処理作業を行っています。
ポリシーの文書では、データ登録の要件や受付可能なフォーマット、エントリーの修正や公開について述べています。 例えば、単粒子解析データ法の登録に際して、フーリエシェル相関(FSC)データのような補助的ファイルだけでなく、 論文内で示される一次マップや生データのマップ(マスクなし、フィルターなし、シャープなし)、マスクなしのハーフマップを含めることを推奨しています。 またEMDBは、バイオキュレーション処理後に作成される公式なwwPDBの検証レポートを、登録者が論文の投稿・レビューを行う過程で、学術誌の編集者や査読者へ提出するよう勧めています。 現在wwPDBの検証レポートには、EMのマップ/トモグラムの検証結果や該当する場合はマップとモデルの検証結果も含まれています。
近年、クライオ電子顕微鏡法のコミュニティ内で、全データを一般に公開しようという要望が高まってきたことに対応して、 EMDBは、全ての原子モデル座標をPDBへ、全ての電子顕微鏡再構成データをEMDBへ、 そして全ての生データ(トモグラムの傾斜シリーズを含む)をEMPIARへ登録することを強く推奨しています。 これらアーカイブ内の関連するエントリーは、相互に参照でき、ユーザは必要な情報を簡単に見つけてデータにアクセスできます。
EMDBチームと母体組織のwwPDBは、ユーザや登録者からのポリシーと処理手順についてコメントを歓迎しています。 コメント等ございましたら、emdbhelp@ebi.ac.ukまで、メール(英語)にてご連絡ください。
[ wwPDB News ]
1/29(水)、蛋白研セミナー:ケンブリッジ構造データベース(CSD)利用セミナーを開催します
蛋白研セミナー:ケンブリッジ構造データベース(CSD)利用セミナー
- 日時
- 2020年1月29日(水)13:00—17:15
- 場所
- 大阪大学 蛋白質研究所 本館1階講堂
※本セミナーは、 2020年1月30日のPDBj・BINDSユニット連携講習会「創薬・生命科学における構造データの利用法」と 協同で蛋白研セミナーとして開催いたします。両日参加ではなく1日だけの参加でもかまいません。
<プログラム>
- 13:00-13:15
- 「趣旨説明 & Intro」栗栖 源嗣 (大阪大学 蛋白質研究所)
- 13:15-14:00
- 「CSD-System:ConQuest, Mercury」 岡村 高明(大阪大学・理)/ 藤内 謙光(大阪大学・工)
- 14:00-14:15
- 「Deposit StructuresとenCIFer 」 藤内 謙光(大阪大学・工)/ 岡村 高明(大阪大学・理)
- 14:15-14:30
- 休憩
- 14:30-15:00
- 「Mogul & IsoStar (+ FIM/SuperStar) 」桜井 尋海(化学情報協会)
- 15:00-15:45
- 「CSD-Materials: DASH, Mercury-Materials 」植草 秀裕(東工大)
- 15:45-16:15
- 休憩
- 16:15-17:00
- 「CSD-Discovery」ケンブリッジ結晶学データセンター(CCDC)スタッフ(※TV会議による)
- 17:00-17:15
- 質問タイム
- 【オーガナイザー】
- 栗栖 源嗣、川端 猛(大阪大学 蛋白質研究所)
- 【主催】
- 大阪大学 蛋白質研究所
- 【連絡先】
- PDBjのお問い合わせページ をご利用ください。