+ データを開く
データを開く
- 基本情報
基本情報
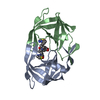
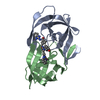
| 登録情報 | データベース: PDB / ID: 4b3p | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structures of HIV-1 RT and RNA-DNA Complex Reveal a Unique RT Conformation and Substrate Interface | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | HYDROLASE/RNA/DNA / HYDROLASE-RNA-DNA COMPLEX / RNASE H / SUBUNIT INTERFACE / HYBRID | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報integrase activity / Integration of viral DNA into host genomic DNA / Autointegration results in viral DNA circles / Minus-strand DNA synthesis / Plus-strand DNA synthesis / 2-LTR circle formation / Uncoating of the HIV Virion / Vpr-mediated nuclear import of PICs / Early Phase of HIV Life Cycle / Integration of provirus ...integrase activity / Integration of viral DNA into host genomic DNA / Autointegration results in viral DNA circles / Minus-strand DNA synthesis / Plus-strand DNA synthesis / 2-LTR circle formation / Uncoating of the HIV Virion / Vpr-mediated nuclear import of PICs / Early Phase of HIV Life Cycle / Integration of provirus / APOBEC3G mediated resistance to HIV-1 infection / Binding and entry of HIV virion / viral life cycle / HIV-1 retropepsin / symbiont-mediated activation of host apoptosis / retroviral ribonuclease H / exoribonuclease H / exoribonuclease H activity / Assembly Of The HIV Virion / Budding and maturation of HIV virion / protein processing / host multivesicular body / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency / telomerase activity / viral penetration into host nucleus / RNA stem-loop binding / RNA-DNA hybrid ribonuclease activity / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / peptidase activity / host cell / viral nucleocapsid / DNA recombination / DNA-directed DNA polymerase / aspartic-type endopeptidase activity / 加水分解酵素; エステル加水分解酵素 / DNA-directed DNA polymerase activity / symbiont-mediated suppression of host gene expression / symbiont entry into host cell / viral translational frameshifting / lipid binding / host cell nucleus / host cell plasma membrane / virion membrane / structural molecule activity / DNA binding / zinc ion binding / identical protein binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   HUMAN IMMUNODEFICIENCY VIRUS 1 (ヒト免疫不全ウイルス) HUMAN IMMUNODEFICIENCY VIRUS 1 (ヒト免疫不全ウイルス)SYNTHETIC CONSTRUCT (人工物) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 4.839 Å 分子置換 / 解像度: 4.839 Å | |||||||||
 データ登録者 データ登録者 | Lapkouski, M. / Tian, L. / Miller, J.T. / Le Grice, S.F.J. / Yang, W. | |||||||||
 引用 引用 |  ジャーナル: Nat.Struct.Mol.Biol. / 年: 2013 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2013タイトル: Complexes of HIV-1 RT, Nnrti and RNA/DNA Hybrid Reveal a Structure Compatible with RNA Degradation 著者: Lapkouski, M. / Tian, L. / Miller, J.T. / Le Grice, S.F.J. / Yang, W. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4b3p.cif.gz 4b3p.cif.gz | 223.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4b3p.ent.gz pdb4b3p.ent.gz | 169.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4b3p.json.gz 4b3p.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b3/4b3p https://data.pdbj.org/pub/pdb/validation_reports/b3/4b3p ftp://data.pdbj.org/pub/pdb/validation_reports/b3/4b3p ftp://data.pdbj.org/pub/pdb/validation_reports/b3/4b3p | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 64380.832 Da / 分子数: 1 / 変異: YES / 由来タイプ: 組換発現 由来: (組換発現)   HUMAN IMMUNODEFICIENCY VIRUS 1 (ヒト免疫不全ウイルス) HUMAN IMMUNODEFICIENCY VIRUS 1 (ヒト免疫不全ウイルス)発現宿主:  参照: UniProt: P04585, RNA-directed DNA polymerase, DNA-directed DNA polymerase, retroviral ribonuclease H, HIV-1 retropepsin, exoribonuclease H |
|---|---|
| #2: タンパク質 | 分子量: 52973.785 Da / 分子数: 1 / 変異: YES / 由来タイプ: 組換発現 由来: (組換発現)   HUMAN IMMUNODEFICIENCY VIRUS 1 (ヒト免疫不全ウイルス) HUMAN IMMUNODEFICIENCY VIRUS 1 (ヒト免疫不全ウイルス)発現宿主:  参照: UniProt: P04585, RNA-directed DNA polymerase, DNA-directed DNA polymerase, retroviral ribonuclease H, HIV-1 retropepsin |
| #3: DNA鎖 | 分子量: 8962.752 Da / 分子数: 1 / Fragment: PRIMER DNA / 由来タイプ: 合成 / 由来: (合成) SYNTHETIC CONSTRUCT (人工物) |
| #4: RNA鎖 | 分子量: 10646.402 Da / 分子数: 1 / Fragment: TEMPLATE RNA / 由来タイプ: 合成 / 由来: (合成) SYNTHETIC CONSTRUCT (人工物) |
| 配列の詳細 | P66 MUTATIONS S68G, R83K, I411V, N447S, R461K, Y483H, D498A, V559I P51 MUTATIONS S68G, R83K, I411V, ...P66 MUTATIONS S68G, R83K, I411V, N447S, R461K, Y483H, D498A, V559I P51 MUTATIONS S68G, R83K, I411V, N-TERMINAL MRGSHHHHHH |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.93 Å3/Da / 溶媒含有率: 62 % / 解説: NONE |
|---|---|
| 結晶化 | 温度: 277 K / 手法: 蒸気拡散法 / pH: 6.5 詳細: RT COMPLEX WAS MIXED WITH RESERVOIR SOLUTION CONTAINING 50MM SODIUM CACODYLATE PH6.5), 10MM MGCL2, 0.2M KCL, AND 10% PEG4000 (W/V). VAPOR DIFFUSION 4C. |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 22-BM / 波長: 1 / ビームライン: 22-BM / 波長: 1 |
| 検出器 | タイプ: MARMOSAIC 225 mm CCD / 検出器: CCD / 日付: 2011年4月19日 / 詳細: MIRRORS VERTICAL FOCUSING |
| 放射 | モノクロメーター: DOUBLE CRYSTAL - SI (220) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1 Å / 相対比: 1 |
| 反射 | 解像度: 4.85→50 Å / Num. obs: 9097 / % possible obs: 99.7 % / Observed criterion σ(I): 2 / 冗長度: 11.6 % / Biso Wilson estimate: 185.31 Å2 / Rsym value: 0.16 / Net I/σ(I): 14.7 |
| 反射 シェル | 解像度: 4.85→5.02 Å / 冗長度: 7.5 % / Mean I/σ(I) obs: 2.1 / Rsym value: 0.63 / % possible all: 99 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ID 1RTD, 1FK9 解像度: 4.839→46.096 Å / SU ML: 1.06 / σ(F): 1.34 / 位相誤差: 50.33 / 立体化学のターゲット値: ML
| ||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 185 Å2 | ||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 4.839→46.096 Å
| ||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー































































































 PDBj
PDBj




































































