-検索条件
-検索結果
検索 (著者・登録者: zhang & xc)の結果100件中、1から50件目までを表示しています

EMDB-39025: 
Structure of HCoV-HKU1A spike in the functionally anchored-3up conformation with 3TMPRSS2

EMDB-39036: 
Structure of HCoV-HKU1C spike in the functionally anchored-1up conformation with 1TMPRSS2

EMDB-39037: 
Structure of HCoV-HKU1C spike in the functionally anchored-2up conformation with 2TMPRSS2

EMDB-39038: 
Structure of HCoV-HKU1C spike in the functionally anchored-3up conformation with 2TMPRSS2

EMDB-39039: 
Structure of HCoV-HKU1C spike in the functionally anchored-3up conformation with 3TMPRSS2

EMDB-39040: 
Local structure of HCoV-HKU1C spike in complex with TMPRSS2 and glycan
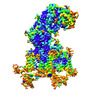
EMDB-34880: 
Cryo-EM structure of human high-voltage activated L-type calcium channel CaV1.2 (apo)

EMDB-34891: 
Cryo-EM structure of human high-voltage activated L-type calcium channel CaV1.2 in complex with tetrandrine (TET)
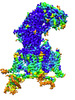
EMDB-34892: 
Cryo-EM structure of human high-voltage activated L-type calcium channel CaV1.2 in complex with benidipine (BEN)

EMDB-35492: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with ceramide

EMDB-35493: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with diacylglycerol/phosphoethanolamine

EMDB-37383: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with ceramide/phosphoethanolamine

EMDB-37385: 
The cryo-EM structure of human sphingomyelin synthase-related protein

EMDB-33808: 
Structure of human R-type voltage-gated CaV2.3-alpha2/delta1-beta1 channel complex in the topiramate-bound state

EMDB-29281: 
Cryo-EM structure of STING oligomer bound to cGAMP and NVS-STG2

EMDB-29282: 
Cryo-EM structure of STING oligomer bound to cGAMP, NVS-STG2 and C53
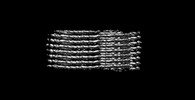
EMDB-40305: 
Cryo-EM structure of insulin amyloid-like fibril that is composed of two antiparallel protofilaments

EMDB-32252: 
Cryo-EM structure of human cohesin-CTCF-DNA complex

EMDB-28536: 
FAM46C/BCCIPalpha/Nanobody complex

EMDB-33241: 
Cryo-EM Structure of Human Niacin Receptor HCA2-Gi protein complex

EMDB-33285: 
Structure of human R-type voltage-gated CaV2.3-alpha2/delta1-beta1 channel complex in the ligand-free (apo) state

EMDB-32356: 
Computational design of a potent Epstein-Barr virus fusion protein gB nanoparticle vaccine

EMDB-32088: 
Computational design of a potent Epstein-Barr virus fusion protein gB nanoparticle vaccine

EMDB-32971: 
Structure of a human NHE3-CHP1 complex in the autoinhibited state

EMDB-26455: 
Cryo-EM structure of a synaptobrevin-Munc18-1-syntaxin-1 complex class 2

EMDB-26456: 
cryo-EM structures of a synaptobrevin-Munc18-1-syntaxin-1 complex class1

EMDB-32341: 
cryo-EM structure of human NaV1.3/beta1/beta2-bulleyaconitineA

EMDB-32343: 
Cryo-EM structure of human NaV1.3/beta1/beta2-ICA121431

EMDB-25188: 
Full-length insulin receptor bound with site 1 binding deficient mutant insulin (A-V3E)

EMDB-25189: 
Full-length insulin receptor bound with site 2 binding deficient mutant insulin (A-L13R) -- asymmetric conformation

EMDB-25190: 
Full-length insulin receptor bound with site 2 binding deficient mutant insulin (A-L13R) -- symmetric conformation

EMDB-25191: 
Full-length insulin receptor bound with site 2 binding deficient mutant insulin (B-L17R) -- asymmetric conformation

EMDB-25192: 
Full-length insulin receptor bound with site 2 binding deficient mutant insulin (B-L17R) -- symmetric conformation

EMDB-25193: 
Full-length insulin receptor bound with both site 1 binding deficient mutant insulin (A-V3E) and site 2 binding deficient mutant insulin (A-L13R)

EMDB-25428: 
Full-length insulin receptor bound with unsaturated insulin WT (2 insulin bound) symmetric conformation

EMDB-25429: 
Full-length insulin receptor bound with unsaturated insulin WT (1 insulin bound) asymmetric conformation

EMDB-25430: 
Full-length insulin receptor bound with unsaturated insulin WT (2 insulins bound) asymmetric conformation (Conformation 1)

EMDB-25431: 
Full-length insulin receptor bound with unsaturated insulin WT (2 insulins bound) asymmetric conformation (Conformation 2)

EMDB-32336: 
Structure of a human glycosylphosphatidylinositol (GPI) transamidase

EMDB-32452: 
Structure of a human glycosylphosphatidylinositol (GPI) transamidase-RNF121 complex

EMDB-25142: 
Human STING bound to both cGAMP and 1-[(2-chloro-6-fluorophenyl)methyl]-3,3-dimethyl-2-oxo-N-[(2,4,6-trifluorophenyl)methyl]-2,3-dihydro-1H-indole-6-carboxamide (Compound 53)

EMDB-31442: 
Lysophospholipid acyltransferase LPCAT3 in complex with lysophosphatidylcholine

EMDB-31443: 
Lysophospholipid acyltransferase LPCAT3 in a complex with Arachidonoyl-CoA

EMDB-31958: 
Human N-type voltage gated calcium channel CaV2.2-alpha2/delta1-beta1 complex, apo state

EMDB-31959: 
Human N-type voltage gated calcium channel CaV2.2-alpha2/delta1-beta1 complex, bound to ziconotide

EMDB-31960: 
Human N-type voltage gated calcium channel CaV2.2-alpha2/delta1-beta1 complex, bound to PD173212

EMDB-31961: 
Human N-type voltage gated calcium channel CaV2.2-alpha2/delta1-beta1 complex, bound to CaV2.2-blocker1

EMDB-31459: 
DNQX-bound GluK2-1xNeto2 complex, with asymmetric LBD

EMDB-31460: 
Kainate-bound GluK2-1xNeto2 complex, at the desensitized state

EMDB-31462: 
DNQX-bound GluK2-1xNeto2 complex
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
