-検索条件
-検索結果
検索 (著者・登録者: yvon & m)の結果93件中、1から50件目までを表示しています

EMDB-50071: 
Cryo-EM structure of the icosahedral lumazine synthase from Vicia faba.

PDB-9ez8: 
Cryo-EM structure of the icosahedral lumazine synthase from Vicia faba.

EMDB-15903: 
Automated simulation-based refinement of maltoporin into a cryo-EM density

PDB-8b7v: 
Automated simulation-based refinement of maltoporin into a cryo-EM density
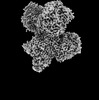
EMDB-16376: 
CryoEM structure of a tungsten-containing aldehyde oxidoreductase from Aromatoleum aromaticum

PDB-8c0z: 
CryoEM structure of a tungsten-containing aldehyde oxidoreductase from Aromatoleum aromaticum

EMDB-15894: 
KimA from B. subtilis with nucleotide second-messenger c-di-AMP bound

EMDB-15895: 
Upright KimA dimer with bound c-di-AMP from B. subtilis

PDB-8b70: 
KimA from B. subtilis with nucleotide second-messenger c-di-AMP bound

PDB-8b71: 
Upright KimA dimer with bound c-di-AMP from B. subtilis

EMDB-11953: 
SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2 (State 1) - Composite Map

EMDB-11954: 
SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2 (State 2)

EMDB-14810: 
SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2 (State 1) - Consensus Map

EMDB-14811: 
SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2 (State 1) - Focused Refinement

EMDB-25448: 
Negative-stain EM reconstruction of SpFN_1B-06-PL, a SARS-CoV-2 spike fused to H.pylori ferritin nanoparticle vaccine candidate
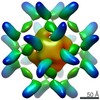
EMDB-25449: 
RFN_131, a Ferritin-based Nanoparticle Vaccine Candidate Displaying the SARS-CoV-2 Receptor-Binding Domain

EMDB-25450: 
pCoV146, a Ferritin-based Nanoparticle Vaccine Candidate Displaying the SARS-CoV-2 Spike Receptor-Binding and N-Terminal Domains

EMDB-25451: 
pCoV111, a Ferritin-based Nanoparticle Vaccine Candidate Displaying the SARS-CoV-2 Spike S1 Subunit

EMDB-22995: 
Spike protein trimer

EMDB-13428: 
Pfu sArlG (PF0333, N-d31-ArlG)

EMDB-12675: 
GLIC, pentameric ligand gated ion channel, pH 3 State 2

EMDB-12677: 
GLIC, pentameric ligand-gated ion channel, pH 5, state 2

EMDB-12678: 
GLIC, pentameric ligand-gated ion channel, pH 7 state 2

EMDB-11202: 
GLIC pentameric ligand-gated ion channel, pH 7

EMDB-11208: 
GLIC pentameric ligand-gated ion channel, pH 5

EMDB-11209: 
GLIC pentameric ligand-gated ion channel, pH 3

PDB-6zgd: 
GLIC pentameric ligand-gated ion channel, pH 7

PDB-6zgj: 
GLIC pentameric ligand-gated ion channel, pH 5

PDB-6zgk: 
GLIC pentameric ligand-gated ion channel, pH 3

EMDB-22993: 
SARS-CoV-2 spike glycoprotein:Fab 5A6 complex I

EMDB-22994: 
SARS-CoV-2 Spike protein in complex with Fab 2H4

EMDB-22997: 
SARS-CoV-2 spike glycoprotein:Fab 3D11 complex

EMDB-23707: 
SARS-CoV-2 Spike:5A6 Fab complex I focused refinement

EMDB-23709: 
SARS-CoV-2 Spike:Fab 3D11 complex focused refinement

PDB-7kqb: 
SARS-CoV-2 spike glycoprotein:Fab 5A6 complex I

PDB-7kqe: 
SARS-CoV-2 spike glycoprotein:Fab 3D11 complex

PDB-7m71: 
SARS-CoV-2 Spike:5A6 Fab complex I focused refinement

PDB-7m7b: 
SARS-CoV-2 Spike:Fab 3D11 complex focused refinement

EMDB-23211: 
Cryo-EM structure of human ACE2 receptor bound to protein encoded by vaccine candidate BNT162b1

EMDB-23215: 
Cryo-EM structure of protein encoded by vaccine candidate BNT162b2

PDB-7l7f: 
Cryo-EM structure of human ACE2 receptor bound to protein encoded by vaccine candidate BNT162b1

PDB-7l7k: 
Cryo-EM structure of protein encoded by vaccine candidate BNT162b2

EMDB-21389: 
Negative Staining Reconstruction of Tryptase Tetramer bound to E104 Fab

EMDB-20714: 
Full length Glycine receptor reconstituted in lipid nanodisc in Apo/Resting conformation

EMDB-20715: 
Full length Glycine receptor reconstituted in lipid nanodisc in Gly-bound desensitized conformation

EMDB-20731: 
Full length Glycine receptor reconstituted in lipid nanodisc in Gly/PTX-bound open/blocked conformation

EMDB-21234: 
Full length Glycine receptor reconstituted in lipid nanodisc in Gly/IVM-conformation (State-1)

EMDB-21236: 
Full length Glycine receptor reconstituted in lipid nanodisc in Gly/IVM-conformation (State-2)

EMDB-21237: 
Full length Glycine receptor reconstituted in lipid nanodisc in Gly/IVM-conformation (State-3)

PDB-6ubs: 
Full length Glycine receptor reconstituted in lipid nanodisc in Apo/Resting conformation
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
