-検索条件
-検索結果
検索 (著者・登録者: yamada & y)の結果66件中、1から50件目までを表示しています
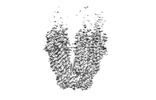
EMDB-27770: 
Structure of LRRC8C-LRRC8A(IL125) Chimera, Class 1
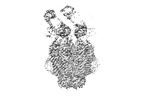
EMDB-27771: 
Structure of LRRC8C-LRRC8A(IL125) Chimera, Class 2
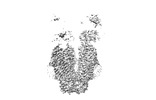
EMDB-27772: 
Structure of LRRC8C-LRRC8A(IL125) Chimera, Class 3

EMDB-27773: 
Structure of LRRC8C-LRRC8A(IL125) Chimera, Class 4
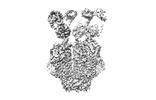
EMDB-27774: 
Structure of LRRC8C-LRRC8A(IL125) Chimera, Class 5

PDB-8dxn: 
Structure of LRRC8C-LRRC8A(IL125) Chimera, Class 1

PDB-8dxo: 
Structure of LRRC8C-LRRC8A(IL125) Chimera, Class 2

PDB-8dxp: 
Structure of LRRC8C-LRRC8A(IL125) Chimera, Class 3

PDB-8dxq: 
Structure of LRRC8C-LRRC8A(IL125) Chimera, Class 4

PDB-8dxr: 
Structure of LRRC8C-LRRC8A(IL125) Chimera, Class 5

EMDB-32041: 
Complex structure of Clostridioides difficile enzymatic component (CDTa) and binding component (CDTb) pore with short stem

EMDB-32043: 
Complex structure of Clostridioides difficile enzymatic component (CDTa) and binding component (CDTb) pore with long stem

EMDB-33188: 
Complex map of Clostridioides difficile enzymatic component (CDTa) and binding component (CDTb) di-heptamer

EMDB-34136: 
Complex structure of Clostridioides difficile binary toxin folded CDTa-bound CDTb-pore (short).

EMDB-34137: 
Complex structure of Clostridioides difficile binary toxin unfolded CDTa-bound CDTb-pore (short).

PDB-7vnj: 
Complex structure of Clostridioides difficile enzymatic component (CDTa) and binding component (CDTb) pore with short stem

PDB-7vnn: 
Complex structure of Clostridioides difficile enzymatic component (CDTa) and binding component (CDTb) pore with long stem

PDB-7yvq: 
Complex structure of Clostridioides difficile binary toxin folded CDTa-bound CDTb-pore (short).

PDB-7yvs: 
Complex structure of Clostridioides difficile binary toxin unfolded CDTa-bound CDTb-pore (short).

EMDB-33045: 
SARS-CoV-2 spike glycoprotein in complex with neutralizing antibody UT28K

EMDB-31089: 
Cryo-EM structure of the octameric state of C-phycocyanin from Thermoleptolyngbya sp. O-77

EMDB-31090: 
Cryo-EM structure of the hexameric state of C-phycocyanin from Thermoleptolyngbya sp. O-77

PDB-7eh7: 
Cryo-EM structure of the octameric state of C-phycocyanin from Thermoleptolyngbya sp. O-77

PDB-7eh8: 
Cryo-EM structure of the hexameric state of C-phycocyanin from Thermoleptolyngbya sp. O-77

EMDB-13120: 
Mature capsid of bacteriophage phiRSA1

PDB-7oz4: 
Mature capsid of bacteriophage phiRSA1

EMDB-11179: 
Head of Semi-jumbo phage RP13

EMDB-11180: 
Head reconstruction of full jumbo phage XacN1

EMDB-0869: 
Cryo-EM structure of the MgtE Mg2+ channel under Mg2+-free conditions

PDB-6lbh: 
Cryo-EM structure of the MgtE Mg2+ channel under Mg2+-free conditions

EMDB-12160: 
Cilia from MOT7 deletion mutant of Chlamydomonas

EMDB-12161: 
Chlamydomonas cilia with MOT7-BCCP labeled

EMDB-12162: 
Microtubule doublet structure from WT Chlamydomonas as a control for MOT7 mutants

EMDB-11178: 
Jumbo Bacteriophage RSL2 - Full icosahedral capsid

EMDB-0730: 
CryoEM map and model of Nitrite Reductase at pH 6.2

EMDB-0731: 
CryoEM map and model of Nitrite Reductase at pH 8.1

PDB-6knf: 
CryoEM map and model of Nitrite Reductase at pH 6.2

PDB-6kng: 
CryoEM map and model of Nitrite Reductase at pH 8.1

EMDB-30362: 
2.05 angstrom resolution structure determination of sulfur oxygenase reductase using 200kV cryo-EM

EMDB-30073: 
Cryo-EM structure of sulfur oxygenase reductase from Sulfurisphaera tokodaii

PDB-6m3x: 
Cryo-EM structure of sulfur oxygenase reductase from Sulfurisphaera tokodaii

EMDB-11049: 
Electron cryo-tomography reconstruction and subvolume averaging of the proximal basal body triplet from Trichonympha

EMDB-0713: 
Complex structure of Iota toxin enzymatic component (Ia) and binding component (Ib) pore with short stem

EMDB-0720: 
Complex structure of Iota toxin enzymatic component (Ia) and binding component (Ib) pore with long stem

EMDB-0721: 
Pore structure of Iota toxin binding component (Ib)

PDB-6klo: 
Complex structure of Iota toxin enzymatic component (Ia) and binding component (Ib) pore with short stem

PDB-6klw: 
Complex structure of Iota toxin enzymatic component (Ia) and binding component (Ib) pore with long stem

PDB-6klx: 
Pore structure of Iota toxin binding component (Ib)

EMDB-0728: 
Structure of human cardiac thin filament in the calcium free state

EMDB-0729: 
Structure of human cardiac thin filament in the calcium bound state
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
