-検索条件
-検索結果
検索 (著者・登録者: woodward & jd)の結果全39件を表示しています

EMDB-19910: 
Cyanide dihydratase from Bacillus pumilus C1 E155R variant with altered helical twist.

EMDB-17409: 
Cyanide dihydratase from Bacillus pumilus C1

EMDB-18031: 
Cryo-EM structure of the DyP peroxidase-loaded encapsulin nanocompartment from Mycobacterium tuberculosis with icosahedral symmetry imposed.

EMDB-16437: 
Cyanide dihydratase from Bacillus pumilus C1 variant - Q86R,H305K,H308K,H323K

EMDB-13797: 
Structure of full-length, monomeric, soluble somatic angiotensin I-converting enzyme showing the N- and C-terminal ellipsoid domains

EMDB-13799: 
Local refinement structure of the N-domain of full-length, monomeric, soluble somatic angiotensin I-converting enzyme

EMDB-13801: 
Local refinement structure of the C-domain of full-length, monomeric, soluble somatic angiotensin I-converting enzyme

EMDB-13802: 
Structure of full-length, dimeric, soluble somatic angiotensin I-converting enzyme

EMDB-13803: 
Local refinement structure of the two interacting N-domains of full-length, dimeric, soluble somatic angiotensin I-converting enzyme

EMDB-13804: 
Local refinement structure of a single N-domain of full-length, dimeric, soluble somatic angiotensin I-converting enzyme

EMDB-13420: 
Cryo-EM structure of Mycobacterium tuberculosis encapsulin

EMDB-11596: 
Shotgun EM of Mycobacterial protein complexes during stationary phase stress

EMDB-11598: 
Shotgun EM of Mycobacterial protein complexes during stationary phase stress.

EMDB-11599: 
Shotgun EM of Mycobacterial protein complexes during stationary phase stress

EMDB-11595: 
Shotgun EM of Mycobacterial protein complexes during stationary phase stress.

EMDB-11597: 
Shotgun EM of Mycobacterial protein complexes during stationary phase stress.

EMDB-4175: 
Native encapsulin from Mycobacterium smegmatis

EMDB-10004: 
Native encapsulated dye decolourising type peroxidase from Mycobacterium smegmatis

EMDB-10005: 
Native bacterioferritin from Mycobacterium smegmatis

EMDB-10008: 
Native encapsulin with bound dye decolourising peroxidase on three fold axis

EMDB-4418: 
Cryo-EM informed directed evolution of Nitrilase 4 leads to a change in quaternary structure.

EMDB-4420: 
Cryo-EM informed directed evolution of Nitrilase 4 leads to a change in quaternary structure.

EMDB-4406: 
Cryo-EM informed directed evolution of Nitrilase 4 leads to a change in quaternary structure.

EMDB-4407: 
Cryo-EM informed directed evolution of Nitrilase 4 leads to a change in quaternary structure.
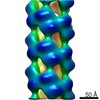
EMDB-4804: 
Cryo-EM informed directed evolution of Nitrilase 4 leads to a change in quaternary structure.

EMDB-0320: 
Cryo-EM informed directed evolution of Nitrilase 4 leads to a change in quaternary structure.

EMDB-4186: 
Glutamine synthetase I from Mycobacterium smegmatis

EMDB-4185: 
Human GroEL obtained by grid-blotting from blue native PAGE

EMDB-3486: 
Substrate specificity in plant nitrilase helical assemblies is determined by their twist.
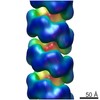
EMDB-3496: 
Substrate specificity in plant nitrilase helical assemblies is determined by their twist.

EMDB-3497: 
Substrate specificity in plant nitrilase helical assemblies is determined by their twist.

EMDB-3498: 
Substrate specificity in plant nitrilase helical assemblies is determined by their twist.

EMDB-3499: 
Substrate specificity in plant nitrilase helical assemblies is determined by their twist.

EMDB-3500: 
Substrate specificity in plant nitrilase helical assemblies is determined by their twist.

EMDB-3501: 
Substrate specificity in plant nitrilase helical assemblies is determined by their twist.

EMDB-3503: 
Substrate specificity in plant nitrilase helical assemblies is determined by their twist.

EMDB-3504: 
Substrate specificity in plant nitrilase helical assemblies is determined by their twist.

EMDB-3505: 
Substrate specificity in plant nitrilase helical assemblies is determined by their twist.

EMDB-1870: 
Three-Dimensional Reconstruction of Heterocapsa circularisquama RNA Virus (HcRNAV-109) by Cryo-Electron Microscopy
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
