-検索条件
-検索結果
検索 (著者・登録者: shi & l)の結果7,038件中、1から50件目までを表示しています

EMDBエントリ 画像なし
EMDB-38418: 
A neutralizing nanobody VHH60 against wt SARS-CoV-2

PDB-8xki: 
A neutralizing nanobody VHH60 against wt SARS-CoV-2

EMDBエントリ 画像なし
EMDB-39901: 
cryo-EM structure of the octreotide-bound SSTR5-Gi complex

EMDBエントリ 画像なし
EMDB-39931: 
Cryo-EM structure of the pasireotide-bound SSTR5-Gi complex

PDB-8zbe: 
cryo-EM structure of the octreotide-bound SSTR5-Gi complex

PDB-8zcj: 
Cryo-EM structure of the pasireotide-bound SSTR5-Gi complex

EMDBエントリ 画像なし
EMDB-18135: 
Cryo-EM structure of the methanogenic Na+ translocating N5-methyl-H4MPT:CoM methyltransferase complex

PDB-8q3v: 
Cryo-EM structure of the methanogenic Na+ translocating N5-methyl-H4MPT:CoM methyltransferase complex

EMDBエントリ 画像なし
EMDB-35361: 
Overall structure of the LAT1-4F2hc bound with tyrosine

PDB-8ida: 
Overall structure of the LAT1-4F2hc bound with tyrosine

EMDBエントリ 画像なし
EMDB-36074: 
Overall structure of the LAT1-4F2hc bound with tryptophan

PDB-8j8m: 
Overall structure of the LAT1-4F2hc bound with tryptophan

EMDB-37105: 
ATP-bound hMRP5 outward-open

EMDB-37554: 
wt-hMRP5 inward-open

EMDB-37555: 
RD-hMRP5-inward open

EMDB-37556: 
ND-hMRP5-inward open

EMDB-37557: 
m6-hMRP5 inward open

EMDB-37558: 
M5PI-bound hMRP5

PDB-8kci: 
ATP-bound hMRP5 outward-open

PDB-8wi0: 
wt-hMRP5 inward-open

PDB-8wi2: 
RD-hMRP5-inward open

PDB-8wi3: 
ND-hMRP5-inward open

PDB-8wi4: 
m6-hMRP5 inward open

PDB-8wi5: 
M5PI-bound hMRP5

EMDB-40971: 
Atomic model of the mammalian mouse Mediator complex with CKM module

EMDB-38502: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2.86 spike protein(6P) in complex with human ACE2

PDB-8xnf: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2.86 spike protein(6P) in complex with human ACE2

EMDB-36083: 
Cryo-EM structure of DDM1-nucleosome complex

EMDB-36084: 
Cryo-EM structure of nucleosome containing Arabidopsis thaliana histones

EMDB-36085: 
Cryo-EM structure of nucleosome containing Arabidopsis thaliana H2A.W

EMDB-38827: 
Cryo-EM structure of SARS-CoV-2 Omicron JN.1 RBD in complex with human ACE2 (local refinement from the spike protein)

EMDB-38460: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2.86 spike protein(6P), 1-RBD-up state

EMDB-38463: 
Cryo-EM structure of SARS-CoV-2 Omicron EG.5 spike protein(6P), RBD-closed state

EMDB-38476: 
Cryo-EM structure of SARS-CoV-2 Omicron HV.1 spike protein(6P), RBD-closed state

EMDB-38488: 
Cryo-EM structure of SARS-CoV-2 Omicron EG.5.1 spike protein(6P), RBD-closed state

EMDB-38495: 
SARS-CoV-2 Omicron EG.5.1 RBD in complex with human ACE2 (local refined from the spike protein)

EMDB-38496: 
SARS-CoV-2 Omicron HV.1 RBD in complex with human ACE2 (local refinement from the spike protein)

EMDB-38498: 
Cryo-EM structure of SARS-CoV-2 Omicron EG.5.1 spike protein(6P) in complex with human ACE2

EMDB-38505: 
Cryo-EM structure of SARS-CoV-2 Omicron HV.1 spike protein(6P) in complex with human ACE2

PDB-8xlv: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2.86 spike protein(6P), 1-RBD-up state

PDB-8xm5: 
Cryo-EM structure of SARS-CoV-2 Omicron EG.5 spike protein(6P), RBD-closed state

PDB-8xmg: 
Cryo-EM structure of SARS-CoV-2 Omicron HV.1 spike protein(6P), RBD-closed state

PDB-8xmt: 
Cryo-EM structure of SARS-CoV-2 Omicron EG.5.1 spike protein(6P), RBD-closed state

PDB-8xn2: 
SARS-CoV-2 Omicron EG.5.1 RBD in complex with human ACE2 (local refined from the spike protein)

PDB-8xn3: 
SARS-CoV-2 Omicron HV.1 RBD in complex with human ACE2 (local refinement from the spike protein)

PDB-8xn5: 
Cryo-EM structure of SARS-CoV-2 Omicron EG.5.1 spike protein(6P) in complex with human ACE2

PDB-8xnk: 
Cryo-EM structure of SARS-CoV-2 Omicron HV.1 spike protein(6P) in complex with human ACE2
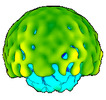
EMDB-50358: 
In vitro-induced genome-releasing intermediate of Rhodobacter microvirus Ebor computed with C5 symmetry
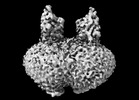
EMDB-44293: 
Cryo-EM structure of MraY in complex with analogue 2
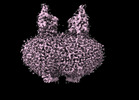
EMDB-44294: 
Cryo-EM structure of MraY in complex with analogue 3
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
