-検索条件
-検索結果
検索 (著者・登録者: rini & j)の結果272件中、1から50件目までを表示しています

EMDB-37007: 
Mycobacterium smegmatis 50S ribosomal subunit-HflX complex

EMDB-38788: 
Mycobacterium smegmatis 50S ribosomal subunit with Erythromycin

PDB-8kab: 
Mycobacterium smegmatis 50S ribosomal subunit-HflX complex

PDB-8xz3: 
Mycobacterium smegmatis 50S ribosomal subunit with Erythromycin

EMDB-41107: 
CryoEM structure of TR-TRAP

PDB-8t9d: 
CryoEM structure of TR-TRAP

EMDB-18592: 
E.coli DNA gyrase in complex with 217 bp substrate DNA and LEI-800

PDB-8qqi: 
E.coli DNA gyrase in complex with 217 bp substrate DNA and LEI-800

EMDB-19075: 
Conformational Landscape of the Type V-K CRISPR-associated TransposonIntegration Assembly CAST V-K composite map

EMDB-19282: 
Conformational Landscape of the Type V-K CRISPR-associated TransposonIntegration Assembly CAST V-K Cas12k domain local-refinement map

EMDB-19283: 
Conformational Landscape of the Type V-K CRISPR-associated TransposonIntegration Assembly CAST V-K TnsC domain local-refinement map

EMDB-19284: 
Conformational Landscape of the Type V-K CRISPR-associated TransposonIntegration Assembly CAST V-K TnsB domain local-refinement map

EMDB-19286: 
consensus map of the V-K CRISPR-associated Transposon Integration Assembly

PDB-8rdu: 
Conformational Landscape of the Type V-K CRISPR-associated TransposonIntegration Assembly CAST V-K composite map

PDB-8rkt: 
Conformational Landscape of the Type V-K CRISPR-associated TransposonIntegration Assembly CAST V-K Cas12k domain local-refinement map

PDB-8rku: 
Conformational Landscape of the Type V-K CRISPR-associated TransposonIntegration Assembly CAST V-K TnsC domain local-refinement map

PDB-8rkv: 
Conformational Landscape of the Type V-K CRISPR-associated TransposonIntegration Assembly CAST V-K TnsB domain local-refinement map

EMDB-40968: 
Atomic model of the mammalian Mediator complex with MED26 subunit

EMDB-40972: 
CryoEM map of TR-TRAP

EMDB-40975: 
CryoEM map of mouse mediator complex with alternate conformation CKM module

PDB-8t1i: 
Atomic model of the mammalian Mediator complex with MED26 subunit

EMDB-42837: 
Cryo-EM structure of GeoCas9 in complex with sgRNA and target DNA

EMDB-42838: 
Cryo-EM structure of iGeoCas9 in complex with sgRNA and target DNA

PDB-8uza: 
Cryo-EM structure of GeoCas9 in complex with sgRNA and target DNA

PDB-8uzb: 
Cryo-EM structure of iGeoCas9 in complex with sgRNA and target DNA

EMDB-43736: 
Umb1 umbrella toxin particle

EMDB-43737: 
Umb1 umbrella toxin particle (local refinement of UmbB1 bound ALF of UmbC1 and UmbA1)

PDB-8w20: 
Umb1 umbrella toxin particle

PDB-8w22: 
Umb1 umbrella toxin particle (local refinement of UmbB1 bound ALF of UmbC1 and UmbA1)

EMDB-15697: 
Cryo-EM structure of shCas12k-sgRNA-dsDNA ternary complex (type V-K CRISPR-associated transposon)

PDB-8axa: 
Cryo-EM structure of shCas12k-sgRNA-dsDNA ternary complex (type V-K CRISPR-associated transposon)

EMDB-15698: 
Cryo-EM structure of Cas12k-sgRNA binary complex (type V-K CRISPR-associated transposon)

PDB-8axb: 
Cryo-EM structure of Cas12k-sgRNA binary complex (type V-K CRISPR-associated transposon)
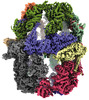
EMDB-19477: 
Saccharomyces cerevisiae FAS type I

EMDB-19489: 
Tobacco mosaic virus from scanning transmission electron microscopy at CSA=2.0 mrad

EMDB-40783: 
MBP-tagged fusion fatty acid synthase from S. cerevisiae

EMDB-40784: 
MBP tagged and Mixed chain fatty acid synthase, FAS2_deltaACP

EMDB-40785: 
MBP tagged mixed chain FAS, fusFAS_deltaACP

EMDB-16321: 
Structure of the human nuclear cap-binding complex bound to NCBP3(560-620) and cap-analogue m7GpppG

PDB-8by6: 
Structure of the human nuclear cap-binding complex bound to NCBP3(560-620) and cap-analogue m7GpppG

EMDB-17763: 
Structure of the human nuclear cap-binding complex bound to ARS2[147-871] and m7GTP

EMDB-17784: 
Structure of the human nuclear cap-binding complex bound to PHAX and m7G-capped RNA

PDB-8pmp: 
Structure of the human nuclear cap-binding complex bound to ARS2[147-871] and m7GTP

PDB-8pnt: 
Structure of the human nuclear cap-binding complex bound to PHAX and m7G-capped RNA
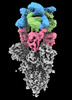
EMDB-16453: 
SARS-CoV-2 Omicron Variant Spike Trimer in complex with three 17T2 Fabs

EMDB-16473: 
SARS-CoV-2 spike in complex with the 17T2 neutralizing antibody Fab fragment (local refinement of RBD and Fab)

PDB-8c89: 
SARS-CoV-2 spike in complex with the 17T2 neutralizing antibody Fab fragment (local refinement of RBD and Fab)

EMDB-18874: 
Cofactor-free Tau 4R2N isoform

PDB-8r3t: 
Cofactor-free Tau 4R2N isoform

EMDB-17584: 
RAD51 filament on ssDNA bound by the BRCA2 c-terminus
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
