-検索条件
-検索結果
検索 (著者・登録者: park & kh)の結果74件中、1から50件目までを表示しています

EMDB-42074: 
Representative tomogram of Enterococcus faecium WT Com15

EMDB-42086: 
Representative tomogram of Enterococcus faecium SagA complementation strain

EMDB-42087: 
Representative tomogram of Enterococcus faecium SagA deletion strain

EMDB-43033: 
Structure of 80alpha portal protein expressed in E. coli

EMDB-43142: 
SaPI1 mature capsid structure containing DNA

EMDB-43143: 
SaPI1 mature capsid structure without DNA

EMDB-43145: 
SaPI1 portal structure in mature capsids containing DNA

EMDB-43146: 
SaPI1 portal structure in mature capsids without DNA

EMDB-43147: 
SaPI1 portal-capsid interface in mature capsids with DNA

PDB-8v8b: 
Structure of 80alpha portal protein expressed in E. coli

PDB-8vd4: 
SaPI1 mature capsid structure containing DNA

PDB-8vd5: 
SaPI1 mature capsid structure without DNA

PDB-8vd8: 
SaPI1 portal structure in mature capsids containing DNA

PDB-8vdc: 
SaPI1 portal structure in mature capsids without DNA

PDB-8vde: 
SaPI1 portal-capsid interface in mature capsids with DNA

PDB-8c5v: 
Chemotaxis core signalling unit from E protein lysed E. coli cells

EMDB-27703: 
Structure of RBD directed antibody DH1047 in complex with SARS-CoV-2 spike: Local refinement of RBD-Fab interace

PDB-8dtk: 
Structure of RBD directed antibody DH1047 in complex with SARS-CoV-2 spike: Local refinement of RBD-Fab interace

EMDB-15641: 
Structure of the Native Chemotaxis Core Signalling Complex from E-gene lysed E. coli cells.

EMDB-15642: 
Native Chemotaxis Core Signalling Complex from E. coli, with focused alignment on the baseplate (CheA-CheW)
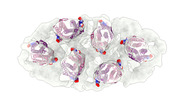
EMDB-15643: 
Native Chemotaxis Core Signalling Complex from E. coli, Focused alignment on ligand binding domain
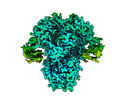
EMDB-26727: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the proximal conformation
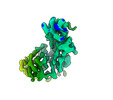
EMDB-26729: 
CCoV-HuPn-2018 S in the proximal conformation (local refinement of domain 0)
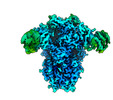
EMDB-26730: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the swung out conformation
EMDB-26731: 
CCoV-HuPn-2018 S in the swung out conformation (local refinement of domain 0)

PDB-7us6: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the proximal conformation

PDB-7us9: 
CCoV-HuPn-2018 S in the proximal conformation (local refinement of domain 0)

PDB-7usa: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the swung out conformation

PDB-7usb: 
CCoV-HuPn-2018 S in the swung out conformation (local refinement of domain 0)

EMDB-26676: 
Three RBD-down state of SARS-CoV-2 D614G spike in complex with the SP1-77 neutralizing antibody Fab fragment

EMDB-26677: 
Three RBD-down state of SARS-CoV-2 D614G spike in complex with the SP1-77 neutralizing antibody Fab fragment (local refinement of the RBD and Fab variable domains)

EMDB-26678: 
An antibody from single human VH-rearranging mouse neutralizes all SARS-CoV-2 variants through BA.5 by inhibiting membrane fusion

EMDB-27043: 
Negative stain electron microscopy single particle reconstruction of monoclonal antibody SP1-77 Fab in complex with SARS-CoV2 2P spike

EMDB-27044: 
Negative stain electron microscopy single particle reconstruction of monoclonal antibody VHH7-7-53 Fab in complex with SARS-CoV2 2P spike

EMDB-27046: 
Negative stain electron microscopy single particle reconstruction of monoclonal antibody VHH7-5-82 Fab in complex with SARS-CoV2 2P spike

PDB-7upw: 
Three RBD-down state of SARS-CoV-2 D614G spike in complex with the SP1-77 neutralizing antibody Fab fragment

PDB-7upx: 
Three RBD-down state of SARS-CoV-2 D614G spike in complex with the SP1-77 neutralizing antibody Fab fragment (local refinement of the RBD and Fab variable domains)

PDB-7upy: 
An antibody from single human VH-rearranging mouse neutralizes all SARS-CoV-2 variants through BA.5 by inhibiting membrane fusion

EMDB-25105: 
Structure of SARS-CoV S protein in complex with Receptor Binding Domain antibody DH1047

PDB-7sg4: 
Structure of SARS-CoV S protein in complex with Receptor Binding Domain antibody DH1047

EMDB-21514: 
map of the mammalian Mediator complex

PDB-6w1s: 
Atomic model of the mammalian Mediator complex

EMDB-22309: 
Dimeric Immunoglobin A (dIgA)

EMDB-22310: 
Secretory Immunoglobin A (SIgA)

PDB-7jg1: 
Dimeric Immunoglobin A (dIgA)

PDB-7jg2: 
Secretory Immunoglobin A (SIgA)

EMDB-22829: 
Human Tom70 in complex with SARS CoV2 Orf9b

PDB-7kdt: 
Human Tom70 in complex with SARS CoV2 Orf9b

PDB-6zet: 
Crystal structure of proteinase K nanocrystals by electron diffraction with a 20 micrometre C2 condenser aperture

PDB-6zeu: 
Crystal structure of proteinase K lamella by electron diffraction with a 50 micrometre C2 condenser aperture
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
