-検索条件
-検索結果
検索 (著者・登録者: mizuno & n)の結果全42件を表示しています
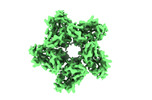
EMDB-35143: 
Cryo-EM structure of the zeaxanthin-bound kin4B8

PDB-8i2z: 
Cryo-EM structure of the zeaxanthin-bound kin4B8

EMDB-26794: 
Tomogram of platelet filopodia in presence of SARS-CoV-2 spike protein

EMDB-26796: 
Tomogram of platelet protrusion with microtubule in presence of SARS-CoV-2 spike protein

EMDB-26798: 
SARS-CoV-2 spike protein closed conformation

EMDB-33293: 
Cryo-EM structure of Cytochrome bo3 from Escherichia coli, apo structure with DMSO

EMDB-33294: 
Cryo-EM structure of Cytochrome bo3 from Escherichia coli, the structure complexed with an allosteric inhibitor N4

PDB-7xmc: 
Cryo-EM structure of Cytochrome bo3 from Escherichia coli, apo structure with DMSO

PDB-7xmd: 
Cryo-EM structure of Cytochrome bo3 from Escherichia coli, the structure complexed with an allosteric inhibitor N4

EMDB-25485: 
In situ cryo-electron tomography reveals local cellular machineries for axon branch development (premature branch)

EMDB-25486: 
In situ cryo-electron tomography reveals local cellular machineries for axon branch development (mature branch)

EMDB-12637: 
Cryo-EM structure of human integrin alpha5beta1 in the half-bent conformation

PDB-7nxd: 
Cryo-EM structure of human integrin alpha5beta1 in the half-bent conformation

EMDB-12634: 
Cryo-EM structure of human integrin alpha5beta1 (open form) in complex with fibronectin and TS2/16 Fv-clasp

PDB-7nwl: 
Cryo-EM structure of human integrin alpha5beta1 (open form) in complex with fibronectin and TS2/16 Fv-clasp

EMDB-12160: 
Cilia from MOT7 deletion mutant of Chlamydomonas

EMDB-12161: 
Chlamydomonas cilia with MOT7-BCCP labeled

EMDB-12162: 
Microtubule doublet structure from WT Chlamydomonas as a control for MOT7 mutants

EMDB-10262: 
Yeast 80S ribosome stalled on SDD1 mRNA.

EMDB-10315: 
The cryo-EM structure of SDD1-stalled collided trisome.

PDB-6snt: 
Yeast 80S ribosome stalled on SDD1 mRNA.

PDB-6sv4: 
The cryo-EM structure of SDD1-stalled collided trisome.

EMDB-4772: 
Cryo-EM structure of autoinhibited human talin-1

PDB-6r9t: 
Cryo-EM structure of autoinhibited human talin-1

EMDB-4188: 
Cryo-EM structure of microtubule co-polymerized with SSNA1.

EMDB-3192: 
Helical reconstruction of amphiphysin N-BAR with a membrane tube radius of 140 Angstrom by cryo-electron microscopy

EMDB-3193: 
Helical reconstruction of amphiphysin N-BAR with a membrane tube radius of 156 Angstrom by cryo-electron microscopy

EMDB-3194: 
Helical reconstruction of amphiphysin N-BAR with a membrane tube radius of 131 Angstrom by cryo-electron microscopy

EMDB-3195: 
Helical reconstruction of amphiphysin N-BAR with a membrane tube radius of 125 Angstrom by cryo-electron microscopy

EMDB-3196: 
Helical reconstruction of amphiphysin N-BAR with a membrane tube radius of 121 Angstrom by cryo-electron microscopy

EMDB-2946: 
cryo-EM structure of HET-s prion infectious form

EMDB-2789: 
Cryo-EM map of the Timeless-Tipin-RPA Complex

EMDB-2673: 
Cryo-EM map of CAP-Gly-microtubule complex

EMDB-2674: 
Cryo-EM map of p150 CAP-Gly-microtubule complex

EMDB-2675: 
Cryo-EM map of CAP-Gly-microtubule complex
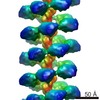
EMDB-2395: 
MuB is an AAA+ ATPase that forms helical filaments to control target selection for DNA transposition
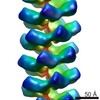
EMDB-2398: 
MuB is an AAA+ ATPase that forms helical filaments to control target selection for DNA transposition
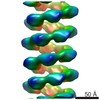
EMDB-2400: 
MuB is an AAA+ ATPase that forms helical filaments to control target selection for DNA transposition

PDB-4bs1: 
MuB is an AAAplus ATPase that forms helical filaments to control target selection for DNA transposition

PDB-4bt0: 
MuB is an AAAplus ATPase that forms helical filaments to control target selection for DNA transposition

PDB-4bt1: 
MuB is an AAAplus ATPase that forms helical filaments to control target selection for DNA transposition

EMDB-2018: 
CryoEM reconstruction of endophilin N-BAR tubes
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
