-検索条件
-検索結果
検索 (著者・登録者: ma & ss)の結果7,545件中、1から50件目までを表示しています

EMDBエントリ 画像なし
EMDB-16426: 
CryoEM structure of the Hendra henipavirus nucleocapsid sauronoid assembly multimer

PDB-8c4h: 
CryoEM structure of the Hendra henipavirus nucleocapsid sauronoid assembly multimer

PDB-8cbw: 
CryoEM structure of the Hendra henipavirus nucleocapsid sauronoid assembly monomer

EMDBエントリ 画像なし
EMDB-19929: 
Structural basis of D9-THC analog activity at the Cannabinoid 1 receptor

PDB-9erx: 
Structural basis of D9-THC analog activity at the Cannabinoid 1 receptor

EMDBエントリ 画像なし
EMDB-50296: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

PDB-9fbv: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

EMDBエントリ 画像なし
EMDB-40046: 
CryoEM structure of Influenza A virus A/Melbourner/1/1946 (H1N1) hemagglutinin bound to GS10-X6-BE4 Fab

PDB-8ghk: 
CryoEM structure of Influenza A virus A/Melbourner/1/1946 (H1N1) hemagglutinin bound to GS10-X6-BE4 Fab

EMDBエントリ 画像なし
EMDB-18202: 
Copper-transporting ATPase HMA4 in E1 state apo

EMDBエントリ 画像なし
EMDB-18203: 
Copper-transporting ATPase HMA4 in E1 state with Cu

EMDBエントリ 画像なし
EMDB-18204: 
Copper-transporting ATPase HMA4 in E2P state with AlF

EMDBエントリ 画像なし
EMDB-18205: 
Copper-transporting ATPase HMA4 in E2P state with BeF

PDB-8q73: 
Copper-transporting ATPase HMA4 in E1 state apo

PDB-8q74: 
Copper-transporting ATPase HMA4 in E1 state with Cu

PDB-8q75: 
Copper-transporting ATPase HMA4 in E2P state with AlF

PDB-8q76: 
Copper-transporting ATPase HMA4 in E2P state with BeF

EMDBエントリ 画像なし
EMDB-41874: 
CryoEM structure of A/Solomon Islands/3/2006 H1 HA in complex with 05.GC.w2.3C10-H1_SI06

EMDB-19163: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

EMDB-19164: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

EMDB-19165: 
Trimeric HSV-2F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

EMDB-19166: 
Trimeric HSV-2G gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

PDB-8rgz: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

PDB-8rh0: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

PDB-8rh1: 
Trimeric HSV-2F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

PDB-8rh2: 
Trimeric HSV-2G gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

EMDB-16375: 
SARS-CoV2 Omicron BA.1 RBD in complex with CAB-A17 antibody

PDB-8c0y: 
SARS-CoV2 Omicron BA.1 RBD in complex with CAB-A17 antibody

EMDB-28966: 
CryoEM map of de novo designed oligomeric protein C4-71_6x

EMDB-28967: 
CryoEM map of de novo designed oligomeric protein C4-71_8x

EMDB-28968: 
CryoEM map of de novo designed oligomeric protein C6-71

EMDB-28969: 
CryoEM map of de novo designed oligomeric protein C6-71_6x

EMDB-28970: 
CryoEM map of de novo designed oligomeric protein C6-71_8x

EMDB-28971: 
CryoEM map of de novo designed oligomeric protein C8-71_6x

EMDB-28972: 
CryoEM map of de novo designed oligomeric protein C8-71_8x
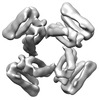
EMDB-28973: 
CryoEM map of de novo designed oligomeric protein C4-81

EMDB-28974: 
CryoEM map of designed oligomeric protein C4-71

EMDB-17197: 
Human TPC2 in Complex with Antagonist (S)-SG-094

EMDB-19108: 
Human TPC2 in Complex withAntagonist (R)-SG-094

PDB-8ouo: 
Human TPC2 in Complex with Antagonist (S)-SG-094

PDB-8p2w: 
Structure of human SIT1 (focussed map / refinement)

PDB-8p2z: 
Structure of human SIT1 bound to L-pipecolate (focussed map / refinement)

PDB-8p30: 
Structure of human SIT1:ACE2 complex (open PD conformation) bound to L-pipecolate

EMDB-17377: 
Structure of human SIT1 (focussed map / refinement)

EMDB-17378: 
Structure of human SIT1:ACE2 complex (open PD conformation)

EMDB-17379: 
Structure of human SIT1:ACE2 complex (closed PD conformation)

EMDB-17380: 
Structure of human SIT1 bound to L-pipecolate (focussed map / refinement)

EMDB-17381: 
Structure of human SIT1:ACE2 complex (open PD conformation) bound to L-pipecolate

EMDB-17382: 
Structure of human SIT1:ACE2 complex (closed PD conformation) bound to L-pipecolate

PDB-8p2x: 
Structure of human SIT1:ACE2 complex (open PD conformation)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
