-検索条件
-検索結果
検索 (著者・登録者: liang & y)の結果2,233件中、1から50件目までを表示しています

EMDB-38873: 
cryo-EM structure of Staphylococcus aureus(ATCC 29213) 50S ribosome in complex with MCX-190.

EMDB-38874: 
Cryo-EM structure of Staphylococcus aureus (15B196) 50S ribosome in complex with MCX-190.

EMDB-38875: 
Cryo-EM structure of Staphylococcus aureus 70S ribosome (strain 15B196) in complex with MCX-190.

EMDB-38876: 
cryo-EM structure of Staphylococcus aureus(ATCC 29213) 70S ribosome in complex with MCX-190.

PDB-8y36: 
cryo-EM structure of Staphylococcus aureus(ATCC 29213) 50S ribosome in complex with MCX-190.

PDB-8y37: 
Cryo-EM structure of Staphylococcus aureus (15B196) 50S ribosome in complex with MCX-190.

PDB-8y38: 
Cryo-EM structure of Staphylococcus aureus 70S ribosome (strain 15B196) in complex with MCX-190.

PDB-8y39: 
cryo-EM structure of Staphylococcus aureus(ATCC 29213) 70S ribosome in complex with MCX-190.

EMDB-44351: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3, V1

PDB-9b8p: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3, V1
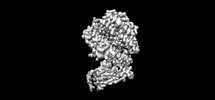
EMDB-38784: 
The structure of fox ACE2 and PT RBD complex

EMDB-38792: 
The structure of fox ACE2 and SARS-CoV RBD complex

PDB-8xyz: 
The structure of fox ACE2 and PT RBD complex

PDB-8xzb: 
The structure of fox ACE2 and SARS-CoV RBD complex

EMDB-44350: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3, Vo

EMDB-44352: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3, peripheral stalks

EMDB-44353: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3

EMDB-44354: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 2

EMDB-44355: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 1

PDB-9b8o: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3, Vo

PDB-9b8q: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3, peripheral stalks

PDB-9brb: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 1

PDB-9brc: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 2

PDB-9brd: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3

EMDB-39858: 
Cryo-EM structure of the insect olfactory receptor OR5-Orco heterocomplex from Acyrthosiphon pisum bound with geranyl acetate
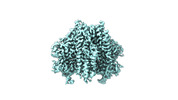
EMDB-39873: 
Cryo-EM structure of the insect olfactory receptor OR5-Orco heterocomplex from Acyrthosiphon pisum

PDB-8z9a: 
Cryo-EM structure of the insect olfactory receptor OR5-Orco heterocomplex from Acyrthosiphon pisum bound with geranyl acetate

PDB-8z9z: 
Cryo-EM structure of the insect olfactory receptor OR5-Orco heterocomplex from Acyrthosiphon pisum

EMDB-38216: 
Cryo-EM structure of SARS-CoV-2 S-BQ.1 in complex with antibody O5C2

PDB-8xbf: 
Cryo-EM structure of SARS-CoV-2 S-BQ.1 in complex with antibody O5C2
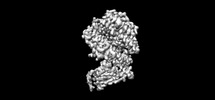
EMDB-38793: 
The structure of fox ACE2 and Omicron BF.7 RBD complex

PDB-8xzd: 
The structure of fox ACE2 and Omicron BF.7 RBD complex

EMDB-37414: 
Structure of PSII-ACPII supercomplex from cryptophyte algae

EMDB-38419: 
Structure of ACPII-CCPII from cryptophyte algae

PDB-8wb4: 
Structure of PSII-ACPII supercomplex from cryptophyte algae

PDB-8xkl: 
Structure of ACPII-CCPII from cryptophyte algae

EMDB-18313: 
Retron-Eco1 filament with ADP-ribosylated Effector (local map with 1 segment)

EMDB-18314: 
Retron-Eco1 filament with inactive effector (E106A, 2 segments)

EMDB-18315: 
Retron-Eco1 filament with ADP-ribosylated Effector (full map with 2 segments)

EMDB-18317: 
Retron-Eco1 filament (2 segments)

EMDB-19792: 
Retron-Eco1 -1 turn mutant filament with ADP-ribosylated Effector (Consensus refinement)

EMDB-19793: 
Retron-Eco1 filament with ADP-ribosylated Effector (Consensus refinement)

PDB-8qbk: 
Retron-Eco1 filament with ADP-ribosylated Effector (local map with 1 segment)

PDB-8qbl: 
Retron-Eco1 filament with inactive effector (E106A, 2 segments)

PDB-8qbm: 
Retron-Eco1 filament with ADP-ribosylated Effector (full map with 2 segments)

EMDB-39920: 
SARS-CoV-2 Omicron BA.2 spike trimer (6P) in complex with D1F6 Fab, head-to-head aggregate

EMDB-39924: 
SARS-CoV-2 Omicron BA.4 spike trimer (6P) in complex with D1F6 Fab, head-to-head aggregate

PDB-8zc2: 
SARS-CoV-2 Omicron BA.2 spike trimer (6P) in complex with D1F6 Fab, head-to-head aggregate

PDB-8zc6: 
SARS-CoV-2 Omicron BA.4 spike trimer (6P) in complex with D1F6 Fab, head-to-head aggregate

EMDB-42676: 
5-HT2AR bound to Lisuride in complex with a mini-Gq protein and an active-state stabilizing single-chain variable fragment (scFv16) obtained by cryo-electron microscopy (cryoEM)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
