-検索条件
-検索結果
検索 (著者・登録者: lei & ht)の結果133件中、1から50件目までを表示しています
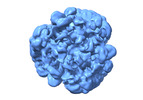
EMDB-19395: 
CryoEM structure of recombinant human Bri2 BRICHOS oligomers

PDB-8rnu: 
CryoEM structure of recombinant human Bri2 BRICHOS oligomers
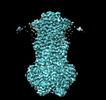
EMDB-40180: 
MsbA bound to cerastecin C

PDB-8gk7: 
MsbA bound to cerastecin C
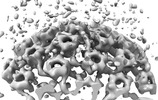
EMDB-18482: 
Herpes simplex virus 1 capsid (WT) vertices in perinuclear NEC-coated vesicles determined in situ

EMDB-18484: 
Herpes simplex virus 1 nuclear egress complex (WT) determined in situ from perinuclear vesicles

EMDB-17974: 
Pseudorabies virus cytosolic C-capsid (US3 KO) vertices determined in situ

EMDB-17975: 
Pseudorabies virus primary enveloped (perinuclear) C-capsid (US3 KO) vertices determined in situ

EMDB-17976: 
Pseudorabies nuclear C-capsids (US3 KO) vertices determined in situ

EMDB-18473: 
Subtomogram average of pseudorabies virus nuclear egress complex helical form (UL31/34) determined in situ

EMDB-18474: 
Subtomogram average of pseudorabies virus nuclear egress complex (UL31/34) determined in situ

EMDB-18479: 
Pseudorabies virus cytosolic C-capsid (WT) vertices determined in situ

EMDB-18480: 
Pseudorabies virus nuclear C-capsid (WT) vertices determined in situ

EMDB-18481: 
Herpes simplex virus 1 cytosolic C-capsid (WT) vertices determined in situ
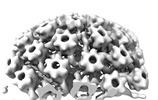
EMDB-18483: 
Herpes simplex virus 1 nuclear C-capsid (WT) vertices determined in situ

EMDB-16255: 
Focus refinement of soluble domain of Adenylyl cyclase 8 bound to stimulatory G protein, Forskolin, ATPalphaS, and Ca2+/Calmodulin in lipid nanodisc conditions

PDB-8bv5: 
Focus refinement of soluble domain of Adenylyl cyclase 8 bound to stimulatory G protein, Forskolin, ATPalphaS, and Ca2+/Calmodulin in lipid nanodisc conditions

EMDB-16249: 
Structure of Adenylyl cyclase 8 bound to stimulatory G protein, Forskolin, ATPalphaS, and Ca2+/Calmodulin in lipid nanodisc

EMDB-16252: 
Structure of Adenylyl cyclase 8 bound to stimulatory G-protein, Ca2+/Calmodulin, Forskolin and MANT-GTP

EMDB-16253: 
Focused refinement of soluble domain of Adenylyl cyclase 8 bound to stimulatory G-protein, Ca2+/Calmodulin, Forskolin and MANT-GTP

EMDB-16254: 
Focused refinement of transmembrane domain of Adenylyl cyclase 8 bound to stimulatory G-protein, Ca2+/Calmodulin, Forskolin and MANT-GTP

PDB-8buz: 
Structure of Adenylyl cyclase 8 bound to stimulatory G-protein, Ca2+/Calmodulin, Forskolin and MANT-GTP

EMDB-27641: 
The structure of the interleukin 11 signalling complex, truncated gp130

EMDB-27642: 
The structure of the IL-11 signalling complex, with full-length extracellular gp130

PDB-8dps: 
The structure of the interleukin 11 signalling complex, truncated gp130

PDB-8dpt: 
The structure of the IL-11 signalling complex, with full-length extracellular gp130

EMDB-16458: 
Electron cryo-tomography and subtomogram averaging of cytoplasmic lattice filaments from mammalian oocytes

EMDB-16472: 
In situ cryo-electron tomogram of cytoplasmic lattice filaments from a mouse oocyte

EMDB-41158: 
CS2it1p2_F7K local refinement for HCMV Trimer in complex with CS2it1p2_F7K Fab and CS4tt1p1_E3K Fab

EMDB-41180: 
Global reconstruction for HCMV Pentamer in complex with CS2pt1p2_A10L Fab and CS3pt1p4_C1L Fab

EMDB-34813: 
Structure of transforming growth factor beta induced protein (TGFBIp) G623R fibril

PDB-8hia: 
Structure of transforming growth factor beta induced protein (TGFBIp) G623R fibril

EMDB-33845: 
Cryo-EM map of Rpd3S complex

EMDB-33846: 
Cryo-EM map of Rpd3S:head-bridge-right arm

EMDB-33847: 
Cryo-EM map of Rpd3S:bridge-left arm

EMDB-33848: 
Cryo-EM map of Eaf3 CHD bound to H3K36me3 nucleosome

EMDB-33849: 
Cryo-EM map of Rpd3S in loose-state Rpd3S-NCP complex

EMDB-33850: 
Cryo-EM map of Rpd3S in close-state Rpd3S-NCP complex

EMDB-33851: 
Cryo-EM map of Rpd3S complex bound to H3K36me3 nucleosome in close state

EMDB-33852: 
Cryo-EM msp of Rpd3S complex bound to H3K36me3 nucleosome in loose state

EMDB-27148: 
Zebrafish MFSD2A isoform B in inward open ligand bound conformation

EMDB-27149: 
Zebrafish MFSD2A isoform B in inward open ligand-free conformation

EMDB-27150: 
Zebrafish MFSD2A isoform B in inward open ligand 1A conformation

EMDB-27151: 
Zebrafish MFSD2A isoform B in inward open ligand 1B conformation

EMDB-27152: 
Zebrafish MFSD2A isoform B in inward open ligand 2B conformation

EMDB-27153: 
Zebrafish MFSD2A isoform B in inward open ligand 3C conformation

EMDB-16128: 
Tomogram of a late endosome of A549 cell infected with influenza A virus.

EMDB-16129: 
Tomogram of a late endosome of A549 cell infected with influenza A virus (Figure 6C).

EMDB-16130: 
Tomogram of a late endosome of A549 cell infected with influenza A virus (Figure 6E)

EMDB-16131: 
Tomogram of a late endosome of A549 cell infected with influenza A virus (Figure 6G)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
