-検索条件
-検索結果
検索 (著者・登録者: kim & kw)の結果137件中、1から50件目までを表示しています
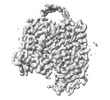
EMDB-35377: 
Cryo-EM structure of GPR156 of GPR156-miniGo-scFv16 complex (local refine)

EMDB-35378: 
Cryo-EM structure of miniGo-scFv16 of GPR156-miniGo-scFv16 complex (local refine)

EMDB-35380: 
Cryo-EM structure of GPR156-miniGo-scFv16 complex

EMDB-35382: 
Cryo-EM structure of GPR156A/B of G-protein free GPR156 (local refine)

EMDB-35389: 
Cryo-EM structure of GPR156C/D of G-protein free GPR156 (local refine)

EMDB-35390: 
Cryo-EM structure of G-protein free GPR156

PDB-8ieb: 
Cryo-EM structure of GPR156 of GPR156-miniGo-scFv16 complex (local refine)

PDB-8iec: 
Cryo-EM structure of miniGo-scFv16 of GPR156-miniGo-scFv16 complex (local refine)

PDB-8ied: 
Cryo-EM structure of GPR156-miniGo-scFv16 complex

PDB-8iei: 
Cryo-EM structure of GPR156A/B of G-protein free GPR156 (local refine)

PDB-8iep: 
Cryo-EM structure of GPR156C/D of G-protein free GPR156 (local refine)

PDB-8ieq: 
Cryo-EM structure of G-protein free GPR156

EMDB-35122: 
Human TRiC-PhLP2A complex in the closed state

EMDB-35199: 
The focused refinement of CCT3-PhLP2A from TRiC-PhLP2A complex in the open state

PDB-8i1u: 
Human TRiC-PhLP2A complex in the closed state

PDB-8i6j: 
The focused refinement of CCT3-PhLP2A from TRiC-PhLP2A complex in the open state

EMDB-34155: 
Designed pH-responsive P22 VLP

PDB-8gn5: 
Designed pH-responsive P22 VLP

EMDB-35284: 
Human TRiC-PhLP2A complex in the open state

PDB-8i9u: 
Human TRiC-PhLP2A complex in the open state

EMDB-35280: 
The focused refinement of CCT4-PhLP2A from TRiC-PhLP2A complex in the open state

EMDB-35335: 
Human TRiC-PhLP2A-actin complex in the closed state

PDB-8i9q: 
The focused refinement of CCT4-PhLP2A from TRiC-PhLP2A complex in the open state

PDB-8ib8: 
Human TRiC-PhLP2A-actin complex in the closed state

EMDB-18941: 
SARS-CoV-2 S (Spike) protein (BA.1) in complex with VHH Ma16B06 (sub-volume of two adjacent RBD-VHH modules)

EMDB-28281: 
Subtomogram average of the T4SS of Coxiella Burnetii at pH 4.75
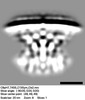
EMDB-28282: 
Subtomogram average of T4SS of Coxiella burnetii at pH 7
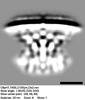
EMDB-28283: 
Subtomogram average of T4SS of Coxiella burnetii at pH7 with an inner membrane mask

EMDB-34813: 
Structure of transforming growth factor beta induced protein (TGFBIp) G623R fibril

PDB-8hia: 
Structure of transforming growth factor beta induced protein (TGFBIp) G623R fibril

EMDB-33734: 
Cryo-EM structure of SARS-CoV-2 spike in complex with K202.B bispecific antibody

PDB-7yc5: 
Cryo-EM structure of SARS-CoV-2 spike in complex with K202.B bispecific antibody

EMDB-33021: 
Cryo-EM structure of the human TRPC5 ion channel in lipid nanodiscs, class1

EMDB-34300: 
Cryo-EM structure of the human TRPC5 ion channel in lipid nanodiscs, class2

EMDB-34301: 
Cryo-EM structure of the human TRPC5 ion channel in complex with G alpha i3 subunits, class2

PDB-7x6c: 
Cryo-EM structure of the human TRPC5 ion channel in lipid nanodiscs, class1

PDB-8gvw: 
Cryo-EM structure of the human TRPC5 ion channel in lipid nanodiscs, class2

PDB-8gvx: 
Cryo-EM structure of the human TRPC5 ion channel in complex with G alpha i3 subunits, class2

EMDB-33022: 
Cryo-EM structure of the human TRPC5 ion channel in complex with G alpha i3 subunits, class1

PDB-7x6i: 
Cryo-EM structure of the human TRPC5 ion channel in complex with G alpha i3 subunits, class1

EMDB-26735: 
Hantavirus ANDV Gn(H) protein in complex with 2 Fabs ANDV-5 and ANDV-34
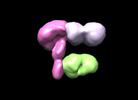
EMDB-26736: 
Hantavirus MAPV Gn(H)/Gc protein in complex with 2 Fabs SNV-24 and SNV-53

EMDB-27318: 
CryoEM structure of Hantavirus ANDV Gn(H) protein complex with 2Fabs ANDV-5 and ANDV-34

PDB-8dbz: 
CryoEM structure of Hantavirus ANDV Gn(H) protein complex with 2Fabs ANDV-5 and ANDV-34
EMDB-33984: 
Complex structure of Neuropeptide Y Y2 receptor in complex with PYY(3-36) and Gi

EMDB-33985: 
Complex structure of Neuropeptide Y Y2 receptor in complex with NPY and Gi

EMDB-35494: 
Complex structure of Neuropeptide Y Y2 receptor in complex with NPY and Gi (Consensus map)

EMDB-35495: 
Complex structure of Neuropeptide Y Y2 receptor in complex with NPY and Gi (Focused map on NPY-Y2R)

EMDB-35496: 
Complex structure of Neuropeptide Y Y2 receptor in complex with NPY and Gi (Focused map on Gi-scFv16)

EMDB-35497: 
Complex structure of Neuropeptide Y Y2 receptor in complex with PYY(3-36) and Gi (Consensus map)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
