-検索条件
-検索結果
検索 (著者・登録者: ita & s)の結果2,718件中、1から50件目までを表示しています

EMDB-60561: 
Cryo-EM structure of uropathogenic Escherichia coli CysK:CdiA:tRNA complex A

EMDB-60562: 
Cryo-EM structure of uropathogenic Escherichia coli CysK:CdiA:tRNA complex B

EMDB-60563: 
Cryo-EM structure of uropathogenic Escherichia coli 2:1 CysK:CdiA complex

EMDB-60564: 
Cryo-EM structure of uropathogenic Escherichia coli 2:2 CysK:CdiA complex

PDB-8zyc: 
Cryo-EM structure of uropathogenic Escherichia coli CysK:CdiA:tRNA complex A

PDB-8zyd: 
Cryo-EM structure of uropathogenic Escherichia coli CysK:CdiA:tRNA complex B

EMDB-41570: 
Cryo-EM structure of the rat P2X7 receptor in the apo closed state

EMDB-41581: 
Cryo-EM structure of the rat P2X7 receptor in complex with the high-affinity agonist BzATP

EMDB-42976: 
Cryo-EM structure of the rat P2X7 receptor in the apo closed state purified in the absence of sodium

PDB-8tr5: 
Cryo-EM structure of the rat P2X7 receptor in the apo closed state

PDB-8trj: 
Cryo-EM structure of the rat P2X7 receptor in complex with the high-affinity agonist BzATP

PDB-8v4s: 
Cryo-EM structure of the rat P2X7 receptor in the apo closed state purified in the absence of sodium

EMDB-38300: 
Cryo-EM structure of partial dimeric WDR11-FAM91A1 complex

EMDB-39863: 
Cryo-EM structure of dimeric WDR11-FAM91A1 complex

EMDB-39943: 
Cryo-EM structure of dimeric WDR11-FAM91A1 complex Body2

EMDB-39947: 
Cryo-EM structure of dimeric WDR11-FAM91A1 complex Body1

EMDB-39949: 
Cryo-EM structure of WDR11-dm-FAM91A1 complex

PDB-8xfb: 
Cryo-EM structure of partial dimeric WDR11-FAM91A1 complex

PDB-8z9m: 
Cryo-EM structure of dimeric WDR11-FAM91A1 complex
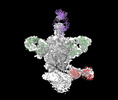
EMDB-44246: 
Cryo-EM structure of HIV-1 JRFL v6 Env in complex with vaccine-elicited, Membrane Proximal External Region (MPER) directed antibody DH1317.4.

EMDB-60959: 
Salmonella enterica serovar Typhimurium FliC(G426A)delta(204-292) forming the L-type straight filament

PDB-9iwq: 
Salmonella enterica serovar Typhimurium FliC(G426A)delta(204-292) forming the L-type straight filament

EMDB-39482: 
mouse proteasome 20S subunit in complex with compound 1

EMDB-39565: 
canine immunoproteasome 20S subunit in complex with compound 2

EMDB-39600: 
canine immunoproteasome 20S subunit in complex with compound 1

EMDB-39612: 
canine immunoproteasome 20S subunit in complex with compound 1

PDB-8ypk: 
mouse proteasome 20S subunit in complex with compound 1

PDB-8ysx: 
canine immunoproteasome 20S subunit in complex with compound 2

PDB-8yvg: 
canine immunoproteasome 20S subunit in complex with compound 1

PDB-8yvp: 
canine immunoproteasome 20S subunit in complex with compound 1
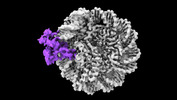
EMDB-39594: 
Cryo-EM structure of CDCA7 bound to nucleosome including hemimethylated CpG site in Widom601 positioning sequence.

PDB-8yv8: 
Cryo-EM structure of CDCA7 bound to nucleosome including hemimethylated CpG site in Widom601 positioning sequence.
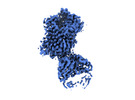
EMDB-19978: 
Outward-open structure of Drosophila dopamine transporter bound to an atypical non-competitive inhibitor
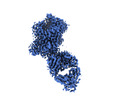
EMDB-19979: 
Inhibitor-free outward-open structure of Drosophila dopamine transporter

PDB-9euo: 
Outward-open structure of Drosophila dopamine transporter bound to an atypical non-competitive inhibitor

PDB-9eup: 
Inhibitor-free outward-open structure of Drosophila dopamine transporter

EMDB-39119: 
Cryo-EM structure of human nucleosome core particle composed of the Widom 601 DNA sequence

EMDB-39120: 
Cryo-EM structure of the human nucleosome containing the H3.1 E97K mutant

PDB-8ybj: 
Cryo-EM structure of human nucleosome core particle composed of the Widom 601 DNA sequence

PDB-8ybk: 
Cryo-EM structure of the human nucleosome containing the H3.1 E97K mutant

EMDB-39025: 
Structure of HCoV-HKU1A spike in the functionally anchored-3up conformation with 3TMPRSS2

EMDB-39026: 
Local structure of HCoV-HKU1A spike in complex with TMPRSS2 and glycan

EMDB-39036: 
Structure of HCoV-HKU1C spike in the functionally anchored-1up conformation with 1TMPRSS2

EMDB-39037: 
Structure of HCoV-HKU1C spike in the functionally anchored-2up conformation with 2TMPRSS2

EMDB-39038: 
Structure of HCoV-HKU1C spike in the functionally anchored-3up conformation with 2TMPRSS2

EMDB-39039: 
Structure of HCoV-HKU1C spike in the functionally anchored-3up conformation with 3TMPRSS2

EMDB-39040: 
Local structure of HCoV-HKU1C spike in complex with TMPRSS2 and glycan

EMDB-39041: 
Structure of HCoV-HKU1C spike in the inactive-closed conformation

EMDB-39042: 
Structure of HCoV-HKU1C spike in the inactive-1up conformation

EMDB-39043: 
Structure of HCoV-HKU1C spike in the inactive-2up conformation
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
