-検索条件
-検索結果
検索 (著者・登録者: heinz & v)の結果全49件を表示しています

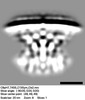
EMDB-28281: 
Subtomogram average of the T4SS of Coxiella Burnetii at pH 4.75
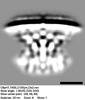
EMDB-28282: 
Subtomogram average of T4SS of Coxiella burnetii at pH 7
EMDB-28283: 
Subtomogram average of T4SS of Coxiella burnetii at pH7 with an inner membrane mask

EMDB-36754: 
SLC15A4 inhibitor complex

PDB-8jzx: 
SLC15A4 inhibitor complex

PDB-7uih: 
PSMD2 Structure

PDB-7ujd: 
PSMD2 Structure bound to MC1 and Fab8/14

EMDB-28596: 
CryoEM Structure of NLRP3 NACHT domain in complex with G2394

PDB-8etr: 
CryoEM Structure of NLRP3 NACHT domain in complex with G2394

EMDB-24742: 
PSMD2 with bound macrocycle MC1

EMDB-24743: 
PSMD2

EMDB-13930: 
3D reconstruction of the membrane domains of the sialic acid TRAP transporter HiSiaQM from Haemophilus influenzae in lipid nanodiscs bound to a high affinity megabody

PDB-7qe5: 
Structure of the membrane domains of the sialic acid TRAP transporter HiSiaQM from Haemophilus influenzae

EMDB-12676: 
Vibrio vulnificus stressosome

PDB-7o0i: 
Vibrio vulnificus stressosome

EMDB-12324: 
In situ cryo-electron tomogram of the Synechocystis wild-type VIPP1 strain grown in high light

EMDB-12325: 
In situ cryo-electron tomogram of the Synechocystis F4E VIPP1 mutant grown in high light

EMDB-12326: 
In situ cryo-electron tomogram of the Synechocystis V11E VIPP1 mutant grown in high light

EMDB-12327: 
In situ cryo-electron tomogram of a cluster of VIPP1-mCherry structures inside the Chlamydomonas chloroplast

EMDB-12328: 
In situ cryo-electron tomogram of a cluster of VIPP1-mCherry structures inside the Chlamydomonas chloroplast

EMDB-12329: 
In situ cryo-electron tomogram of a cluster of VIPP1-mCherry structures inside the Chlamydomonas chloroplast

EMDB-12330: 
In situ cryo-electron tomogram of a cluster of VIPP1-mCherry structures inside the Chlamydomonas chloroplast

EMDB-12710: 
Structural basis for VIPP1 oligomerization and maintenance of thylakoid membrane integrity

EMDB-12711: 
Structural basis for VIPP1 oligomerization and maintenance of thylakoid membrane integrity

EMDB-12712: 
Structural basis for VIPP1 oligomerization and maintenance of thylakoid membrane integrity

EMDB-12713: 
Structural basis for VIPP1 oligomerization and maintenance of thylakoid membrane integrity

EMDB-12714: 
Structural basis for VIPP1 oligomerization and maintenance of thylakoid membrane integrity

PDB-7o3w: 
Structural basis for VIPP1 oligomerization and maintenance of thylakoid membrane integrity

PDB-7o3x: 
Structural basis for VIPP1 oligomerization and maintenance of thylakoid membrane integrity

PDB-7o3y: 
Structural basis for VIPP1 oligomerization and maintenance of thylakoid membrane integrity

PDB-7o3z: 
Structural basis for VIPP1 oligomerization and maintenance of thylakoid membrane integrity

PDB-7o40: 
Structural basis for VIPP1 oligomerization and maintenance of thylakoid membrane integrity

EMDB-23156: 
SARS-CoV 2 Spike Protein bound to LY-CoV555

PDB-7l3n: 
SARS-CoV 2 Spike Protein bound to LY-CoV555

EMDB-10266: 
Homo sapiens WRB/CAML heterotetramer in complex with a TRC40 dimer

EMDB-11607: 
Saccharomyces cerevisiae Get1/Get2 heterotetramer in complex with a Get3 dimer

PDB-6so5: 
Homo sapiens WRB/CAML heterotetramer in complex with a TRC40 dimer
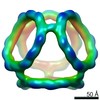
EMDB-21026: 
Truncated Tetrahedral RNA Nanostructures

EMDB-3450: 
Structure of the K+ transporter KtrAB from Vibrio alginolyticus in the ADP-bound state

EMDB-3523: 
cryoEM Structure of Polycystin-2 in complex with cations and lipids

EMDB-3524: 
cryoEM Structure of Polycystin-2 in complex with calcium and lipids

PDB-5mke: 
cryoEM Structure of Polycystin-2 in complex with cations and lipids

PDB-5mkf: 
cryoEM Structure of Polycystin-2 in complex with calcium and lipids
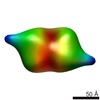
EMDB-2640: 
Negative-stain electron microscopy of the human GINS complex

EMDB-5694: 
Type III secretion system (Injectisome) of Yersinia enterocolitica in situ

PDB-3ixv: 
Scorpion Hemocyanin resting state pseudo atomic model built based on cryo-EM density map

PDB-3ixw: 
Scorpion Hemocyanin activated state pseudo atomic model built based on cryo-EM density map

EMDB-5100: 
24-meric Scorpion Hemocyanin Resting State cryo-EM Density Map at 6.8 Angstrom Resolution

EMDB-5101: 
24-meric Scorpion Hemocyanin Activated State cryo-EM Density Map at 8 Angstrom Resolution
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
