-検索条件
-検索結果
検索 (著者・登録者: harris & cl)の結果83件中、1から50件目までを表示しています
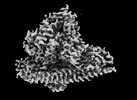
EMDB-41617: 
CryoEM structure of PI3Kalpha

PDB-8tu6: 
CryoEM structure of PI3Kalpha

EMDB-26520: 
Post-fusion ectodomain of HSV-1 gB in complex with BMPC-23 Fab

EMDB-26521: 
Post-fusion ectodomain of HSV-1 gB in complex with HSV010-13 Fab

PDB-7uhz: 
Post-fusion ectodomain of HSV-1 gB in complex with BMPC-23 Fab

PDB-7ui0: 
Post-fusion ectodomain of HSV-1 gB in complex with HSV010-13 Fab

EMDB-14531: 
CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11 nanobody complex

EMDB-14539: 
CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11-H6 nanobody complex

EMDB-14543: 
CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11-B5 nanobody complex

EMDB-14544: 
CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11-H4 Q98R H100E nanobody complex in 1Up2Down conformation

EMDB-14575: 
CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11-A10 nanobody complex

EMDB-14576: 
CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11-H4 Q98R H100E nanobody complex in 2Up1Down conformation

PDB-7z6v: 
CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11 nanobody complex

PDB-7z7x: 
CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11-H6 nanobody complex

PDB-7z85: 
CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11-B5 nanobody complex

PDB-7z86: 
CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11-H4 Q98R H100E nanobody complex in 1Up2Down conformation

PDB-7z9q: 
CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11-A10 nanobody complex

PDB-7z9r: 
CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11-H4 Q98R H100E nanobody complex in 2Up1Down conformation

EMDB-14332: 
1.58 A STRUCTURE OF HUMAN APOFERRITIN OBTAINED FROM TITAN KRIOS 2 AT eBIC, DLS UNDER COMMISSIONING SESSION CM26464-2

PDB-7r5o: 
1.58 A STRUCTURE OF HUMAN APOFERRITIN OBTAINED FROM TITAN KRIOS 2 AT eBIC, DLS UNDER COMMISSIONING SESSION CM26464-2

EMDB-14153: 
SARS-CoV-2 Spike, C3 symmetry

PDB-7qus: 
SARS-CoV-2 Spike, C3 symmetry

EMDB-14152: 
SARS-CoV-2 Spike with ethylbenzamide-tri-iodo Siallyllactose, C3 symmetry

EMDB-14154: 
SARS-CoV-2 Spike with ethylbenzamide-tri-iodo Siallyllactose, C1 symmetry

EMDB-14155: 
SARS-CoV-2 Spike, C1 symmetry

PDB-7qur: 
SARS-CoV-2 Spike with ethylbenzamide-tri-iodo Siallyllactose, C3 symmetry

EMDB-24715: 
Focused Refined Map BORF2-APOBEC3Bctd Complex

EMDB-24716: 
BORF2-APOBEC3Bctd Complex

EMDB-24709: 
Composite Map BORF2-APOBEC3Bctd Complex

PDB-7rw6: 
BORF2-APOBEC3Bctd Complex

EMDB-25794: 
Cryo-EM structure of SARS-CoV-2 spike in complex with antibodies B1-182.1 and A19-61.1

EMDB-25797: 
Locally refined region of SARS-CoV-2 spike in complex with antibodies B1-182.1 and A19-61.1

EMDB-25806: 
Locally refined region of SARS-CoV-2 spike in complex with antibody A19-46.1

EMDB-25807: 
Cryo-EM structure of SARS-CoV-2 Omicron spike in complex with antibody A19-46.1
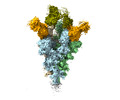
EMDB-25808: 
Cryo-EM structure of SARS-CoV-2 Omicron spike in complex with antibodies A19-46.1 and B1-182.1

EMDB-26256: 
Local refinement of cryo-EM structure of the interface of the Omicron RBD in complex with antibodies B-182.1 and A19-46.1

PDB-7tb8: 
Cryo-EM structure of SARS-CoV-2 spike in complex with antibodies B1-182.1 and A19-61.1

PDB-7tbf: 
Locally refined region of SARS-CoV-2 spike in complex with antibodies B1-182.1 and A19-61.1

PDB-7tc9: 
Locally refined region of SARS-CoV-2 spike in complex with antibody A19-46.1

PDB-7tca: 
Cryo-EM structure of SARS-CoV-2 Omicron spike in complex with antibody A19-46.1

PDB-7tcc: 
Cryo-EM structure of SARS-CoV-2 Omicron spike in complex with antibodies A19-46.1 and B1-182.1

PDB-7u0d: 
Local refinement of cryo-EM structure of the interface of the Omicron RBD in complex with antibodies B-182.1 and A19-46.1

EMDB-22788: 
Cryo-EM structure of the HCMV pentamer bound by Fabs 2-18 and 8I21

EMDB-23629: 
Cryo-EM structure of the HCMV pentamer bound by human neuropilin 2

EMDB-23640: 
Cryo-EM structure of the HCMV pentamer bound by antibodies 1-103, 1-32 and 2-25

PDB-7kbb: 
Cryo-EM structure of the HCMV pentamer bound by Fabs 2-18 and 8I21

PDB-7m22: 
Cryo-EM structure of the HCMV pentamer bound by human neuropilin 2

PDB-7m30: 
Cryo-EM structure of the HCMV pentamer bound by antibodies 1-103, 1-32 and 2-25

EMDB-12777: 
Nanobody C5 bound to Spike

PDB-7oan: 
Nanobody C5 bound to Spike
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
