-検索条件
-検索結果
検索 (著者・登録者: de & la & cruz & f)の結果60件中、1から50件目までを表示しています

EMDB-41583: 
Actin 1 from T. gondii in filaments bound to MgADP

EMDB-41584: 
Actin 1 from T. gondii in filaments bound to MgADP and jasplakinolide

PDB-8trm: 
Actin 1 from T. gondii in filaments bound to MgADP

PDB-8trn: 
Actin 1 from T. gondii in filaments bound to MgADP and jasplakinolide
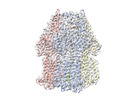
EMDB-17350: 
Single particle cryo-EM co-structure of Klebsiella pneumoniae AcrB with the BDM91288 efflux pump inhibitor at 2.97 Angstrom resolution

PDB-8p1i: 
Single particle cryo-EM co-structure of Klebsiella pneumoniae AcrB with the BDM91288 efflux pump inhibitor at 2.97 Angstrom resolution
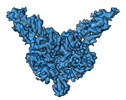
EMDB-15213: 
Cryo-EM density map of Tn4430 TnpA hyperactive mutant (TnpA3X) in complex with IR48 substrate.

EMDB-15218: 
Medium resolution cryo-EM density map of Tn4430 TnpA transposase from Tn3 family in apo state

EMDB-25132: 
Androgen receptor bound to DNA - Entrenched state

EMDB-25133: 
Androgen receptor bound to DNA - Splayed state

EMDB-25134: 
Androgen receptor bound to DNA - Divorced state

EMDB-25105: 
Structure of SARS-CoV S protein in complex with Receptor Binding Domain antibody DH1047

PDB-7sg4: 
Structure of SARS-CoV S protein in complex with Receptor Binding Domain antibody DH1047

EMDB-22170: 
BG505 SOSIPv5.2 in complex with PGT122 and two RM20E1 Fabs

EMDB-22171: 
BG505 SOSIPv5.2 in complex with PGT122 and three RM20E1 Fabs

EMDB-22172: 
C3 symmetric BG505 SOSIPv5.2 in complex with PGT122 and three RM20E1 Fabs

EMDB-22173: 
BG505 SOSIPv5.2 in complex with PGT122 and one RM20E1 Fab

EMDB-22178: 
BG505 SOSIPv5.2 in complex with PGT122 and no RM20E1 Fabs

EMDB-22179: 
C3 symmetric BG505 SOSIPv5.2 in complex with PGT122 and no RM20E1 Fabs

EMDB-22137: 
Complex of SARS-CoV-2 receptor binding domain with the Fab fragments of two neutralizing antibodies

PDB-6xdg: 
Complex of SARS-CoV-2 receptor binding domain with the Fab fragments of two neutralizing antibodies

EMDB-20711: 
Cryo-EM structure of cofilactin from partially cofilin-decorated actin filaments.

PDB-6vao: 
Human cofilin-1 decorated actin filament

EMDB-20719: 
Bare actin from partially cofilin-decorated actin filaments

EMDB-20721: 
Isolated cofilin bound to an actin filament

EMDB-20724: 
Isolated S3D-cofilin bound to an actin filament

EMDB-20726: 
Barbed end side of a cofilactin cluster

PDB-6uby: 
Isolated cofilin bound to an actin filament

PDB-6uc0: 
Isolated S3D-cofilin bound to an actin filament

PDB-6uc4: 
Barbed end side of a cofilactin cluster

PDB-6vau: 
Bare actin filament from a partially cofilin-decorated sample

EMDB-7321: 
Integrative Structure and Functional Anatomy of a Nuclear Pore Complex
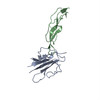
PDB-5ty4: 
MicroED structure of a complex between monomeric TGF-b and its receptor, TbRII, at 2.9 A resolution

PDB-5k7n: 
MicroED structure of tau VQIVYK peptide at 1.1 A resolution

PDB-5k7o: 
MicroED structure of lysozyme at 1.8 A resolution

PDB-5k7p: 
MicroED structure of xylanase at 2.3 A resolution

PDB-5k7q: 
MicroED structure of thaumatin at 2.5 A resolution

PDB-5k7r: 
MicroED structure of trypsin at 1.7 A resolution

PDB-5k7s: 
MicroED structure of proteinase K at 1.6 A resolution

PDB-5k7t: 
MicroED structure of thermolysin at 2.5 A resolution

PDB-5l08: 
Cryo-EM structure of Casp-8 tDED filament

EMDB-8300: 
Cryo-EM structure of Casp-8 tDED filament (CASP target)

EMDB-8216: 
MicroED structure of tau VQIVYK peptide at 1.1 A resolution

EMDB-8217: 
MicroED structure of lysozyme at 1.8 A resolution

EMDB-8218: 
MicroED structure of xylanase at 2.3 A resolution

EMDB-8219: 
MicroED structure of thaumatin at 2.5 A resolution

EMDB-8220: 
MicroED structure of trypsin at 1.7 A resolution

EMDB-8221: 
MicroED structure of proteinase K at 1.6 A resolution

EMDB-8222: 
MicroED structure of thermolysin at 2.5 A resolution

EMDB-8472: 
MicroED structure of a complex between monomeric TGF-b and its receptor, TbRII, at 2.9 A resolution
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
