-検索条件
-検索結果
検索 (著者・登録者: baric & r)の結果109件中、1から50件目までを表示しています

EMDB-44644: 
SARS CoV2 spike in complex with NTD-directed antibody Fab DH1052

EMDB-18245: 
Plunge-frozen (control) map of beta-galactosidase

EMDB-18244: 
ESIBD structure of beta-galactosidase

PDB-8q7y: 
ESIBD structure of beta-galactosidase

EMDB-40272: 
BtCoV-422 in complex with neutralizing antibody JC57-11

EMDB-40306: 
BtCoV-422 spike in complex with JC57-11 Fab, global map

EMDB-40310: 
BtCoV-422 spike in complex with JC57-11 Fab, local map

PDB-8sak: 
BtCoV-422 in complex with neutralizing antibody JC57-11

EMDB-27703: 
Structure of RBD directed antibody DH1047 in complex with SARS-CoV-2 spike: Local refinement of RBD-Fab interace

PDB-8dtk: 
Structure of RBD directed antibody DH1047 in complex with SARS-CoV-2 spike: Local refinement of RBD-Fab interace
EMDB-25618: 
SARS-CoV-2 VFLIP spike boung to 2 Ab12 Fab fragments
EMDB-25663: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with Three (3) RBDs Up, Bound to Antibody 2-7 scFv, composite map

EMDB-25689: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with Three (3) RBDs Up, Bound to Antibody 2-7 scFv, global map with poorly-resolved RBDs and scFvs

EMDB-25690: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with Three (3) RBDs Up, Bound to Antibody 2-7 scFv, local refinement map

EMDB-25711: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with One(1) RBD Up

PDB-7t3m: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with Three (3) RBDs Up, Bound to Antibody 2-7 scFv, composite map

PDB-7t67: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with One(1) RBD Up

EMDB-27438: 
Cryo-EM structure of SARS-CoV-2 Beta (B.1.351) spike protein in complex with VH domain F6

EMDB-27439: 
Cryo-EM structure of SARS-CoV-2 Beta (B.1.351) spike protein in complex with VH domain F6 (focused refinement of RBD and VH F6)

PDB-8di5: 
Cryo-EM structure of SARS-CoV-2 Beta (B.1.351) spike protein in complex with VH domain F6 (focused refinement of RBD and VH F6)

EMDB-26372: 
SARS-2 CoV 6P Mut7 in complex with Fab CC84.5

EMDB-26365: 
SARS-2 CoV 6P Mut7 in complex with Fab CC25.1
EMDB-26366: 
SARS-2 CoV 6P Mut7 in complex with Fab CC25.54

EMDB-26367: 
SARS-2 CoV 6P Mut7 + Fab CC25.52

EMDB-26368: 
SARS-2 CoV 6P Mut7 in complex with Fab CC25.56
EMDB-26369: 
SARS-2 CoV 6P Mut7 in complex with Fab CC25.36

EMDB-26370: 
SARS-2 CoV 6P Mut7 in complex with Fab CC25.3
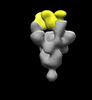
EMDB-26371: 
SARS-2 CoV 6P Mut7 in complex with Fab CC25.11

EMDB-26373: 
SARS-2 CoV 6P Mut7 in complex with Fab CC84.2

EMDB-26374: 
SARS-2 CoV 6P Mut7 in complex with Fab CC84.10
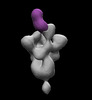
EMDB-26375: 
SARS-2 CoV 6P Mut7 in complex with Fab CC84.24

EMDB-25792: 
Cryo-EM structure of the spike of SARS-CoV-2 Omicron variant of concern

PDB-7tb4: 
Cryo-EM structure of the spike of SARS-CoV-2 Omicron variant of concern

EMDB-25105: 
Structure of SARS-CoV S protein in complex with Receptor Binding Domain antibody DH1047

PDB-7sg4: 
Structure of SARS-CoV S protein in complex with Receptor Binding Domain antibody DH1047

EMDB-25072: 
Fab22 bound to MERS-CoV Spike

EMDB-25073: 
Structure of Fab22 complexed SARS-CoV-2 spike

EMDB-23914: 
Cryo-EM structure of SARS-CoV-2 spike in complex with neutralizing antibody B1-182.1 that targets the receptor-binding domain

EMDB-23915: 
Cryo-EM structure of SARS-CoV-2 spike in complex with neutralizing antibody B1-182.1 that targets the receptor-binding domain

PDB-7mlz: 
Cryo-EM structure of SARS-CoV-2 spike in complex with neutralizing antibody B1-182.1 that targets the receptor-binding domain

PDB-7mm0: 
Cryo-EM structure of SARS-CoV-2 spike in complex with neutralizing antibody B1-182.1 that targets the receptor-binding domain

EMDB-23498: 
Cryo-EM structure of SARS-CoV-2 spike in complex with neutralizing antibody A23-58.1 that targets the receptor-binding domain

EMDB-23499: 
Cryo-EM structure of SARS-CoV-2 spike in complex with neutralizing antibody A23-58.1 that targets the receptor-binding domain

PDB-7lrs: 
Cryo-EM structure of SARS-CoV-2 spike in complex with neutralizing antibody A23-58.1 that targets the receptor-binding domain

PDB-7lrt: 
Cryo-EM structure of SARS-CoV-2 spike in complex with neutralizing antibody A23-58.1 that targets the receptor-binding domain

EMDB-23717: 
SARS-CoV-2 S-NTD + Fab CM25

PDB-7m8j: 
SARS-CoV-2 S-NTD + Fab CM25

EMDB-23400: 
SARS-CoV-2 Spike Protein Trimer bound to DH1043 fab

PDB-7ljr: 
SARS-CoV-2 Spike Protein Trimer bound to DH1043 fab

EMDB-23246: 
CryoEM map of SARS-CoV-2 S protein in complex with Receptor Binding Domain antibody DH1041
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
