-検索条件
-検索結果
検索 (著者・登録者: baran & k)の結果全48件を表示しています

EMDB-41363: 
Cryo-EM structure of DDB1deltaB-DDA1-DCAF5

PDB-8tl6: 
Cryo-EM structure of DDB1deltaB-DDA1-DCAF5

EMDB-17391: 
Subtomogram average of ribosomes in germinated polar tubes of Vairimorpha necatrix

EMDB-17467: 
Subtomogram representing a segment of the outer wall of empty germinated polar tubes from Vairimorpha necatrix.

EMDB-17468: 
Subtomogram representing a segment of the outer wall of cargo-filled, germinated polar tubes from Vairimorpha necatrix.

EMDB-15311: 
Dedicated chaperone at the ribosome safeguards the proteostasis network during eEF1A biogenesis

EMDB-27256: 
Human DNA polymerase alpha/primase elongation complex I bound to primer/template
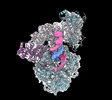
EMDB-27258: 
Human DNA polymerase-alpha/primase elongation complex II bound to primer/template

PDB-8d96: 
Human DNA polymerase alpha/primase elongation complex I bound to primer/template

PDB-8d9d: 
Human DNA polymerase-alpha/primase elongation complex II bound to primer/template
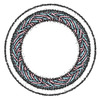
EMDB-14630: 
Membrane-bound CHMP2A-CHMP3 filament (430 Angstrom diameter)
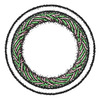
EMDB-14631: 
Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom diameter)

PDB-7zcg: 
CHMP2A-CHMP3 heterodimer (430 Angstrom diameter)

PDB-7zch: 
CHMP2A-CHMP3 heterodimer (410 Angstrom diameter)

EMDB-15365: 
Vairimorpha necatrix 20S proteasome from spores

EMDB-15366: 
Vairimorpha necatrix 26S proteasome from sporoplasms

EMDB-15367: 
Vairimorpha necatrix 20S proteasome from sporoplasms

PDB-8adn: 
Vairimorpha necatrix 20S proteasome from spores

EMDB-12816: 
Enterococcus faecalis EfrCD in complex with a nanobody

PDB-7ocy: 
Enterococcus faecalis EfrCD in complex with a nanobody

EMDB-13776: 
Structure of formaldehyde cross-linked SARS-CoV-2 S glycoprotein

PDB-7q1z: 
Structure of formaldehyde cross-linked SARS-CoV-2 S glycoprotein

EMDB-13185: 
Helical structure of the toxin MakA from Vibrio cholera

PDB-7p3r: 
Helical structure of the toxin MakA from Vibrio cholera

EMDB-11437: 
Structure of the Paranosema locustae ribosome in complex with Lso2

PDB-6zu5: 
Structure of the Paranosema locustae ribosome in complex with Lso2

EMDB-4931: 
Cryo-EM structure of the microsporidian ribosome: Multibody-refined map body 1 (LSU)

EMDB-4932: 
Cryo-EM structure of the microsporidian ribosome: Multibody-refined map body 2 (SSU-body)

EMDB-4933: 
Cryo-EM structure of the microsporidian ribosome: Multibody-refined map body 3 (SSU-head)

EMDB-4934: 
Cryo-EM structure of the microsporidian ribosome: State 1, Multibody-refined map body 3 (SSU-head)

EMDB-4935: 
Evolutionary compaction and adaptation visualized by the structure of the dormant microsporidian ribosome

PDB-6rm3: 
Evolutionary compaction and adaptation visualized by the structure of the dormant microsporidian ribosome

EMDB-0441: 
Conformational switches control early maturation of the eukaryotic small ribosomal subunit

PDB-6nd4: 
Conformational switches control early maturation of the eukaryotic small ribosomal subunit

EMDB-7324: 
Yeast nucleolar pre-60S ribosomal subunit (state 2)

EMDB-7445: 
Yeast nucleolar pre-60S ribosomal subunit (state 3)

PDB-6c0f: 
Yeast nucleolar pre-60S ribosomal subunit (state 2)

PDB-6cb1: 
Yeast nucleolar pre-60S ribosomal subunit (state 3)

EMDB-8859: 
The complete structure of the small subunit processome

PDB-5wlc: 
The complete structure of the small subunit processome

EMDB-8473: 
Architecture of the yeast small subunit processome

PDB-5tzs: 
Architecture of the yeast small subunit processome

EMDB-8223: 
Random conical tilt reconstruction of Saccharomyces cerevisiae UtpB

EMDB-6370: 
3D-Structure of negatively stained Schistosome myosin filament obtained by low-dose electron microscopy

PDB-3jax: 
Heavy meromyosin from Schistosoma mansoni muscle thick filament by negative stain EM

EMDB-1769: 
Perforin Pore

EMDB-1772: 
Perforin monomer, conformation 1

EMDB-1773: 
Perforin monomer, conformation 2
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
