-検索条件
-検索結果
検索 (著者・登録者: bae & c)の結果326件中、1から50件目までを表示しています

EMDB-42918: 
Cryo-EM Structure of Wildtype Smooth Muscle Gamma Actin (ACTG2)

EMDB-42939: 
Smooth Muscle Gamma Actin (ACTG2) Filament Mutant R40C

EMDB-42938: 
Cryo-EM Structure of Smooth Muscle Gamma Actin (ACTG2) Mutant R257C

EMDB-18609: 
Cryo-EM structure of the light-driven sodium pump ErNaR in the pentameric form at pH 8.0

EMDB-18610: 
Cryo-EM structure of the light-driven sodium pump ErNaR in the pentameric form at pH 4.3

PDB-8qqz: 
Cryo-EM structure of the light-driven sodium pump ErNaR in the pentameric form at pH 8.0

PDB-8qr0: 
Cryo-EM structure of the light-driven sodium pump ErNaR in the pentameric form at pH 4.3

EMDB-43091: 
hGBP1 conformer on the bacterial outer membrane

EMDB-43153: 
hGBP1 conformer on the bacterial outer membrane

EMDB-29400: 
Acinetobacter baylyi LptB2FG bound to lipopolysaccharide and a macrocyclic peptide

EMDB-29401: 
Acinetobacter baylyi LptB2FG bound to lipopolysaccharide.

EMDB-29402: 
Acinetobacter baylyi LptB2FG bound to lipopolysaccharide and RG6006

EMDB-29403: 
Acinetobacter baylyi LptB2FG bound to lipopolysaccharide and a macrocyclic peptide

EMDB-29404: 
Acinetobacter baylyi LptB2FGC

EMDB-42206: 
Acinetobacter baylyi LptB2FG bound to Acinetobacter baylyi lipopolysaccharide

EMDB-42207: 
Acinetobacter baylyi LptB2FG bound to Acinetobacter baylyi lipopolysaccharide and a macrocyclic peptide

PDB-8frl: 
Acinetobacter baylyi LptB2FG bound to lipopolysaccharide and a macrocyclic peptide

PDB-8frm: 
Acinetobacter baylyi LptB2FG bound to lipopolysaccharide.

PDB-8frn: 
Acinetobacter baylyi LptB2FG bound to lipopolysaccharide and Zosurabalpin

PDB-8fro: 
Acinetobacter baylyi LptB2FG bound to lipopolysaccharide and a macrocyclic peptide

PDB-8frp: 
Acinetobacter baylyi LptB2FGC

PDB-8ufg: 
Acinetobacter baylyi LptB2FG bound to Acinetobacter baylyi lipopolysaccharide

PDB-8ufh: 
Acinetobacter baylyi LptB2FG bound to Acinetobacter baylyi lipopolysaccharide and a macrocyclic peptide

EMDB-41193: 
Shaker in low K+ (4mM K+)

PDB-8teo: 
Shaker in low K+ (4mM K+)

EMDB-35010: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein

EMDB-35770: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein (Consensus map)

EMDB-35772: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein (Receptor-focused map)

EMDB-35773: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein (G protein-focused map)
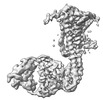
EMDB-35971: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c-bRIL
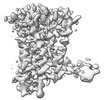
EMDB-37336: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c only

PDB-8hti: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein

PDB-8j46: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c-bRIL

PDB-8w77: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c only
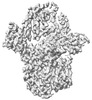
EMDB-16891: 
Structure of the UBE1L activating enzyme bound to ISG15 and UBE2L6
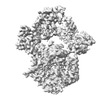
EMDB-18589: 
Map of UBE1L activating enzyme bound to ISG15 (FL) and UBE2L6

PDB-8oif: 
Structure of the UBE1L activating enzyme bound to ISG15 and UBE2L6

EMDB-29452: 
Structure Of Respiratory Syncytial Virus Polymerase with Novel Non-Nucleoside Inhibitor

PDB-8fu3: 
Structure Of Respiratory Syncytial Virus Polymerase with Novel Non-Nucleoside Inhibitor

EMDB-17804: 
Bat-Hp-CoV Nsp1 and eIF1 bound to the human 40S small ribosomal subunit

EMDB-17805: 
MERS-CoV Nsp1 bound to the human 43S pre-initiation complex

PDB-8ppk: 
Bat-Hp-CoV Nsp1 and eIF1 bound to the human 40S small ribosomal subunit

PDB-8ppl: 
MERS-CoV Nsp1 bound to the human 43S pre-initiation complex

EMDB-40349: 
CryoEM structure of rat Kv2.1(1-598) wild type in nanodiscs

EMDB-40350: 
CryoEM structure of rat Kv2.1(1-598) L403A mutant in nanodiscs

PDB-8sd3: 
CryoEM structure of rat Kv2.1(1-598) wild type in nanodiscs

PDB-8sda: 
CryoEM structure of rat Kv2.1(1-598) L403A mutant in nanodiscs

EMDB-28063: 
CryoEM of the soluble OPA1 dimer from the GDP-AlFx bound helical assembly on a lipid membrane

EMDB-28074: 
CryoEM of the soluble OPA1 dimer from the apo helical assembly on a lipid membrane

EMDB-40192: 
CryoEM map of the locally refined soluble OPA1 Z-clip from the GDP-AlFx bound helical assembly on a lipid membrane
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
