+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4e4d | ||||||
|---|---|---|---|---|---|---|---|
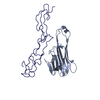
| タイトル | Crystal structure of mouse RANKL-OPG complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | Cytokine/Signaling Protein / TNF-Related Activation-Induced Cytokine-Receptor / Cysteine-Rich Domain /  Jelly-Roll Fold / Cytokine-Signaling Protein complex Jelly-Roll Fold / Cytokine-Signaling Protein complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of odontogenesis of dentin-containing tooth / positive regulation of corticotropin-releasing hormone secretion / positive regulation of fever generation by positive regulation of prostaglandin secretion / : /  tooth eruption / positive regulation of osteoclast development / TNF receptor superfamily (TNFSF) members mediating non-canonical NF-kB pathway / osteoclast proliferation / : / TNFs bind their physiological receptors ...negative regulation of odontogenesis of dentin-containing tooth / positive regulation of corticotropin-releasing hormone secretion / positive regulation of fever generation by positive regulation of prostaglandin secretion / : / tooth eruption / positive regulation of osteoclast development / TNF receptor superfamily (TNFSF) members mediating non-canonical NF-kB pathway / osteoclast proliferation / : / TNFs bind their physiological receptors ...negative regulation of odontogenesis of dentin-containing tooth / positive regulation of corticotropin-releasing hormone secretion / positive regulation of fever generation by positive regulation of prostaglandin secretion / : /  tooth eruption / positive regulation of osteoclast development / TNF receptor superfamily (TNFSF) members mediating non-canonical NF-kB pathway / osteoclast proliferation / : / TNFs bind their physiological receptors / tooth eruption / positive regulation of osteoclast development / TNF receptor superfamily (TNFSF) members mediating non-canonical NF-kB pathway / osteoclast proliferation / : / TNFs bind their physiological receptors /  tumor necrosis factor receptor superfamily binding / TNFR2 non-canonical NF-kB pathway / response to xenobiotic stimulus => GO:0009410 / positive regulation of homotypic cell-cell adhesion / regulation of osteoclast differentiation / tumor necrosis factor receptor superfamily binding / TNFR2 non-canonical NF-kB pathway / response to xenobiotic stimulus => GO:0009410 / positive regulation of homotypic cell-cell adhesion / regulation of osteoclast differentiation /  傍分泌 / response to arsenic-containing substance / positive regulation of osteoclast differentiation / 傍分泌 / response to arsenic-containing substance / positive regulation of osteoclast differentiation /  tumor necrosis factor receptor binding / negative regulation of bone resorption / osteoclast development / mammary gland epithelial cell proliferation / positive regulation of intracellular signal transduction / monocyte chemotaxis / response to magnesium ion / mammary gland alveolus development / positive regulation of bone resorption / calcium ion homeostasis / lymph node development / positive regulation of phosphorylation / JNK cascade / tumor necrosis factor receptor binding / negative regulation of bone resorption / osteoclast development / mammary gland epithelial cell proliferation / positive regulation of intracellular signal transduction / monocyte chemotaxis / response to magnesium ion / mammary gland alveolus development / positive regulation of bone resorption / calcium ion homeostasis / lymph node development / positive regulation of phosphorylation / JNK cascade /  bone resorption / ERK1 and ERK2 cascade / tumor necrosis factor-mediated signaling pathway / bone resorption / ERK1 and ERK2 cascade / tumor necrosis factor-mediated signaling pathway /  細胞外マトリックス / cellular response to leukemia inhibitory factor / extracellular matrix organization / 細胞外マトリックス / cellular response to leukemia inhibitory factor / extracellular matrix organization /  骨化 / osteoclast differentiation / response to nutrient / phosphatidylinositol 3-kinase/protein kinase B signal transduction / 骨化 / osteoclast differentiation / response to nutrient / phosphatidylinositol 3-kinase/protein kinase B signal transduction /  cytokine activity / calcium-mediated signaling / animal organ morphogenesis / positive regulation of JNK cascade / cytokine activity / calcium-mediated signaling / animal organ morphogenesis / positive regulation of JNK cascade /  骨 / cytokine-mediated signaling pathway / response to estrogen / positive regulation of DNA-binding transcription factor activity / positive regulation of T cell activation / positive regulation of canonical NF-kappaB signal transduction / 骨 / cytokine-mediated signaling pathway / response to estrogen / positive regulation of DNA-binding transcription factor activity / positive regulation of T cell activation / positive regulation of canonical NF-kappaB signal transduction /  receptor ligand activity / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / receptor ligand activity / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction /  免疫応答 / apoptotic process / positive regulation of gene expression / negative regulation of transcription by RNA polymerase II / 免疫応答 / apoptotic process / positive regulation of gene expression / negative regulation of transcription by RNA polymerase II /  シグナル伝達 / positive regulation of transcription by RNA polymerase II / シグナル伝達 / positive regulation of transcription by RNA polymerase II /  extracellular space / identical protein binding / extracellular space / identical protein binding /  細胞膜 / 細胞膜 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.7 Å 分子置換 / 解像度: 2.7 Å | ||||||
 データ登録者 データ登録者 | Nelson, C.A. / Fremont, D.H. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 2012 ジャーナル: Structure / 年: 2012タイトル: RANKL Employs Distinct Binding Modes to Engage RANK and the Osteoprotegerin Decoy Receptor. 著者: Nelson, C.A. / Warren, J.T. / Wang, M.W. / Teitelbaum, S.L. / Fremont, D.H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4e4d.cif.gz 4e4d.cif.gz | 136.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4e4d.ent.gz pdb4e4d.ent.gz | 106 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4e4d.json.gz 4e4d.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/e4/4e4d https://data.pdbj.org/pub/pdb/validation_reports/e4/4e4d ftp://data.pdbj.org/pub/pdb/validation_reports/e4/4e4d ftp://data.pdbj.org/pub/pdb/validation_reports/e4/4e4d | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 17376.463 Da / 分子数: 1 / 断片: UNP residues 162-316 / 由来タイプ: 組換発現 / 由来: (組換発現)   Mus musculus (ハツカネズミ) / 遺伝子: CD254, ODF, Opgl, Rankl, Tnfsf11, Trance / プラスミド: pGEX-6P-1 / 発現宿主: Mus musculus (ハツカネズミ) / 遺伝子: CD254, ODF, Opgl, Rankl, Tnfsf11, Trance / プラスミド: pGEX-6P-1 / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21CodonPlus(DE3)-RIL / 参照: UniProt: O35235 Escherichia coli (大腸菌) / 株 (発現宿主): BL21CodonPlus(DE3)-RIL / 参照: UniProt: O35235 | ||
|---|---|---|---|
| #2: タンパク質 | 分子量: 20372.166 Da / 分子数: 1 / 断片: UNP residues 22-197 / 由来タイプ: 組換発現 / 由来: (組換発現)   Mus musculus (ハツカネズミ) / 株: C57BL/6 B / 遺伝子: Ocif, Opg, Osteoprotegerin, Tnfrsf11b Mus musculus (ハツカネズミ) / 株: C57BL/6 B / 遺伝子: Ocif, Opg, Osteoprotegerin, Tnfrsf11b発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)株 (発現宿主): HIGH FIVE / 参照: UniProt: O08712 | ||
| #3: 化合物 |  塩化物 塩化物#4: 水 | ChemComp-HOH / |  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.71 Å3/Da / 溶媒含有率: 66.83 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 4.1 詳細: 100 mM sodium phosphate/citrate buffer, 14% polyethylene glycol 8000 and 250 mM sodium chloride, pH 4.10, VAPOR DIFFUSION, HANGING DROP, temperature 293K |
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ALS ALS  / ビームライン: 4.2.2 / 波長: 0.9951 Å / ビームライン: 4.2.2 / 波長: 0.9951 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: NOIR-1 / 検出器: CCD / 日付: 2007年1月1日 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長 : 0.9951 Å / 相対比: 1 : 0.9951 Å / 相対比: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2.7→50 Å / Num. obs: 14915 / % possible obs: 99.3 % / 冗長度: 2.9 % / Biso Wilson estimate: 48.42 Å2 / Rmerge(I) obs: 0.084 / Net I/σ(I): 12.8 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル |
|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 1JTZ 解像度: 2.7→40.7 Å / Occupancy max: 1 / Occupancy min: 1 / SU ML: 0.29 / 位相誤差: 23.59 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 71.12 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.7→40.7 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj








